PCR
Sigla di polymerase chain reaction («reazione a catena della polimerasi»), metodologia utilizzata per ottenere quantità che ammontano a μg di copie di segmenti specifici di DNA o di RNA, partendo da quantità minime (anche una sola molecola) presenti in una preparazione di acidi nucleici. Ideata negli anni 1980 da K. Mullis, ha avuto negli anni successivi un tale sviluppo da rivoluzionare molti campi della ricerca di base e applicata. Le sue numerose applicazioni riguardano la genetica molecolare, la diagnostica, la medicina forense, le analisi alimentari e microbiologiche e gli studi di filogenesi molecolare.
Descrizione della tecnica
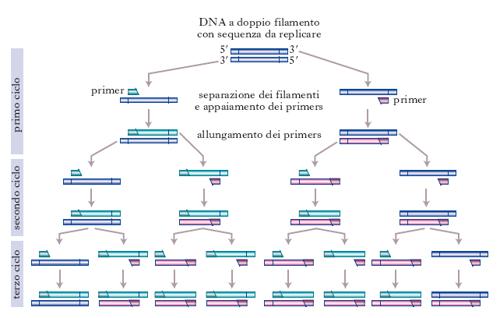
Il materiale di partenza della PCR è il DNA contenente la sequenza che deve essere amplificata. I due filamenti della doppia elica dell’acido nucleico che servono da stampo sono separati mediante riscaldamento a una temperatura di 94 °C, seguito da un successivo raffreddamento per permettere l’ibridazione dei primers o inneschi, oligonucleotidi costituiti da circa 16 paia di basi che servono da inneschi per la sintesi del DNA in vitro. Gli inneschi sono complementari alle sequenze fiancheggianti la regione che deve essere amplificata, e si appaiano sui filamenti opposti del DNA stampo (v. fig.). La temperatura raggiunta in questa fase, detta temperatura di annealing o di appaiamento, è un parametro variabile, capace di determinare la specificità della PCR in relazione alla regione di DNA che deve essere amplificata. La polimerizzazione delle molecole di DNA a partire dai primers è condotta da una DNA-polimerasi che deve essere termostabile, in modo che non si inattivi in seguito a riscaldamento: a tale scopo sono utilizzati enzimi isolati da batteri termofili, come, per es., la Taq polimerasi, isolata da Thermophilus aquaticus. La fase di sintesi del nuovo filamento si verifica a circa 72 °C, che è la temperatura ottimale per l’attività della Taq polimerasi. Nella miscela di reazione deve essere presente anche una certa quantità dei quattro nucleotidi precursori. Successivamente all’allungamento dell’innesco oligonucleotidico e quindi alla polimerizzazione di filamenti di DNA complementari alla molecola di partenza, si sottopone la reazione a un nuovo ciclo di riscaldamento-raffreddamento-allungamento per ottenere la specifica molecola di DNA che si vuole amplificare, la cui lunghezza corrisponde alla distanza fra i due inneschi. Questo frammento di DNA di nuova sintesi serve a sua volta da stampo per una nuova reazione di polimerizzazione: ogni ciclo raddoppia la quantità di DNA sintetizzato nel ciclo precedente e quindi la cinetica della reazione è di tipo esponenziale.
Diagnostica
Numerose sono le applicazioni della PCR in ambito diagnostico, in particolare nella individuazione di mutazioni geniche che determinano forme tumorali oppure malattie ereditarie. Se un gene presenta ampie delezioni nucleotidiche, come nel caso del gene mutato nella distrofia di Duchenne (➔ distrofia), il tratto di DNA amplificato di quella regione risulterà più corto rispetto a quello derivato dall’amplificazione del gene normale. Nella diagnosi di distrofia di Duchenne, la PCR è anche utilizzata per amplificare un locus polimorfico di ripetizioni in numero variabile del dinucleotide citosina-adenina (➔ microsatellite), che può essere utilizzato come marcatore associato al gene mutato. Se il gene da amplificare presenta invece il cambiamento di un solo nucleotide (mutazione puntiforme), è possibile mettere in evidenza la variazione mediante una PCR nella quale sia utilizzato un primer contenente una sequenza specifica chiamata ASO (allele-specific oligonucleotide; ➔ oligonucleotide). In ambito oncologico la PCR permette di controllare la comparsa di recidive in pazienti leucemici che siano stati sottoposti a terapia. L’applicazione della tecnica riguarda in maniera particolare i linfomi insorti a causa di traslocazioni cromosomiche che interessano geni noti, come, per es., la traslocazione fra i cromosomi 14 e 18. La PCR può, inoltre, essere impiegata nella diagnosi delle infezioni batteriche e virali.
Filogenesi molecolare
La tecnica della PCR è impiegata negli studi di evoluzione molecolare, al fine di determinare il grado di correlazione fra le specie e costruire alberi genealogici simili a quelli realizzati con i classici metodi comparativi. Viene assunto che tanto più le specie divergono da un antenato comune, tanto più differiscono le loro sequenze nucleotidiche e, viceversa, un maggior grado di conservazione di sequenze nucleotidiche fra specie potrebbe indicare una minore distanza fra di esse dal punto di vista evolutivo. Nell’ambito dell’analisi filogenetica la PCR è inoltre applicata negli studi sull’evoluzione di popolazioni umane, in particolare mediante l’analisi dei siti ipervariabili del DNA mitocondriale.
Mappa del genoma
Un’importante applicazione della PCR nel campo della biologia molecolare riguarda la mappa del genoma, in particolare in quelle metodiche che utilizzano gli ibridi per radiazioni, o RH (radiation hybrid), e i cromosomi artificiali di lievito, o YAC (yeast artificial chromosome) per la costruzione di contigui. Gli RH sono cellule ibride uomo-roditore, ottenute in vitro dalla fusione di cellule umane esposte a radiazioni ionizzanti con cellule di roditore non irradiate. Le radiazioni determinano frammenti di DNA che possono essere incorporati stabilmente nei cromosomi dei roditori e che possono contenere siti cromosoma-specifici, a sequenza unica, amplificabili e quindi identificabili mediante PCR, denominati siti di sequenze-etichetta o STS (sequence tagged sites), con dimensioni di 200-500 paia di basi. Il fondamento teorico della mappatura RH è l’assunto che quanto più due siti sul cromosoma sono vicini l’uno all’altro, tanto maggiore è la probabilità che entrambi siano mantenuti sullo stesso frammento di DNA indotto da radiazioni. Gli STS sono utilizzati anche nella mappatura fisica dei geni mediante YAC: tali cromosomi artificiali di lievito possono contenere porzioni di genoma umano da 100.000 a 1.000.000 di paia di basi. Nell’ambito di tale sequenza può essere messa in evidenza, mediante PCR, la presenza di STS, che possono costituire marcatori per disporre i frammenti di genoma contenuti nei vettori YAC nell’ordine che dovrebbero avere su uno specifico cromosoma (➔ mappa).