operone
(o operon) In genetica, gruppo di geni strutturali adiacenti, che sono trascritti tutti in una singola molecola di RNAm e che sono pertanto soggetti a una regolazione coordinata. In Escherichia coli, i geni che codificano proteine coinvolte nello stesso processo metabolico o i geni che specificano funzioni strettamente correlate, sono regolati in modo coordinato ossia sono attivati, disattivati o modulati finemente da un unico evento regolativo. Spesso i geni regolati in questo modo sono tra loro associati nel genoma e sono trascritti in una unica molecola di RNA a partire dal promotore all’estremo 5′ di tutto il gruppo di geni. L’RNA trascritto viene definito policistronico e l’insieme dei geni espressi coordinatamente prende il nome di operone.
Si distinguono due tipi di o.: quelli che regolano l’attività di sistemi catabolici, cioè di degradazione delle sostanze che vengono dall’ambiente, e quelli che regolano l’attività di sistemi anabolici o biosintetici. Gli enzimi di un processo catabolico sono codificati quando è presente il substrato da digerire e repressi quando tutto il substrato è stato consumato: gli enzimi di un processo anabolico sono invece codificati quando alla cellula occorre un certo prodotto e repressi quando sono stati sintetizzati sufficienti livelli di esso.
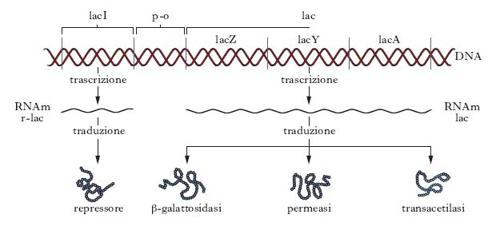
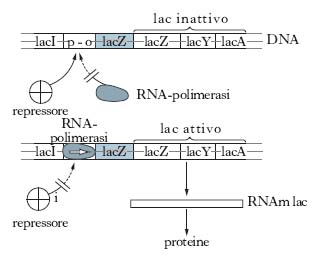
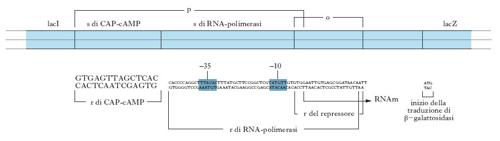
La prima e principale scoperta che ha portato alla comprensione del controllo genico avvenne negli anni 1950 con l’analisi genetica dettagliata, di F. Jacob e J. Monod, degli enzimi del metabolismo del lattosio in Escherichia coli e culminò, nel 1961, nella formulazione del modello dell’o. per la regolazione genica. In larga misura, gli studi sull’o. del lattosio (lac) sono serviti da modello per quelli di altri meccanismi di regolazione genica nei protisti e negli organismi superiori. L’o. lac comprende una regione del cromosoma di Escherichia coli separabile in due porzioni funzionalmente distinte: i geni strutturali e gli elementi di controllo. I geni strutturali codificano tre proteine: la β-galattosidasi, la lattosiopermeasi e la tiogalattoside transacetilasi. La β-galattosidasi (codificata dal gene lacZ) idrolizza i β-galattosidi, in particolare il lattosio, per produrre galattosio e glucosio. La permeasi (codificata dal gene lacY) è associata con i sistemi di trasporto attivo dei β-galattosidi nell’interno della cellula. Il ruolo fisiologico della tiogalattoside transacetilasi (codificata dal gene lacA) non è ancora chiarito. Gli elementi di controllo sono: il promotore (p), sequenza nucleotidica alla quale la RNA-polimerasi si lega per iniziare a trascrivere i tre geni strutturali; il gene lacI che codifica il repressore costituito da una catena pelipeptidica di 37.000 dalton; l’operatore (o), sequenza nucleotidica cui si lega il repressore inibendo, quando occorre, la trascrizione dell’operone (fig. 1). Utilizzando batteri mutati nei geni strutturali, o nel gene regolatore, Jacob e Monod compresero il processo di regolazione dell’o. lac (fig. 2). In assenza di lattosio il repressore blocca il sito operatore e non permette alla polimerasi di legarsi al promotore adiacente, impedendo così la trascrizio;ne dei tre geni strutturali. La presenza di lattosio invece impedisce il legame fra repressore e operatore e permette la trascrizione dei geni strutturali. La diminuzione dell’affinità fra o. e repressore è dovuta all’alterazione della struttura tridimensionale del repressore determinata dal suo legame con il lattosio che, in tal modo, funge da induttore della trascrizione dei tre geni strutturali adiacenti all’operatore. Mutazioni che abbassano l’affinità del repressore per l’o. portano alla sintesi costitutiva degli enzimi dell’o. lac, cioè alla produzione dei tre enzimi sia in presenza che in assenza di induttore. Invece mutazioni che causano l’accumulo del repressore entro la cellula o che ne aumentano l’affinità per l’operatore rendono l’o. lac non inducibile. L’o. lac, per poter essere espresso, richiede anche un segnale positivo, oltre alla necessaria eliminazione della repressione. Questo segnale positivo è fornito da un complesso formato da AMP ciclico (cAMP) e da una proteina chiamata CAP (catabolite activator protein). Il cAMP, che influenza molti processi cellulari, si forma a partire dall’ATP in risposta a una varietà di stimoli extra e intracellulari. Quando CAP, un dimero costituito da due polipeptidi identici, si lega al cAMP, interagisce con una sequenza di DNA situata a monte del promotore, per la quale presenta elevata affinità (fig. 3). Questo legame aumenta la trascrizione dell’o. lac di circa 50 volte, perché determina un aumento della capacità di interazione fra la RNA-polimerasi e il promotore (➔ promotore; trascrizione).
Il complesso CAP-cAMP fornisce un segnale positivo anche per la regolazione dell’espressione di altri o., in particolare per quelli che codificano gli enzimi per la regolazione dei carboidrati. Un tipico o. che regola un’attività di tipo anabolico è l’o. per il triptofano. In Escherichia coli il triptofano viene sintetizzato a partire dall’acido corismico tramite una serie di cinque reazioni catalizzate da cinque complessi enzimatici. Le 5 proteine codificate dall’o. trp sono prodotte in modo coordinato, in quantità quasi uguali, mediante la traduzione di un RNA messaggero policistronico trascritto a partire dal promotore dell’operone. Se può disporre di quantità sufficienti di triptofano per la crescita, Escherichia coli produce in quantità insignificanti gli enzimi necessari alla sintesi di questo amminoacido; in assenza di triptofano, invece, le cellule sintetizzano grandi quantità dei cinque enzimi necessari. A seconda del livello intracellulare di triptofano, la produzione di questo gruppo di enzimi in Escherichia coli può variare di circa 700 volte. La variazione è la conseguenza di due eventi regolativi indipendenti: un meccanismo di repressione, messo in atto per modulare la frequenza dell’inizio della trascrizione a partire dal promotore, e un meccanismo di attenuazione che regola la trascrizione mediante un segnale di termine localizzato fra il promotore e il primo gene strutturale. Il segmento leader, trpL, contiene l’attenuatore, una sequenza nucleotidica con una struttura tale da indurre la RNA-polimerasi a terminare la trascrizione prima dell’inizio del primo gene strutturale.