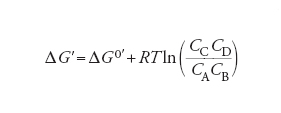Biochimica
Biochimica
La biochimica studia le molecole tipiche della materia o sostanza vivente, le reazioni chimiche che intervengono in essa, i catalizzatori che operano su tali reazioni e le variazioni di energia in gioco. Le molecole che costituiscono la materia vivente sono molto numerose e diverse tra loro e si ritrovano sia all'interno sia all'esterno della cellula, comprese in una categoria che, in prima approssimazione, va sotto il nome di metaboliti. Molte di queste molecole, a basso peso molecolare, sono composti già noti nella chimica organica e in quella inorganica e perciò non del tutto peculiari della materia vivente. È invece una caratteristica particolare della sostanza vivente quella di possedere macromolecole, ossia polimeri particolari ad alto peso molecolare, tra cui le proteine, gli acidi nucleici e i poliosi (amido, glicogeno, cellulosa). Pur raggiungendo notevoli dimensioni, anche le macromolecole biologiche sono costituite da pochi elementi: carbonio, idrogeno, ossigeno, azoto e, in misura minore, fosforo e zolfo. Esse sono considerate polimeri particolari, in quanto la macrodimensione è raggiunta attraverso la successione di unità-base ripetute, unite da specifici legami chimici. Nel caso dei poliosi, l'unità-base è il glucosio. Le molecole di glucosio sono saldate tramite un legame α-glicosidico o β-glicosidico: i poliosi, infatti, svolgono essenzialmente funzione di riserva energetica o di sostegno. Nelle proteine e negli acidi nucleici l'unità ripetitiva non è sempre uguale a se stessa; le proteine sono costituite da 20 differenti amminoacidi, mentre gli acidi nucleici contengono 4 diversi nucleotidi.
Il perché la Natura ricorra, con impressionante ripetitività, alla dimensione macroscopica attraverso l'uso dei polimeri è imputabile alla varietà di funzioni biologiche che devono essere svolte. Per esempio, nel caso degli acidi nucleici è necessaria, in primo luogo, una funzione informativa. Infatti, la molecola di acido nucleico deve essere in grado di immagazzinare una quantità elevata di dati da trasmettere successivamente ad altre molecole, siano esse identiche (replicazione del DNA, trasmissione dei caratteri ereditari) o diverse (sintesi proteica, regolazione dell'espressione genica). Contemporaneamente, è necessaria una poliedricità di funzioni che, nel caso delle proteine, includono la catalisi, il trasporto, il sostegno, la trasduzione dei segnali. Questa poliedricità funzionale implica, in entrambi i casi menzionati, plasticità e specificità. In effetti, è proprio quest'ultima, unitamente alla possibilità di regolare l'evento biologico, a esigere la macrodimensione. Infatti, devono riconoscere il loro substrato in maniera altamente specifica sia l'enzima sia qualsiasi altra proteina delegata a trasmettere un'informazione (o un messaggio); ma la stessa specificità è richiesta all'acido nucleico interagente con una proteina regolatrice. Perciò un enzima deve essere in grado sia di riconoscere la molecola del substrato-legante, tramite interazioni multiple, sia di effettuare le reazioni chimiche necessarie per trasformare il substrato in prodotto. Questi processi di riconoscimento e di trasformazione devono essere regolati, quindi, sia nella proteina sia nell'acido nucleico; sono perciò necessari siti addizionali di legame, sia per molecole di attivatori sia di inibitori. Un tale numero di interazioni non può essere soddisfatto da una molecola di piccole dimensioni, come emerge dalla rilevanza che hanno le macromolecole in tutta la biochimica.
Gli enzimi
Negli organismi viventi avvengono contemporaneamente centinaia di reazioni chimiche, in maniera coordinata e a una velocità compatibile con la vita. Tutto ciò è reso possibile dalla presenza degli enzimi o biocatalizzatori. Sino al 1980 il termine enzima indicava una proteina; attualmente questo termine comprende anche alcuni acidi ribonucleici. Nel caso della proteina, questa può operare da sola o avvalersi dell'aiuto di particolari cofattori, di natura non proteica e di dimensioni molecolari assai ridotte rispetto alla proteina. Molto spesso i cofattori sono derivati delle vitamine e, come gli enzimi, di cui fanno parte, si rigenerano alla fine della reazione. La possibilità di disporre, a partire dal decennio 1950-1960, di un numero sempre più elevato di strutture tridimensionali di proteine enzimatiche, unitamente al continuo affinamento delle tecniche d'indagine cinetica e chimico-fisica, ha permesso di rispondere a molte domande sulle origini del potere catalitico degli enzimi. Infatti, è noto che questi, al pari dei catalizzatori inorganici, possiedono la capacità di aumentare la velocità con cui una reazione raggiunge l'equilibrio, al punto che in molti casi tale velocità può risultare aumentata di un fattore di circa 1011÷1012 rispetto a quella di una reazione non catalizzata.
Gli enzimi sono spesso denominati catalizzatori perfetti perché la velocità della reazione catalitica è dello stesso ordine di grandezza della velocità di diffusione del substrato al centro attivo dell'enzima, rappresentando così il limite massimo della velocità enzimatica. A questo elevato potere catalitico si aggiunge la specificità di reazione, il che significa che l'enzima è in grado di catalizzare la reazione di un solo specifico tipo di substrato o, al massimo, di una classe ristretta di questi, accomunati da chiare analogie chimico-fisiche strutturali. In effetti, la cellula eucariotica, per salvaguardare l'individualità di ogni singola reazione, è strutturata a compartimenti i quali assicurano una specificità di funzioni e di reazioni; tuttavia questa caratteristica da sola non sarebbe sufficiente se non esistessero gli enzimi.
È proprio al potere catalitico e alla specificità d'azione degli enzimi che si è recentemente rivolta l'attenzione degli enzimologi; naturalmente si è tentato anche di decifrare con precisione sempre maggiore i meccanismi della catalisi, cioè quei processi tramite i quali le molecole che costituiscono il substrato vengono trasformate in prodotti. Lo studio del cosiddetto centro attivo, o sito attivo, dell'enzima, cioè il luogo fisico in cui il substrato si lega all'enzima e subisce varie trasformazioni chimiche, ha ricevuto un impulso considerevole dalla risoluzione delle strutture tridimensionali di numerose molecole enzimatiche e dei loro complessi con inibitori o, in opportune condizioni, con substrati. La possibilità, poi, di effettuare a piacimento sostituzioni dei vari residui amminoacidici dell'enzima, coinvolti nel meccanismo di catalisi (tramite la tecnica di mutagenesi sito-specifica), ha contribuito in modo determinante alla sua interpretazione. Si è dunque cominciato a comprendere come sia possibile per un enzima abbassare in modo considerevole l'energia di attivazione di una reazione, aumentando la velocità con cui si raggiunge l'equilibrio fra le molecole del substrato e quelle del prodotto.
Si è avuta altresì conferma della teoria dell'induced fit (adattamento indotto) di Daniel Koshland, secondo la quale l'enzima, nel formare il complesso con il substrato, non si comporta come una molecola rigida, ma va incontro a un cambiamento conformazionale, si modella cioè sul substrato o, meglio ancora, per ragioni termodinamiche, sullo stato di transizione della reazione. La risoluzione ai raggi X della struttura tridimensionale del centro attivo, inoltre, ha confermato e sviluppato le ipotesi già avanzate in precedenza sulla capacità, da parte dell'enzima, di orientare il substrato, di favorirne il legame e di racchiuderne la molecola al proprio interno, attraverso l'azione di alcune sue parti operanti quali veri e propri coperchi flessibili: in tal modo il substrato è sottratto all'azione di agenti competitori. Il vero e proprio meccanismo di trasformazione del substrato è affidato poi alla presenza di vari agenti nucleofili ed elettrofili, rappresentati dalle catene laterali degli amminoacidi dell'enzima, situati in maniera stereochimicamente corretta rispetto alla molecola del substrato.
Lo studio dei meccanismi di catalisi ha portato anche a determinare, con sempre maggior precisione, il cammino cinetico e le costanti catalitiche che regolano la progressione dei vari complessi intermedi della reazione. In questo senso, l'impiego sempre più frequente dell'ingegneria proteica, al fine di produrre in laboratorio, e per scopi industriali, enzimi adatti a determinati compiti, ha introdotto l'uso delle costanti cinetiche: −kcat, la costante catalitica (o numero di turnover), e Km, la costante di Michaelis ‒ come sinonimi di efficienza catalitica di un enzima e come termini di confronto di un enzima rispetto a un altro. Infatti, il rapporto kcat/Km assume il valore di una costante apparente di secondo ordine, che correla la velocità di reazione con la concentrazione di enzima libero ed è valida per ogni concentrazione di substrato; inoltre, per substrati che competono tra loro, essa assume il significato di una vera e propria costante di specificità.
Un'attenzione crescente da parte dei ricercatori è rivolta agli enzimi multicentrici, che in genere sono formati da più subunità e in cui sono presenti più cofattori. Si tratta di enzimi che catalizzano reazioni di trasferimento elettronico intramolecolare dal substrato, attraverso i diversi cofattori, al prodotto. Questa classe di enzimi è l'anello intermedio tra enzimi semplici oppure complessi e i sistemi multienzimatici. In questi ultimi, l'associazione tra più molecole enzimatiche diverse permette di effettuare con maggiore efficienza una determinata trasformazione chimica, o di percorrere più rapidamente una via metabolica (come, per es., la trasformazione del piruvato in acetil-CoA, o la via metabolica di sintesi degli acidi grassi). Con questo sistema, il prodotto della prima reazione viene automaticamente trasferito, quale substrato, al secondo enzima e di qui al successivo, senza essere mai rilasciato a diffondere nel solvente. I sistemi multienzimatici rappresentano una variante costruttiva per aumentare l'efficienza catalitica, tenuto conto dei limiti imposti dalla velocità di diffusione del substrato. Le modalità di azione di un enzima sono varie e complesse, e, a questo riguardo, non si può tralasciare il fatto che molto spesso gli enzimi vengono sintetizzati sotto forma di proenzimi, cioè con una catena polipeptidica più lunga e silente (da un punto di vista catalitico), che viene poi attivata al momento opportuno attraverso un taglio proteolitico e un cambiamento conformazionale della molecola.
L'energia
L'esistenza di macromolecole, o comunque di metaboliti, e quella di enzimi che catalizzano le reazioni a essi collegate non avrebbero alcun significato se non vi fosse associato uno scambio di energia. In questo contesto, preme solo ricordare alcuni elementi essenziali dello scambio energetico che avviene negli organismi viventi. In linea generale, la degradazione dei composti biologici implica, dal punto di vista termodinamico, la liberazione di energia, così come lo svolgersi di reazioni di sintesi comporta un consumo di energia. È noto come nei sistemi biologici l'energia sia necessaria per compiere almeno tre tipi di lavoro: (a) meccanico, che, in breve, è quello impiegato per cambiamenti di luogo o di orientazione; (b) elettrico e di concentrazione, quali la generazione di una differenza di potenziale o il movimento di una molecola o di uno ione contro un gradiente di concentrazione; (c) di sintesi, che coinvolge tutte le operazioni chimiche di costruzione di una nuova molecola più complessa a partire da molecole più semplici. Esempi classici in questo senso sono il lavoro muscolare, la fosforilazione ossidativa, il trasporto di membrana, la sintesi proteica.
Con lo sviluppo della biologia molecolare è venuta alla luce tutta una serie di processi cellulari, largamente associati a scambi di energia: pensiamo ai meccanismi di trasduzione del segnale, al traffico cellulare, al trasporto di membrana, alla respirazione cellulare, ai meccanismi di degradazione delle proteine, al funzionamento degli elementi contrattili, presenti all'interno di ogni singola cellula. Tutti questi processi, all'apparenza così diversi fra loro, vengono ricondotti, dal punto di vista della termodinamica, alla variazione della funzione energia libera di Gibbs. In un sistema in equilibrio a temperatura e pressione costanti l'energia libera è minima e pertanto ogni sua variazione virtuale è nulla. Se il sistema non si trova in condizioni di equilibrio, è soggetto a una trasformazione spontanea associata a una diminuzione di energia libera sino al raggiungimento delle condizioni di equilibrio. Le velocità con le quali le diverse trasformazioni tendono al punto di equilibrio sono influenzate dalla presenza di enzimi, che le rendono compatibili con il tempo proprio dell'organismo vivente. Questi processi includono sia le reazioni di degradazione esoergoniche sia quelle di sintesi per la costruzione di nuove molecole che richiedono apporto di energia.
È in questo contesto che si inseriscono quei composti che sono stati denominati dapprima composti ad alta energia e, successivamente, con più precisione, composti ad alto potenziale di trasferimento di gruppo, tra i quali i più rappresentativi sono l'ATP e altri nucleotidi trifosfati. La loro idrolisi è associata a una reazione di tipo esoergonico, con una variazione di energia libera compresa tra circa −60 e −10 kJ/mol. L'ATP (adenosintrifosfato), il GTP (guanosintrifosfato) e gli altri nucleotidi trifosfati, con un valore di circa 231 kJ/mol, si situano in una zona mediana di questa scala, e perciò possono funzionare da accettori e da donatori di energia libera. Infatti, essi si possono formare a spese di composti energeticamente più ricchi e provvedono alla sintesi di composti energeticamente più poveri, in accordo col principio che in una reazione non è importante il tipo di percorso dell'energia, ma solamente lo stato iniziale e quello finale dei componenti, e che in una serie di reazioni, legate tra loro da intermedi comuni, è possibile sommare algebricamente le singole variazioni di energia libera. In questo modo è possibile accoppiare, con meccanismi diversi, la scissione di un composto ad alto potenziale di trasferimento di gruppo alla sintesi di un composto che non potrebbe formarsi in mancanza di un apporto di energia: è quanto avviene, per esempio, per alcune reazioni della glicolisi. Tutto ciò tenendo presente che, come verrà approfondito in seguito, i processi hanno luogo in sistemi aperti stazionari, che non si trovano in condizione di equilibrio.
La variazione di energia libera associata a una reazione del tipo:
[1] A+B→←C+D
condotta in una soluzione a pH=7, è espressa da:
[2] formula
essendo CA, B… le concentrazioni molari dei diversi componenti, R la costante dei gas e T la temperatura assoluta, ΔG0′ è un particolare valore della variazione di energia libera, detta standard, calcolata in condizioni corrispondenti a valori unitari delle concentrazioni dei reagenti e a pH=7. Essa si può valutare dalla costante di equilibrio Keq della reazione stessa, come segue:
[3] ΔG0′=−RTlnKeq
e i suoi valori per i processi biochimici più importanti sono noti. Agli inizi degli studi di bioenergetica, si pensava che il flusso d'energia attraverso l'uso dei composti ad alto potenziale di trasferimento di gruppo, o tramite l'accoppiamento delle reazioni, fosse solo diretto. L'espressione usata, infatti, era quella di trasferimento a livello di substrato, in quanto l'idrolisi di un composto ad alto potenziale di trasferimento di gruppo dava luogo direttamente alla formazione, per esempio, di ATP.
Con il progredire degli studi, si è scoperto che esiste anche un trasferimento indiretto: tipico il caso della fosforilazione ossidativa, in cui l'ossidazione di un substrato avviene attraverso una serie di componenti intermedi. L'accettore finale è, nella fosforilazione ossidativa mitocondriale, l'O2, che viene ridotto a H2O. In questo caso è in gioco una differenza di potenziale elettrochimico di ossidoriduzione ΔE, che è in relazione con la variazione di energia libera tramite l'equazione:
[4] ΔG0′=−nFΔE′0
dove n è il numero degli elettroni trasferiti, F è la costante di Faraday e ΔE′0 la differenza di potenziale in condizioni standard tra le due coppie di ossidoriduzione. La quantità di energia libera che si rende disponibile in questo tipo di trasferimento indiretto è notevole (la degradazione aerobica del glucosio comporta una liberazione di energia che è maggiore di ca. 20 volte rispetto a quella della degradazione anaerobica) e si traduce nella formazione di un maggior numero di molecole di ATP rispetto al semplice trasferimento diretto. Tenendo poi conto che nella cellula in queste condizioni vige lo stesso tipo di relazione spiegato precedentemente per l'equilibrio chimico, per ogni reazione di scambio di elettroni vale la relazione:
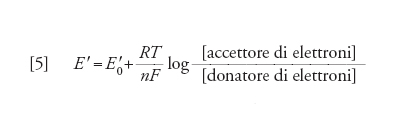
[5] formula
in cui E′0 è il potenziale standard.
In tal modo E′ (a pH=7) rappresenta il potenziale di ossidoriduzione a quel particolare rapporto di concentrazione della specie ossidata e ridotta presente nella cellula, in condizioni diverse da quelle standard. Da tutto ciò consegue che la corrispondente variazione di energia libera tra le due coppie ossidoriducenti può risultare maggiore, portando a un incremento nel numero di moli di ATP formate. Infine, la bioenergetica, oltre a occuparsi delle modalità con cui si effettuano gli scambi di energia, ha compiuto, in questi ultimi anni, progressi considerevoli nel decifrare la struttura e il meccanismo di azione di quelle macchine molecolari che trasformano in situ l'energia chimica con produzione di ATP. Si può ricordare, per esempio, la struttura tridimensionale della molecola della ATP sintasi, l'enzima che catalizza la formazione di ATP sfruttando un gradiente protonico, o la struttura dei complessi fotosintetici, che trasformano l'energia luminosa in energia chimica. Anche in questo caso, la conoscenza della struttura è stata di notevole ausilio a una migliore comprensione del fenomeno biologico.
Il metabolismo
Il metabolismo è stato definito come una serie di reazioni chimiche collegate e azionate da una sorgente esterna di energia. In effetti, l'essenza della biochimica risiede in questa rete estremamente vasta e complessa di reazioni catalizzate, tra loro collegate e regolate da una numerosa serie di attivatori e inibitori. Fino alla metà del Novecento, per metabolismo si intendeva per lo più l'insieme di quelle trasformazioni che negli organismi viventi avvengono a carico dei carboidrati, dei lipidi e delle proteine. Era altresì noto come la divisione della cellula in vari compartimenti ‒ nucleo, citosol, mitocondri, ecc. ‒ fosse strettamente relazionata con le varie fasi del metabolismo di un composto. La glicolisi, la trasformazione del glucosio in piruvato, avviene nel citosol, ma la successiva demolizione del piruvato a CO2 e H2O avviene nel mitocondrio, tramite il ciclo di Krebs. Nella prima metà del secolo era nota l'esistenza di meccanismi di regolazione dei processi metabolici, come gli enzimi allosterici e i cosiddetti cicli di controllo, ma, pensando agli sviluppi attuali del concetto di regolazione di una via metabolica e di trasduzione del segnale, si deve riconoscere che le conoscenze in questo campo hanno fatto enormi progressi.
È con l'avvento della biologia molecolare, e con il conseguente ricorso alle tecniche di espressione genica, che è stato possibile portare alla luce tutto un mondo sconosciuto di componenti cellulari submicroscopici e di molti metaboliti transitori. Si sono così individuate nuove vie metaboliche e si è avuta conferma di come ogni reazione chimica sia a sua volta regolata da altre reazioni, attraverso una complessa rete di interrelazioni che sembra non aver termine e che ha un denominatore comune nelle interazioni proteina-proteina o proteina-acido nucleico. Le vie e i cicli metabolici, così come i concetti di anabolismo e catabolismo, rimangono essenzialmente validi, ma la loro estensione si fa sempre più ampia, unitamente alla loro interpretazione che diviene sempre più complicata. Rimane altrettanto valido il concetto che, per quanto i processi metabolici della cellula siano per la gran parte compartimentalizzati, ciò non equivale a un'assenza di comunicazione tra i vari comparti, come si verifica nei sistemi termodinamici chiusi agli scambi di materia. In effetti, si può ora meglio comprendere quanto detto prima, e cioè che le reazioni chimiche in una cellula non possono essere all'equilibrio (ΔG=0), pena la morte della cellula stessa; infatti, in questo caso verrebbe a mancare la forza trainante dell'energia libera.
Gli organismi viventi evitano questa condizione proprio perché, da un punto di vista termodinamico, non sono sistemi chiusi ma aperti, ai quali si applica una termodinamica di non equilibrio. Più precisamente, gli organismi viventi sono caratterizzati dal fatto di trovarsi in uno stato stazionario, nel quale non esiste accumulo nel tempo dei vari componenti. Infatti, si assiste a un costante flusso di materia o di energia, e in particolare l'energia solare (per es., nel ciclo del carbonio) assume un ruolo preponderante in questo processo. Al mantenimento dello stato stazionario concorre l'insieme dei processi metabolici e, specificamente, dei sistemi di regolazione di tali processi. La conseguenza è che la velocità di sintesi (e di degradazione) dei metaboliti lungo le vie metaboliche è mantenuta costante. Gli enzimi, o meglio la velocità delle reazioni da essi catalizzate rappresentano la variabile mediante la quale si effettua la regolazione di questi processi e che permette il mantenimento dello stato stazionario. È importante sottolineare che non esistono, come si credeva in un primo tempo, punti isolati e distinti di regolazione di una via metabolica; in realtà, è l'insieme di tutte le singole reazioni chimiche che compongono una via metabolica, e, in particolare,è la fine regolazione delle velocità delle reazioni enzimatiche a determinare la costanza di flusso di materia o di energia. Le velocità delle reazioni enzimatiche, a loro volta, sono strettamente dipendenti dalle concentrazioni del substrato-metabolita, ma una regolazione affidata solamente a questo parametro sarebbe di per sé insufficiente per una rete complessa di reazioni quale quella esistente nella cellula. La Natura, in effetti, opera attraverso meccanismi aggiuntivi che, con tempi di risposta diversi, permettono una regolazione molto più precisa della velocità di una reazione.
La biosintesi delle proteine
Alcuni aspetti della sintesi proteica aiutano a comprendere in maniera diretta la complessità dei fenomeni metabolici che hanno luogo negli organismi viventi. La sintesi proteica avviene mediante un sistema a codice che è rappresentato dal DNA. Le 4 basi azotate componenti i nucleotidi del DNA, prese 3 a 3 (le cosiddette triplette), costituiscono le 64 lettere dell'alfabeto (43 combinazioni) per i 20 amminoacidi presenti nelle proteine. Il codice del DNA viene trascritto in un codice complementare di un particolare tipo di RNA, l'RNA messaggero o mRNA, il quale comunica, a livello del ribosoma, il suo messaggio, sempre per via complementare, al tRNA o RNA transfer. Quest'ultimo effettua il processo di traduzione e può essere considerato un vero e proprio adattatore, in quanto reca da un lato della molecola il messaggio complementare in codice (sempre sotto forma di triplette di basi azotate) all'RNA messaggero, mentre dall'altro lato della molecola lega l'amminoacido corrispondente. La complementarità DNA-mRNA-tRNA è data dai legami a idrogeno che uniscono tra loro, in maniera specifica, le basi azotate degli acidi nucleici (citosina-guanina e adenina-timina o adenina-uracile nel caso, rispettivamente, di DNA e RNA). In particolare, la complementarità mRNA-tRNA è la guida per la formazione, a livello del ribosoma, del legame peptidico tra un amminoacido e l'altro, costituendo la catena polipeptidica della proteina.
È difficile dare una risposta al perché la Natura abbia scelto un procedimento a codice per la sintesi delle proteine. La fonte del sistema di replicazione e di sintesi è da ricercarsi nelle origini primordiali dell'RNA, che sembra aver racchiuso in sé la possibilità sia di autoreplicazio-ne sia di attività catalitica. È altrettanto plausibile che la strada della sintesi di una proteina, attraverso l'uso esclusivo di enzimi specifici, sia stata tentata nel corso dell'evoluzione (corte catene polipeptidiche, come quella della gramicidina S, vengono di fatto sintetizzate secondo questa procedura), ma che sia stata tralasciata perché non in grado di assicurare l'enorme varietà delle molecole proteiche e perché enormemente complessa e dispendiosa nel caso della sintesi di proteine a lunga catena polipeptidica. Viceversa, l'uso di un codice permette di immagazzinare una quantità enorme di dati, da convertire in messaggi precisi per la sintesi di un numero estremamente elevato di proteine diverse: è bene comunque precisare che anche un sistema a codice non è sinonimo di semplicità.
Una prima difficoltà è data dalla particolare natura della molecola depositaria delle informazioni, cioè il DNA: la sua struttura a doppia elica, con le catene nucleotidiche disposte in senso antiparallelo, crea problemi alla lettura del codice genetico e la sua lunghezza (ca. 1 m per le 2,9 ∙ 109 paia di basi, nel caso del genoma umano) rende non facile racchiudere il DNA in uno spazio quale quello cellulare, avente dimensioni dell'ordine dei 10−6 m. È proprio per quest'ultima ragione che il DNA è in realtà superavvolto e, nelle cellule eucariotiche, organizzato in speciali formazioni dette nucleosomi. Inoltre, nelle cellule eucariotiche (ma non unicamente in esse), il genoma è organizzato in esoni e introni, in zone, cioè, rispettivamente codificanti per proteine e RNA e zone prive di apparente significato. Agli introni, in un primo tempo era stata assegnata la funzione di facilitare l'assemblaggio degli esoni, e questo in considerazione della particolare struttura a moduli o domini delle proteine. In molte catene polipeptidiche, infatti, si è notata una corrispondenza tra porzioni esoniche del genoma e domini della struttura proteica. Attualmente gli introni sembrano avere un significato più vasto, per esempio, nel caso di molecole di RNA, essi codificano per parti vere e proprie della molecola.
La divisione del genoma in esoni e introni comporta sistemi enzimatici, gli spliceosomi, in grado di riconoscere parti del genoma e catalizzare reazioni di taglio e congiungimento di porzioni genomiche, rispettivamente da scartare o conservare ai fini del successivo processo di traduzione. La ripetitività delle modalità di costruzione e la preservazione della struttura di una proteina, da una generazione cellulare all'altra, richiede la trasmissibilità del codice genetico, processo noto come replicazione del DNA. La replicazione è di tipo semiconservativo, nel senso che ogni filamento del DNA dà origine a un filamento complementare. La lunghezza e la complessità della struttura del DNA rendono difficile la sua replicazione e la successiva trascrizione a RNA messaggero. Queste attività, catalizzate rispettivamente da DNA polimerasi e da RNA polimerasi, richiedono la partecipazione di un elevato numero di proteine ancillari, enzimatiche e non, le quali, per esempio, provvedono a diminuire il grado di superavvolgimento del DNA, a divaricarne i due filamenti e a fornire gli opportuni segnali di inizio e di fine della sintesi.
Ancora più complesso è il processo di trascrizione: infatti, in un determinato momento e all'interno di un genoma dell'ordine di milioni o miliardi di coppie di basi, si deve procedere alla selezione di uno specifico messaggio in codice per una certa proteina o un dato RNA. È quindi necessario un processo di riconoscimento di sequenze specifiche, i promotori, poste immediatamente a monte della sequenza del DNA codificante per quella data proteina. Nei Procarioti la regione del promotore, situata a monte del gene specifico, può comprendere ulteriori messaggi in codice per la sintesi di proteine ancillari, che fungono da attivatori o da inibitori del processo di trascrizione della proteina in questione: l'insieme di questi geni costituisce l'operone. Negli Eucarioti, la trascrizione è ancor più complessa, infatti, oltre ai promotori, ci sono ulteriori sequenze specifiche di riconoscimento, gli enhancers, poste a volte anche a notevole distanza dalla sequenza nucleotidica codificante per la proteina in oggetto. Sequenze di promotori ed enhancers sono il bersaglio specifico per il legame con altrettante proteine, i cosiddetti fattori di trascrizione, che appunto svolgono una funzione regolatrice della trascrizione.
La distanza tra i siti dei promotori e i siti degli enhancers può essere notevole, ma i ripiegamenti del DNA consentono alle proteine regolatrici, che legano questi siti, di entrare in contatto tra di loro e di propagare il loro effetto sul processo della trascrizione. La trasmissione del segnale di regolazione è così operata da interazioni specifiche proteina-proteina, proteina-DNA. Tutto questo permette di avere un sistema di riconoscimento specifico per il codice di una determinata proteina, ma anche, e più in generale, un sistema di regolazione o controllo dell'espressione genica e conseguentemente della sintesi delle proteine. La complessità del sistema a codice non si esaurisce nella trascrizione: a questa fa seguito la traduzione, ovvero la sintesi della catena polipeptidica a livello dei ribosomi, che comprende le fasi di inizio, di allungamento e di terminazione, cui partecipano numerosi enzimi e proteine ancillari.
La sintesi di una proteina, comunque, non termina a questo stadio. Attualmente, infatti, si parla più propriamente di biogenesi di una proteina, a indicare l'insieme di quei processi che intervengono allorché la catena polipeptidica inizia a fuoriuscire dal ribosoma. Essi includono una fase di ripiegamento della catena, il cosiddetto folding, al fine di raggiungere la specifica struttura terziaria. Al folding si associano una serie di modificazioni, denominate post-traduzionali e, per le proteine oligomeriche, il passaggio dalla struttura terziaria a quella quaternaria. Tutte queste operazioni si svolgono in itinere, lungo il percorso che la proteina compie per giungere alla sua destinazione finale, sia essa intracellulare o extracellulare. In particolare, le modificazioni post traduzionali possono avere una funzione di indirizzo all'interno del traffico cellulare, ma anche, e soprattutto, una funzione di rifinitura strutturale in vista dei compiti specifici che la proteina è destinata a svolgere nell'ambito dell'economia cellulare.
La cellula come entità biochimica
Negli ultimi anni si è assistito a una strabiliante evoluzione della biochimica cellulare: essa non si limita a studiare singolarmente l'evento o il processo metabolico (come, per es., il metabolismo del glucosio) isolandoli dal contesto cellulare, ma viceversa si addentra nell'esame delle relazioni che intercorrono tra i vari metaboliti e i processi di sintesi e di degradazione che questi subiscono, allorché sono inseriti in quell'entità in continua trasformazione che è la cellula. Il passaggio dallo studio del singolo evento isolato a quello della complessa rete di fenomeni collegati tra loro è, innanzitutto, il risultato del progredire della ricerca, che pone sempre nuovi e più complessi interrogativi. Questo progresso è dovuto in gran parte all'avvento della biologia molecolare e della biologia strutturale. Queste discipline hanno permesso di scoprire nuovi composti e intermedi chimici, nonché di rilevare, ancora una volta, la fondamentale uniformità di azione delle cellule procariotiche e di quelle eucariotiche, recando così un contributo alla conoscenza dei processi evolutivi.
La cosiddetta trasduzione del segnale comprende tutti quei fenomeni che nella cellula permettono il propagarsi di un segnale e lo scatenarsi di un evento finale a seguito dello stimolo iniziale. Il termine trasduzione sta a indicare come non si tratti di semplice trasmissione, ma, in analogia con quanto avviene in un trasduttore, in molti casi si abbia trasferimento di energia e/o trasformazione di una forma di segnale in un'altra. Lo studio di tali fenomeni trova una sua base storica negli esperimenti miranti a individuare il modo con cui si propaga il segnale ormonale a livello cellulare. A questo proposito gli studi di Earl W. Sutherland jr., insignito nel 1971 del premio Nobel per la medicina, riguardanti l'azione dell'adrenalina sul metabolismo del glucosio, sono risultati paradigmatici perché hanno messo in evidenza un meccanismo che poi si è rivelato il regolatore della trasmissione del segnale a livello cellulare: un susseguirsi di interazioni specifiche proteina-proteina o proteina-acido nucleico.
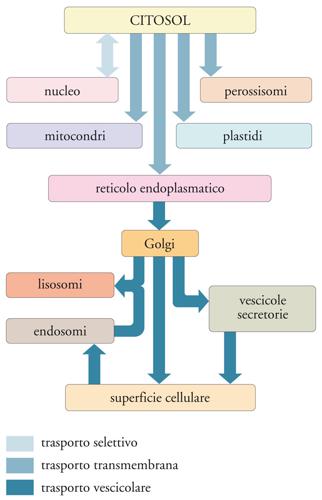
La regolazione del traffico cellulare è il secondo degli aspetti della biochimica cellulare su cui vorremmo soffermarci: con questa espressione intendiamo l'insieme dei processi che dirigono il percorso delle diverse proteine, a partire dal luogo di sintesi a livello dei ribosomi (tralasciamo, in questa sede, le proteine sintetizzate a livello dei mitocondri e dei cloroplasti), sino alla destinazione finale, al loro luogo operativo intracellulare o extracellulare. Nella fig. 5 è mostrata una mappa semplificata delle molteplici vie che una proteina, sintetizzata a livello dei ribosomi, si trova a percorrere a seconda della sua destinazione finale all'interno o all'esterno della cellula. I vari compartimenti cellulari possono essere raggiunti attraverso un trasporto di tipo selettivo transmembrana oppure vescicolare. In alcuni casi (per es., reticolo endoplasmatico e apparato di Golgi), la proteina presenta una sequenza amminoacidica specifica, che funge da sistema di identificazione e di appartenenza a un determinato compartimento. Esistono, inoltre, meccanismi di controllo che provvedono a ricondurre una proteina al proprio compartimento, qualora essa abbia erroneamente percorso vie diverse.
Infine, per quanto riguarda la degradazione delle proteine, questo processo non deve essere confuso con l'apoptosi, o morte programmata della cellula. L'apoptosi è un processo globale, che interessa la vita dell'intera cellula e interviene quando questa ha terminato la sua funzione nell'ambito dell'economia generale dell'organismo o risulta in qualche modo danneggiata. È interessante notare come nel caso dei tumori, in cui la proliferazione cellulare non è più sotto controllo, si assista a un'alterazione dei meccanismi molecolari che regolano l'apoptosi. Tornando alla degradazione delle proteine, questo problema si era posto sin dagli anni Quaranta del Novecento, quando le ricerche di Rudolf Schoenheimer e di altri studiosi avevano messo in evidenza come le proteine endocellulari ed esocellulari possedessero ciascuna un tempo di vita media specifico. Ciò indicava l'esistenza di un'economia di gestione: i processi degradativi delle proteine debbono essere in qualche modo regolati o, se si vuole, ordinati, e ciò presuppone una spesa di energia libera. Quest'ultimo aspetto era il meno evidente: la degradazione di una proteina, coinvolgendo la rottura di un legame peptidico, che è un processo termodinamicamente esoergonico, comportava l'apparente sorpresa della utilizzazione dell'ATP da parte della cellula. D'altro canto, le conoscenze sui meccanismi di trasduzione del segnale hanno fatto comprendere, in retrospettiva, come la presenza di frammenti peptidici, provenienti dalla demolizione di proteine, costituisca un potenziale pericolo per molti processi fisiologici della cellula, basati sul riconoscimento di sequenze-segnale. La cellula necessita quindi di poter disporre di un sistema che rimuova rapidamente tali frammenti attraverso meccanismi idrolitici aventi una precisa collocazione nel tempo e nello spazio.
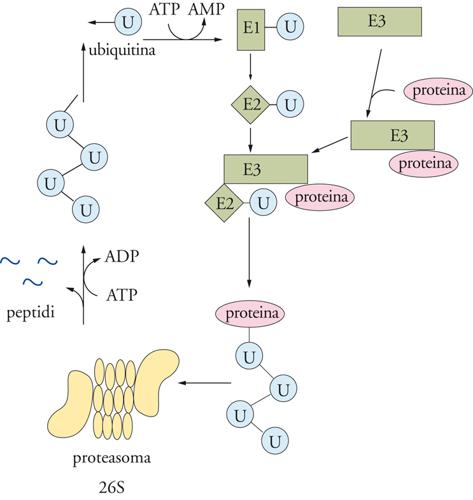
Il principio di un segnale di riconoscimento per la proteina da degradare e la necessità di operare la degradazione stessa in contenitori all'interno della cellula sembrano essere una caratteristica comune. Da tempo era nota la presenza dei lisosomi, organuli cellulari con un ambiente interno decisamente acido (pH≈4,0÷4,5), in cui l'azione endogena di proteinasi acide assicurasse un sistema efficiente di degradazione per tutte quelle proteine che, munite di una sequenza-consenso specifica, pervenissero all'interno del lisosoma stesso. Si è messo chiaramente in luce come al sistema lisosomiale si affianchino altri sistemi degradativi che agiscono nel citosol e quindi, in apparenza, non all'interno di un ambiente chiuso come quello lisosomiale. Negli stessi Procarioti, deficienti di organuli cellulari, è nota, per esempio, la presenza di una proteasi specifica, chiamata La-proteasi, con compiti degradativi. Nelle cellule eucariotiche i sistemi degradativi non lisosomiali possono essere divisi in sistemi ubiquitina-dipendenti e non. L'ubiquitina è una piccola molecola proteica di 76 amminoacidi, che si lega covalentemente alla proteina da degradare. Il processo di ubiquitinazione è complesso, richiedendo l'intervento di almeno 3 enzimi diversi, E1, E2 ed E3 (fig. 6), i quali agiscono in successione e in associazione con il proteosoma. L'associazione ubiquitina-proteina da degradare richiede ovviamente la presenza di un segnale specifico su quest'ultima, che è costituito dalla presenza di particolari amminoacidi all'N-terminale o lungo la sequenza della proteina bersaglio. In quest'ultimo caso, con una certa frequenza si è ritrovata una sequenza caratteristica di residui amminoacidici, la sequenza PEST (residui amminoacidici: prolina, acido glutammico, serina e treonina), che segnala l'inizio del processo di ubiquitinazione e successiva degradazione.
Bibliografia
Alberts 1994: Alberts, Bruce e altri, Molecular biology of the cell, 3. ed., New York, Garland, 1994 (trad. it.: Biologia molecolare della cellula, 3. ed., Bologna, Zanichelli, 1995).
Berg 1981: Berg, Paul, Dissection and reconstruction of genes and chromosomes, ‟Science", 213, 1981, pp. 296-303.
Cooper 1995: Cooper, Geoffrey M., Oncogenes, 2. ed., Boston, Jones and Bartlett, 1995.
Friedberg 1995: Friedberg, Errol C. - Walker, Graham C. - Siede, Wolfram, DNA repair and mutagenesis, Washington ASM, 1995.
Goldberg 1995: Goldberg, Alfred L., Functions of the proteasome. The lysis at the end of the tunnel, ‟Science", 268, 1995, pp. 522-523.
Harold 1986: Harold, Franklin M., The vital force: a study of bioenergetics, New York, Freeman, 1986.
Hartl 1996: Hartl, F. Ulrich, Molecular chaperones in cellular protein folding, ‟Nature", 381, 1996, pp. 571-580.
Huber 1989: Huber, Robert, A structural basis of light energy and electron transfer in biology, ‟EMBO journal", 8, 1989, pp. 2125-2147.
Jacob, Monod 1961: Jacob, François - Monod, Jacques,Genetic regulatory mechanisms in the synthesis of proteins, ‟Journal of molecular biology", 3, 1961, pp. 318-356.
Kalckar 1941: Kalckar, Herman M., The nature of energetic coupling in biological synthesis, ‟Chemical reviews", 28, 1941, pp. 71-178.
Kornberg, Baker 1992: Kornberg, Arthur - Baker, Tania A., DNA replication, 2. ed., New York, Freeman, 1992.
Koshland 1987: Koshland, Daniel E. jr, Evolution of cata-lytic function, ‟Cold Spring Harbor symposia on quantitative biology", 52, 1987, pp. 1-7.
Lipmann 1941: Lipmann, Fritz, Metabolic generation and util-ization of phosphate bond energy, ‟Advances in enzymo-logy", 1, 1941, pp. 99-162.
Maynard Smith, Szathmáry 1995: Maynard Smith, John- Szathmáry, Eörs, The major transitions in evolution, Oxford, Freeman, 1995.
Mullis 1990: Mullis, Kary B., The unusual origin of the polymerase chain reaction, ‟Scientific American", 262, 1990, pp. 56-65 (trad. it.: La scoperta della reazione a catena della polimerasi, ‟Le scienze", 44, 1990, pp. 32-39).
Newsholme, Start 1973: Newsholme, Eric A. - Start, Carole, Regulation in metabolism, London, Wiley-Interscience, 1973.
Nishizuka 1992: Nishizuka, Yasutomi, Signal transduction, ‟Trends in biochemical sciences", 17, 1992, p. 367.
Oliver 1996: Oliver, Stephen G., From DNA sequence to biological function, ‟Nature", 379, 1996, pp. 597-600.
Pain 1994: Mechanisms of protein folding, edited by Roger H. Pain, Oxford, IRL Press, 1994.
Patthy 1995: Patthy, László, Protein evolution by exon-shuff-ling, New York, 1995.
Rechsteiner, Rogers 1996: Rechsteiner, Martin - Rogers, Scott W., PEST sequences and regulation by proteolysis, ‟Trends in biochemical sciences", 21, 1996, pp. 267-271.
Rothman, Wieland 1996: Rothman, James E. - Wieland, Felix T., Protein sorting by transport vesicles, ‟Science", 272, 1996, pp. 227-234.
Srere 1994: Srere, Paul, Complexities of metabolic regulation, ‟Trends in biochemical sciences", 19, 1994, pp. 519-520.
Stryer 1995: Stryer, Lubert, Biochemistry, 4. ed., New York, Freeman, 1995 (trad. it.: Biochimica, 4. ed., Bologna, Zanichelli, 1996).
Thomas 1995: Thomas, Philip J. - Qu, Bao-He - Pedersen, Peter L., Defective protein folding as a basis of human disease, ‟Trends in biochemical sciences", 20, 1995, pp. 456-459.
Walsh 1979: Walsh, Christopher, Enzymatic reaction mech-anisms, San Francisco, Freeman, 1979.
Watson, Crick 1953: Watson, James D. - Crick, Francis H.C., Molecular structure of nucleic acids, ‟Nature", 171, 1953, pp. 737-738.
Watson 1992: Watson, James D. e altri, Recombinant DNA, 2. ed., New York, Scientific American Books, 1992 (trad. it.: DNA ricombinante, Bologna, Zanichelli, 1994).
Watson 1987: Watson, James D. e altri, Molecular biology of the gene, Menlo Park (Cal.), Benjamin/Cummings, 1987 (trad. it.: Biologia molecolare del gene, Bologna 1989), 2 v.
White, Kahan 1994: White, Morris F.-Kahn, C. Ronald, The insulin signaling system, ‟Journal of biological chemistry", 269, 1994, pp. 1-4.