Telomeri e stabilità dei cromosomi
Telomeri e stabilità dei cromosomi
I telomeri sono elementi genetici essenziali per la corretta struttura e funzione dei cromosomi lineari e, in particolare, per la loro stabilità: ne proteggono infatti le estremità da fenomeni di ricombinazione aberrante, degradazione e replicazione incompleta. Le specifiche sequenze di DNA telomerico e le proteine a esse associate formano presso tali estremità un 'cappuccio' che le rende facilmente distinguibili dalle estremità derivate dalla rottura dei cromosomi. In assenza di telomeri, le estremità dei cromosomi ricombinano in modo aberrante, formando cromosomi dicentrici che vanno incontro a ulteriori rotture e ricombinazioni durante la divisione cellulare. I telomeri, inoltre, svolgono un ruolo importante nel posizionamento del cromosoma nell'architettura tridimensionale del nucleo, nella prevenzione della degradazione delle estremità e nella replicazione del cromosoma, nonché nella ricombinazione meiotica. Non è, quindi, sorprendente che nelle cellule normali esistano meccanismi di controllo atti all'identificazione delle estremità cromosomali non protette e che segnali di arresto del ciclo cellulare facciano seguito alla perdita anche di un solo telomero.
La maggior parte delle cellule eucariotiche necessita di una particolare DNA-polimerasi, denominata 'telomerasi', per mantenere invariata la lunghezza del DNA telomerico alle estremità dei cromosomi. Generalmente le cellule umane somatiche sono sprovviste di questo enzima e, con il crescere dell'età replicativa, tendono a perdere in modo graduale i telomeri sia in vitro sia in vivo. Al contrario, la maggior parte delle cellule trasformate o tumorali riattivano la telomerasi durante il processo di immortalizzazione. Queste osservazioni hanno importanti implicazioni per la ricerca, sia di base sia applicata, nel campo delle malattie connesse all'invecchiamento, tra cui il cancro.
La quasi totalità del DNA telomerico eucariotico è costituita da semplici ripetizioni in serie di sequenze ricche di G a ogni estremità 3′ della doppia elica, complementari a regioni ricche di C all'estremità 5′ dell'altro filamento. In alcuni organismi, in determinate fasi del ciclo cellulare, il filamento ricco di G all'estremità 3′ è più lungo e sporge oltre il filamento complementare.
Le regioni telomeriche a singolo filamento ricche di G possono formare strutture particolari, all'interno di un filamento o tra filamenti diversi, basate sul 'quartetto di G'; non è ancora stato chiarito se queste strutture esistano anche in vivo e quale ruolo esse svolgano nella biologia del telomero, ma è possibile ipotizzare che la capacità di formare strutture a 2 o 4 filamenti sia coinvolta nelle specifiche associazioni telomero-telomero o telomero-proteina implicate nell'appaiamento tra cromosomi e nella dinamica del telomero.
Sequenze telomeriche e proteine legate ai telomeri
Le sequenze telomeriche variano da specie a specie, ma tutti i telomeri di un dato organismo presentano le medesime ripetizioni. I Vertebrati possiedono un'unica sequenza telomerica, evolutivamente conservata, alle estremità dei cromosomi (5′TTAGGG3′:5′CCCTAA3′), mentre le sequenze subtelomeriche possono differire. Nell'uomo, è stato stimato che la regione di DNA telomerico a doppio filamento TTAGGG:CCCTAA si estende per 5÷15 kb nelle cellule normali ma è notevolmente più corta in alcune cellule 'anziane' e in molte linee cellulari trasformate e tumorali.
Nei Protozoi, nel lievito e nei Vertebrati sono state identificate diverse proteine in grado di legarsi ai telomeri. La proteina TRF1, che è in grado di legarsi al DNA telomerico umano a doppio filamento, e la cui sequenza codificante è stata clonata, risulta essere coinvolta nella regolazione della lunghezza dei telomeri. Essa è incapace di legarsi a varianti minime della sequenza canonica TTAGGG:CCCTAA ed è probabilmente in questo modo che si spiega il ridotto funzionamento in vivo di sequenze telomeriche alterate. Benché in alcuni Ciliati siano state caratterizzate proteine in grado di legare la sporgenza a singolo filamento ricca di G del telomero, non è ancora stato possibile individuare proteine omologhe nei Mammiferi. Gli studi di genetica condotti sul lievito hanno consentito l'identificazione di molte proteine che interagiscono direttamente o indirettamente con il telomero contribuendo alla stabilità del cromosoma. In particolare, in Saccharomyces cerevisiae, un fattore di trascrizione, RAP1, sembra essere la principale proteina in grado di legarsi ai telomeri. Mutazioni di RAP1 o dei fattori con cui esso interagisce, tra cui le proteine Rif1 e SIR, possono avere un effetto rimarchevole sulla dinamica del telomero, sulla funzionalità del cromosoma e sull'espressione dei geni localizzati in prossimità del telomero stesso. Nei telomeri dei Mammiferi la struttura della cromatina è alterata, probabilmente a causa del legame di proteine specifiche associate ai telomeri o dell'associazione dei telomeri a specifici componenti della matrice nucleare. La regione TTAGGG:CCCTAA più distale ha una struttura cromatinica diffusa che non possiede un'organizzazione nucleosomica canonica, mentre quella più prossimale presenta una spaziatura ridotta dei nucleosomi. Non è chiaro se tali alterazioni strutturali abbiano effettivamente un ruolo biologico nella cellula.
La telomerasi e il problema della replicazione delle estremità
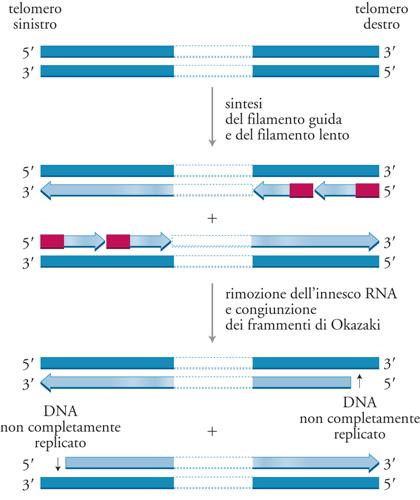
Per apprezzare appieno l'importanza dei telomeri per la stabilità del cromosoma bisogna considerare il cosiddetto 'problema della replicazione delle estremità'. Le estremità di una molecola di DNA lineare a doppio filamento, infatti, non possono essere completamente replicate dal normale complesso della DNA-polimerasi che, per iniziare la sintesi del DNA, richiede un innesco instabile di RNA. Quando il frammento di Okazaki terminale del filamento 'lento' (lagging) del doppio filamento appena replicato viene esteso e legato al frammento di Okazaki più a valle, rimane una regione di DNA non replicato che corrisponde all'estremità 5′ di questo frammento terminale. La lunghezza del frammento non replicato dipende dalla distanza tra il sito di inizio del frammento di Okazaki terminale e l'estremità 3′ del filamento stampo e dalla lunghezza dell'innesco a RNA (fig. 2).
Se non esistessero meccanismi in grado di ovviare al problema della replicazione delle estremità, gli organismi non sarebbero in grado di trasferire alla progenie l'intero patrimonio genetico e, alla fine, andrebbe perduta completamente la funzionalità dei telomeri, causando instabilità cromosomica. Perciò tutte le specie devono possedere, almeno nella linea germinale, un meccanismo in grado di prevenire la replicazione incompleta del proprio genoma. La maggior parte delle specie eucariotiche utilizza la telomerasi, una particolare DNA-polimerasi, per ripristinare ex novo il DNA telomerico, in modo da compensare le delezioni terminali causate dal problema della replicazione delle estremità. In assenza di telomerasi o di preservazione dei telomeri, le cellule eucariotiche, che altrimenti potrebbero essere immortali, vanno incontro a un numero limitato di divisioni prima dell'arresto del ciclo o della morte. Inoltre, è stato dimostrato che la perdita dei telomeri di un singolo cromosoma provoca instabilità cromosomica e arresto del ciclo cellulare.
Struttura e funzione della telomerasi
La telomerasi ha un ruolo fondamentale nel mantenimento dei telomeri e, di conseguenza, nella stabilità dei cromosomi. Questo interessante enzima è stato identificato per la prima volta negli anni Ottanta del Novecento, nel ciliato Tetrahymena, per la sua particolare capacità biochimica di allungare un oligonucleotide di sequenza simile a quella del telomero sintetizzando ex novo DNA telomerico TTGGGG. Poco tempo dopo è stato individuato un enzima umano con proprietà molto simili, ovvero in grado di operare la sintesi di ripetizioni TTAGGG, ma solo nel 1995 è stato identificato dal punto di vista biochimico l'omologo del lievito.
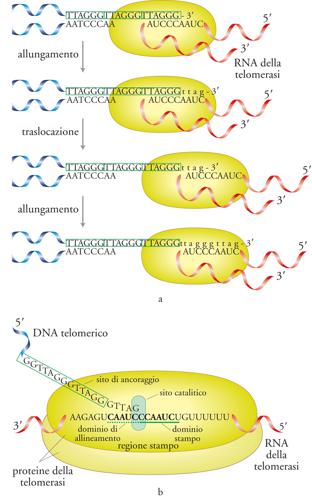
La telomerasi è un complesso ribonucleoproteico (RNP, ribonucleoproteina). Una piccola regione della sua componente a RNA, detta 'regione stampo', è complementare alla sequenza telomerica specie-specifica, e consente l'appaiamento tra l'estremità telomerica di un cromosoma e il sito catalitico del complesso, guidando la sintesi della corretta sequenza di DNA telomerico. La telomerasi è dunque una RNP con attività di trascrittasi inversa: una DNA-polimerasi RNA-dipendente (fig. 3). Si tratta di un enzima 'processivo', ossia in grado di copiare ripetutamente il dominio stampo presente nella componente a RNA senza dissociarsi dalla catena di DNA appena allungata. Dopo essersi legato a un DNA d'innesco, che è in genere la sequenza telomerica, esso agisce mediante cicli composti da una fase di allungamento, in cui i desossiribonucleosidi trifosfati vengono aggiunti sequenzialmente all'estremità 3′ del DNA, e da una fase di 'traslocazione', in cui l'enzima stesso avanza, rispetto al telomero, di un'unità ripetitiva. La copiatura reiterata del dominio stampo spiega la semplice struttura, a moduli ripetuti, del DNA telomerico che si trova alle estremità dei cromosomi della maggior parte delle specie eucariotiche (fig. 4).
La componente a RNA della telomerasi è stata clonata per diverse specie di Ciliati, di lieviti e, recentemente, di Mammiferi. In tutti questi casi, l'autenticità del clone isolato è stata verificata mediante mutagenesi sito-specifica nel dominio stampo del gene che codifica la componente a RNA e successiva dimostrazione che nelle cellule che esprimono il gene mutato la telomerasi sintetizza sequenze telomeriche mutate. Il risultato più importante ottenibile mediante questo tipo di clonaggio è rappresentato dalla possibilità di accertare sperimentalmente se l'inibizione della telomerasi in cellule umane immortalizzate conduca alla progressiva perdita dei telomeri e, dopo un limitato numero di divisioni, alla senescenza o alla morte cellulare. Tale ipotesi è stata verificata in cellule HeLa transfettate con vettori recanti il gene della componente a RNA della telomerasi in orientamento antisenso. In Ciliati e lieviti con attività alterata o ridotta della componente a RNA della telomerasi, si osserva che alla perdita dei telomeri segue una crisi proliferativa con un elevato tasso di mortalità. Nei rari cloni che sopravvivono, la stabilità dei telomeri è ottenuta mediante un meccanismo alternativo che probabilmente coinvolge la ricombinazione. Anche una piccola frazione di linee cellulari umane immortalizzate mostra di possedere un meccanismo che permette di conservare i telomeri indipendentemente dalla telomerasi.
A oggi, sono state clonate sequenze che codificano putative proteine intrinseche della telomerasi di Tetrahymena, ma il loro ruolo nell'attività telomerasica non è ancora stato completamente definito. Analogamente, è stata identificata una proteina associata alla telomerasi dei Mammiferi, ma non le è stata attribuita alcuna attività enzimatica; inoltre, anticorpi specifici contro di essa sono in grado di far precipitare per reazione immunologica solo una piccola frazione dell'attività telomerasica degli estratti cellulari. Poiché quest'enzima può esistere in molteplici stati e sembra avere inoltre un processo di assemblaggio notevolmente complesso, si può ipotizzare che alcune proteine gli si associno soltanto transitoriamente o soltanto in determinati compartimenti cellulari, senza essere tuttavia né essenziali né componenti catalitiche dell'attività enzimatica. Per una piena comprensione della relazione tra telomeri e stabilità del cromosoma è necessario comprendere il ruolo svolto dalle proteine intrinseche e da quelle associate alla telomerasi nella biogenesi, nell'attività enzimatica e nella regolazione della struttura e della funzione del telomero.
Misurazione della lunghezza dei telomeri e dell'attività della telomerasi
Frammenti di restrizione terminali e lunghezza dei telomeri
La tecnica più comunemente usata per stimare la lunghezza dei telomeri si avvale dell'analisi Southern dei frammenti di restrizione terminali (TRF, Terminal restriction fragment); tuttavia, nell'uomo, questi frammenti contengono quantità variabili di DNA non telomerico, ovvero non-TTAGGG, come sequenze degenerate simili a TTAGGG e altre sequenze semplici di DNA (fig. 5). È improbabile che tali sequenze subtelomeriche svolgano un ruolo importante nella normale struttura e nella funzione dei cromosomi, poiché i cromosomi spezzati e successivamente riparati dalla telomerasi ne sono del tutto privi. In ogni caso, la nostra conoscenza ancora parziale delle variazioni intercromosomiche delle sequenze, sia TTAGGG sia non, rende difficile valutare la relazione assoluta tra la lunghezza dei TRF e quella dei telomeri e la lunghezza minima di DNA telomerico funzionante in un determinato cromosoma. Attualmente, allo scopo di misurare la lunghezza della regione di ripetizioni TTAGGG vengono ottimizzate metodologie quantitative in situ, che costituiranno uno strumento importante per la comprensione della relazione tra telomeri e stabilità dei cromosomi.
Attività della telomerasi e TRAP
In estratti cellulari, in presenza di un appropriato sistema tampone e di desossiribonucleosidi trifosfati, la telomerasi umana è in grado di catalizzare l'aggiunta sequenziale di ripetizioni TTAGGG al telomero o a oligonucleotidi di sequenza simile. I prodotti della reazione possono essere identificati mediante misurazione dell'incorporazione di un nucleotide marcato durante l'estensione. Poiché la telomerasi copia ripetutamente lo stampo di 6 nucleotidi, il prodotto della reazione di estensione produce, in elettroforesi su gel, una 'scaletta' di bande distanti l'una dall'altra appunto 6 nucleotidi. La formazione di questa caratteristica scaletta a 6 nucleotidi può essere inibita da un pretrattamento dell'estratto con RNasi, in quanto è la componente a RNA della telomerasi a svolgere la funzione essenziale di stampo per la sintesi del telomero. Un saggio molto sensibile e riproducibile di valutazione dell'attività telomerasica, il TRAP (Telomere repeat amplification protocol), utilizza la reazione a catena della polimerasi (PCR, Polymerase chain reaction) per amplificare il prodotto della telomerasi prima della sua espressione. Con il TRAP è possibile evidenziare la presenza di 1÷10 cellule positive al saggio tra più di 104 cellule negative.
Telomeri e telomerasi in cellule e tessuti
La messa a punto del TRAP ha reso possibile saggiare l'attività telomerasica in una serie di cellule e tessuti sia in vitro sia in vivo. La maggior parte delle cellule somatiche umane normali non presenta una significativa attività telomerasica e risente, pertanto, del problema della replicazione delle estremità. I dati confermano ed estendono studi precedenti in cui era stata dimostrata la sostanziale mancanza di attività telomerasica in fibroblasti, cellule embrionali di rene (in coltura), linfociti e cellule epiteliali umane. Un'attività telomerasica è stata, al contrario, rilevata nei tessuti riproduttivi maschili e femminili in alcune popolazioni di cellule staminali, nonché in un gran numero di linee cellulari e tessuti tumorali, coerentemente con l'allungamento della vita (se non addirittura con l'immortalizzazione), che caratterizza almeno una parte di queste popolazioni cellulari. È stato ipotizzato che una forte repressione della telomerasi nella maggior parte delle cellule somatiche sia stata selezionata in specie, come l'uomo, contraddistinte da una vita media molto lunga, quale meccanismo per contenere la frequenza del cancro.
L'ipotesi telomerica della senescenza e dell'immortalizzazione cellulare
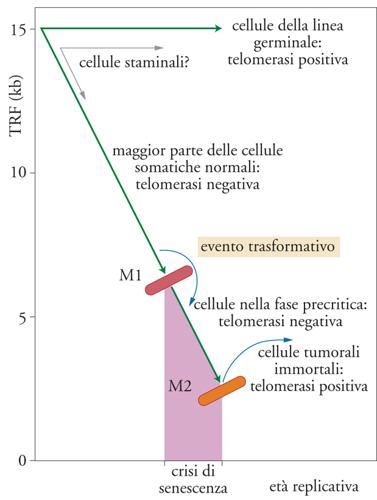
L'ipotesi del ruolo giocato dai telomeri nella senescenza e nell'immortalizzazione cellulare è rappresentata in modo schematico nella fig. 6. I dati sperimentali a sostegno di tale modello sono i seguenti: (a) in tessuti riproduttivi maschili e femminili la telomerasi è facilmente rilevabile e cellule della linea germinale possiedono lunghi telomeri costantemente preservati; (b) in cellule somatiche postnatali, incluse le popolazioni di cellule staminali, non può essere rilevata attività telomerasica, se non transitoriamente o a livelli molto bassi; inoltre, i telomeri si accorciano progressivamente con l'età sia in vitro sia in vivo; (c) in popolazioni di cellule somatiche 'precrisi', trasformate con oncoproteine virali, l'attività telomerasica non può essere rilevata e i telomeri diventano sempre più corti; (d) nella maggior parte delle linee cellulari tumorali e trasformate 'postcrisi', ovvero immortalizzate, si rileva attività telomerasica e la lunghezza dei telomeri è costantemente mantenuta (benché essi siano, generalmente, piuttosto corti).
Il modello illustrato nella fig. 6, comunque, rappresenta qualcosa di più che non semplici osservazioni fenomenologiche: esso lega infatti in modo causale da un lato la perdita di telomeri e la senescenza cellulare (M1), dall'altro la riattivazione della telomerasi e il cancro. Cruciale è in questo senso l'importanza dei telomeri per la stabilità genomica. Le cellule invecchiate mostrano un'elevata frequenza di cromosomi dicentrici, ed esiste una correlazione molto forte tra l'apparente lunghezza critica dei TRF e l'instabilità genomica durante la crisi proliferativa di cellule trasformate. È possibile che al momento della senescenza solo uno o pochi telomeri abbiano raggiunto la lunghezza soglia che induce una certa instabilità genomica, ma generalmente ciò è sufficiente come segnale di arresto del ciclo cellulare per danno al DNA. Al momento della crisi (M2), molti telomeri possono aver raggiunto la lunghezza critica e causare quindi una marcata instabilità genomica nella popolazione cellulare, contribuendo probabilmente a due caratteristiche della cancerogenesi: la perdita di eterozigosi e l'amplificazione genica. L'attivazione della telomerasi e la stabilizzazione dei telomeri permettono alle cellule di sopravvivere alla crisi e di ripristinare una certa stabilità genomica. È inoltre noto che la perdita dei telomeri e l'instabilità genomica sono coinvolti in almeno una malattia genetica, l'atassia-teleangiectasia.
Prospettive: telomeri e patologie umane
La mancanza di telomerasi nella maggior parte dei tessuti somatici, causa della perdita progressiva di telomeri culminante nella senescenza cellulare, e la frequente comparsa di attività telomerasica nei tessuti maligni aprono interessanti prospettive per lo sviluppo di nuove terapie per il trattamento delle patologie legate all'età, tra cui il cancro. La riattivazione della telomerasi in modo transitorio o regolato, così come il rallentamento con altri mezzi della perdita dei telomeri, potrebbe aumentare la durata della vita replicativa delle cellule somatiche, offrendo indubbi vantaggi terapeutici in particolar modo nel caso di sindromi da invecchiamento precoce o da invecchiamento di tessuti in individui sani. Esistono diversi tessuti in cui la senescenza delle cellule che si replicano ha un ruolo chiave nella patologia di malattie correlate all'età, tra cui: i fibroblasti del derma nell'invecchiamento della pelle (diminuzione della capacità di cicatrizzazione e predisposizione a ulcere); le cellule endoteliali nell'invecchiamento vascolare (malattie cardiovascolari); le cellule epiteliali pigmentate della retina (degenerazione maculare correlata all'età); gli osteoblasti nell'osso (osteoporosi); i linfociti nel sangue periferico (predisposizione alle infezioni).
Viceversa, la destabilizzazione della struttura e della funzione dei telomeri, specialmente in cellule malate, potrebbe indurre instabilità cromosomica e morte cellulare e, quindi, costituire un nuovo approccio al trattamento di patologie proliferative come il cancro. L'inibizione della telomerasi rappresenta una soluzione particolarmente attraente per via dell'universalità dell'attivazione di questo enzima nei tumori umani e della sua scarsa o nulla attivazione nei tessuti normali. Prima che tale strategia possa essere utilizzata per il trattamento del cancro umano, tuttavia, sono necessari ulteriori ricerche, e in particolare lo sviluppo di agenti farmacologici, che inibiscano efficientemente e specificatamente la telomerasi, e indagini più accurate riguardanti le conseguenze biologiche della sua inibizione in vivo in tessuti normali e tumorali. Il potenziale impatto di questi studi sulla medicina è di così ampia portata che non c'è dubbio che la ricerca sui telomeri, e sulle proteine e sugli enzimi che sono coinvolti nel loro mantenimento, procederà a ritmo sempre crescente. La caratterizzazione di tutte le proteine associate ai telomeri, incluse le componenti della telomerasi, il clonaggio dei relativi geni e lo sviluppo di anticorpi e di molecole ricombinanti potranno fornire nuovi strumenti per la comprensione di questi importanti determinanti della stabilità cromosomica e del modo in cui essi possono essere utilizzati per il trattamento di varie patologie umane.
Bibliografia
Bacchetti, Counter 1995: Bacchetti, Silvia - Counter, Christopher M., Telomeres and telomerase in human cancer, "International journal of oncology", 7, 1995, pp. 423-432.
Blackburn 1992: Blackburn, Elizabeth H., Telomerases, "Annual review of biochemistry", 61, 1992, pp. 113-129.
Collins 1995: Collins, Kathleen - Kobayashi, Ryuji - Greider, Carol W., Purification of Tetrahymena telomerase and cloning of genes encoding the two protein components of the enzyme, "Cell", 81, 1995, pp. 677-686.
De Lange 1995: De Lange, Titia, Telomere dynamics and genome instability in human cancer, in: Telomeres, edited by Elizabeth H. Blackburn, Carol W. Greider, Plainview (N.Y.), Cold Spring Harbor Laboratory Press, 1995, pp. 265-293.
Feng 1995: Feng, Jiuhuan, The RNA component of human telomerase, "Science", 269, 1995, pp. 1236-1241.
Greider, Blackburn 1985: Greider, Carol W. - Blackburn, Elizabeth H., Identification of a specific telomere terminal transferase activity in Tetrahymena extracts, "Cell", 43, 1985, pp. 405-413.
Hanish 1994: Hanish, John P. - Yanowitz, Judith L. - De Lange, Titia, Stringent sequence requirements for the formation of human telomeres, "Proceedings of the National Academy of Sciences USA", 91, 1994, pp. 8861-8865.
Harley 1990: Harley, Calvin B. - Futcher, A. Bruce - Greider, Carol W., Telomeres shorten during ageing of human fibroblasts, "Nature", 345, 1990, pp. 458-460.
Harley 1991: Harley, Calvin B., Telomere loss: mitotic clock or genetic time bomb?, "Mutation research", 256, 1991, pp. 271-282.
Harley 1994: Harley, Calvin B. e altri, Telomerase, cell immortality, and cancer, "Cold Spring Harbor symposia on quantitative biology", 59, 1994, pp. 307-315.
Harrington 1997: Harrington, L. e altri, A mammalian telomerase-associated protein, "Science", 275, 1997, pp. 973-976.
Kipling 1995: Kipling, David, The telomere, Oxford, Oxford University Press, 1995.
Morin 1989: Morin, Gregg B., The human telomere terminal transferase enzyme is a ribonucleoprotein that synthesizes TTAGGG repeats, "Cell", 59, 1989, pp. 521-529.
Watson 1972: Watson, James D., Origin of concatemeric T7 DNA, "Nature. New biology", 239, 1972, pp. 197-201.
Wright 1996: Wright, Woodring E. e altri, Experimental elongation of telomeres extends the lifespan of immortal x normal cell hybrids, "EMBO journal", 15, 1996, pp. 1734-1741.
Zakian 1995: Zakian, Virginia A., Telomeres: beginning to understand the end, "Science", 270, 1995, pp. 1601-1607.