Manipolazioni di molecole
Manipolazioni di molecole
La manipolazione e lo studio di singole molecole sono diventate aree centrali di ricerca della biofisica moderna. Il recente sviluppo delle nanotecnologie ha permesso di progettare e sviluppare tecniche sperimentali e strumenti scientifici di grande precisione, capaci di visualizzare e manipolare singole molecole così come di misurare forze microscopiche. La manipolazione di singole molecole consente di caratterizzare le proprietà molecolari e di quantificare la cinetica delle reazioni biochimiche. Queste tecniche aprono le porte all'osservazione di nuovi fenomeni difficilmente accessibili con i metodi di analisi biochimici tradizionali. Di seguito verranno introdotte le descrizioni delle diverse metodologie e applicazioni degli esperimenti di manipolazione su singole molecole. Si accennerà brevemente alla microscopia a forze atomiche o AFM (Atomic force microscopy), alle pinze ottiche o OT (Optical tweezers) e alla fluorescenza di singole molecole o SMF (Single molecule fluorescence). Verranno poi presentate le diverse applicazioni di queste tecniche allo studio degli acidi nucleici e delle proteine, e a quello della termodinamica di non-equilibrio nei piccoli sistemi.
La biofisica è una disciplina a cavallo tra la fisica, la chimica e la biologia, che si occupa dello studio delle proprietà fisico-chimiche e della funzione biologica delle diverse componenti cellulari. La biofisica molecolare è una branca della biofisica, che studia la struttura e la funzione biologica di complessi biomolecolari. Dall'analisi degli enzimi, che mostrano diverse reazioni chimiche, fino ai complessi macromolecolari, che costituiscono l'architettura delle membrane cellulari, passando dallo studio delle biomolecole essenziali nella trasmissione di segnali chimici all'interno della cellula, la biofisica molecolare rappresenta uno dei terreni più ricchi e interdisciplinari per la comprensione scientifica della complessità e l'organizzazione della materia vivente.
La manipolazione di singole molecole offre la possibilità di studiare dettagliatamente le proprietà fisico-chimiche e la funzione biologica delle molecole. Ciò è possibile grazie allo sviluppo negli ultimi decenni di strumenti scientifici sufficientemente sensibili e precisi da poter manipolare molecole, misurare forze dell'ordine dei piconewton (1pN=10−12 newton) e distanze dell'ordine dei nanometri (1 nm=10−9 m). Queste tecniche consentono di studiare la risposta meccanica delle molecole ‒ seguendo la loro evoluzione in tempo reale mentre partecipano alla catalisi di reazioni chimiche ‒ o di identificare le loro variazioni conformazionali. Gli esperimenti su singole molecole o SME (Single molecule experiments) sono complementari alle tecniche tradizionali della biologia strutturale e molecolare, come la spettroscopia e la microscopia. Inoltre, essi forniscono moltissime informazioni e dati per la ricerca quantitativa di processi biofisici tramite l'utilizzo di metodi statistici. Infine, permettono l'osservazione di nuovi fenomeni nella biofisica molecolare e cellulare così come la verifica di modelli e teorie della fisica statistica.
Perché singole molecole?
Quali vantaggi offrono le tecniche di manipolazione di singole molecole paragonate alle tecniche biochimiche tradizionali bulk? La differenza principale tra le tradizionali tecniche biochimiche bulk e gli esperimenti su singole molecole o SME consiste nel tipo di media eseguito sulle molecole. Mentre i metodi tradizionali misurano i valori medi di certe proprietà molecolari ottenute da un numero macroscopico di molecole, gli SME permettono di caratterizzare le proprietà di una singola molecola, il che consente di ottenere distribuzioni di probabilità associate a esse, fornendo un'informazione aggiuntiva a quella contenuta nei valori medi di suddette distribuzioni, talvolta non facilmente ottenibili con le analisi biochimiche bulk. Tutto ciò è supportato da tecniche di visualizzazione, che aiutano molto nella comprensione e nell'analisi degli esperimenti.
Un esempio dei vantaggi forniti dagli SME si trova nello studio del ripiegamento delle proteine. Le proteine in soluzione acquosa solitamente si presentano in due configurazioni o stati possibili. Nello stato iniziale o ripiegato, anche detto stato nativo (N), la molecola è biologicamente attiva. Nello stato denaturato o non ripiegato (D), la molecola forma una struttura simile a quella di una random coil (letteralmente bobina casuale) ed è biologicamente inattiva. La transizione tra questi due stati può avvenire per riscaldamento o per diluizione di sostanze denaturanti nella soluzione acquosa (per es., modificando il pH con l'aggiunta di urea). Riscaldando la soluzione acquosa si osserva come la proteina passi dallo stato iniziale a quello denaturato a una temperatura caratteristica Tp, denominata anche temperatura di ripiegamento. Il cambio di stato della proteina è reversibile ed è associato a un calore latente caratteristico delle transizioni di primo ordine. È possibile caratterizzare questa trasformazione utilizzando misure sperimentali con tecnica bulk come, per esempio, la calorimetria o l'assorbimento UV, che mostrano un massimo del calore specifico e un punto di inflessione dell'assorbimento nella zona di transizione. In molte proteine si può anche riscontrare l'esistenza di uno stato intermedio (I) intorno alla temperatura Tp, che si manifesta per la presenza di un ulteriore massimo nella curva del calore specifico in funzione della temperatura corrispondente alle due transizioni D→I e I→N. L'identificazione degli stati I è cruciale per la comprensione del meccanismo di ripiegamento delle proteine, perché si ritiene che questi stati rappresentino un passaggio obbligato durante il processo di ripiegamento della proteina verso lo stato funzionale N. L'osservazione diretta dello stesso dipende dalla durata della sua vita media e dalla frequenza con cui la proteina permane nello stato I nel suo cammino verso lo stato ripiegato N. È difficile individuare la sua esistenza negli esperimenti con tecnica bulk: spesso lo stato I ha una vita breve e dunque solamente una piccolissima frazione di proteine occupa questo stato. Il segnale prodotto nella provetta dalla minuscola frazione di proteine nello stato I è mascherato dal segnale prodotto dal gran numero di proteine che occupano gli stati N o D.
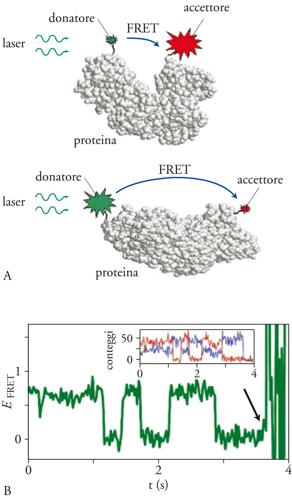
Quali vantaggi offrono gli SME? Negli SME è possibile separare i segnali corrispondenti ai possibili stati della proteina (D, I, N) in diverse famiglie o gruppi. Una delle tecniche sperimentali che permette l'osservazione di variazioni strutturali nelle proteine in tempo reale è il trasferimento di energia a risonanza di fluorescenza o FRET (Fluorescence resonance energy transfer), nota anche come risonanza di Foster. Questa tecnica consiste nell'unire due molecole fluorescenti o fluorofori (definiremo verde il donatore e rosso l'accettore) in specifiche posizioni della catena proteica. Quando la proteina è irradiata con una luce di una certa lunghezza d'onda, il fluoroforo verde riemette per fluorescenza parte della radiazione incidente e trasmette il resto per risonanza al suo compagno rosso, il quale a sua volta riemette una radiazione a una lunghezza d'onda maggiore. La distanza tra i due fluorofori dipende dalla conformazione della proteina e determina l'efficienza con cui la luce viene trasmessa dal fluoroforo verde al rosso (minore è la distanza, maggiore sarà il trasferimento di energia).
Il rapporto tra le intensità di luce emesse dal fluoroforo verde (ID) e rosso (IA) definisce l'efficienza del trasferimento EFRET= IA/(IA+ID). Se la proteina cambia conformazione, la distanza tra fluorofori si modifica e così anche le intensità di luce ID e IA , fino a quando uno dei fluorofori diventa inattivo (photobleaching) e la proteina non emette più quella frequenza (fig. 2). Se la proteina presentasse uno stato intermedio, si darebbe la condizione in cui la luce emessa per ogni frequenza assuma tre possibili valori corrispondenti agli stati D, I, N. La luce emessa a ogni lunghezza d'onda provocherebbe transizioni tra i suddetti stati. Il tempo di permanenza in ognuno degli stati è una misura diretta dei tempi di vita media di ogni stato, ovvero della sua probabilità di occupazione. L'alta risoluzione temporale delle tecniche di fluorescenza abbinata alla possibilità di studiare singole molecole permette l'individuazione di stati intermedi con un tempo di vita molto breve. Il limite degli esperimenti con tecnica bulk non è nella risoluzione temporale, bensì nella difficoltà di separare il segnale emesso da una minuscola frazione di molecole da quello emesso dalle restanti.
Tecniche sperimentali
Negli SME si adoperano due tipi di tecniche. Da un lato, ci sono quelle che permettono la manipolazione delle molecole e la misurazione delle forze. Dall'altro, ci sono le tecniche per visualizzare e ottenere immagini delle molecole che non consentono però una manipolazione diretta. L'esempio della proteina, descritto precedentemente, corrisponde alla seconda categoria degli SME. Tra le tecniche della prima categoria si distinguono: il microscopio a forze atomiche o AFM (Atomic force microscope), le pinze ottiche od OT (Optical tweezers) e le pinze magnetiche o MT (Magnetic tweezers). Esistono anche tecniche a sonda di biomembrana e le microfibre di vetro, ma sono di uso meno frequente di altre. Tra le tecniche della seconda categoria si distinguono la fluorescenza di singola molecola o SMF (Single molecule fluorescence), la spettroscopia Raman e la spettroscopia a due fotoni. La combinazione di tecniche che permettono di manipolare e osservare molecole simultaneamente è una delle sfide sperimentali più promettenti nel campo degli SME. Descriviamo di seguito i principî sui quali si basa ciascuna di queste tecniche.
Microscopio a forze atomiche
Il principio di funzionamento dell'AFM è relativamente semplice. Questo microscopio è stato sviluppato da Gerard Binnig, Calvin Quate e Christoph Gerber nel 1986, nel corso di una collaborazione tra i laboratori IBM di San José e l'Università di Stanford, in California. L'AFM è costituito da una punta di metallo mobile che agisce come sonda per determinare la rugosità di una superficie. Quando la punta si avvicina al substrato viene deviata in modo proporzionale alla distanza che esiste tra essa e la superficie. L'applicazione più importante dell'AFM è nella produzione di immagini degli spessori delle superfici, una tecnica di caratterizzazione di superfici utilizzata in molti laboratori del mondo. L'AFM può anche essere impiegato per la manipolazione di singole molecole. In questo caso la punta dell'AFM e la superficie del substrato vengono ricoperti chimicamente (generalmente con mica oppure oro). Per far ciò il substrato viene ricoperto con le molecole che si desidera manipolare. Si accosta quindi la punta alla superficie finché un'estremità della molecola ancorata alla superficie aderisce alla punta. Se la posizione del substrato è spostata rispetto alla punta, la molecola si distende. La deviazione subita dalla punta permette di misurare la forza che agisce sulla molecola, previa calibratura della rigidità della punta. Questo consente di determinare le proprietà elastiche della molecola attraverso le curve di forza-deformazione, che rappresentano la forza agente sulla punta in funzione della distanza tra la punta e il substrato, corrispondente alla deformazione molecolare.
Lo spettro di forze misurabili con l'AFM va dalle decine di pN fino a vari nN. Questo intervallo è troppo elevato per lo studio di interazioni deboli nel campo della biofisica molecolare, ma ideale nello studio di interazioni covalenti, come per esempio l'elasticità molecolare, o anche di interazioni deboli multiple. L'AFM viene utilizzato per misurare interazioni inter- e intramolecolari, per esempio nella distensione di polisaccaridi, di proteine e di acidi nucleici.
Pinze ottiche
Il principio delle OT si basa sulla forza di gradiente ottico scoperta da Arthur Ashkin nel 1970. L'origine di questa forza sta nella rifrazione o cambiamento di momento lineare della luce quando cambia mezzo. Se su un oggetto refrattario, come per esempio una microsfera immersa in una soluzione acquosa e con un indice di rifrazione maggiore dell'acqua, incide un fascio di luce, allora la forza che agisce su di essa è uguale alla variazione di momento lineare del fascio di luce incidente. Inoltre, questa forza è diretta verso la regione di massima intensità della luce. Se un fascio di luce laser è focalizzato su un punto nello spazio, il profilo di intensità intorno al punto focale sarà di tipo gaussiano e potrà essere utilizzato come trappola ottica per catturare la microsfera al centro del fuoco. In prima approssimazione il potenziale ottico confinante è di tipo armonico ed è quindi possibile utilizzarlo come sensore di forza. La forza è determinata attraverso la relazione F=kx, misurando la distanza x della microsfera al centro del fuoco ‒ per esempio, utilizzando una camera CCD (Charge-coupled device) ‒ e fissando il valore di k (calibrato dalla trappola ottica). Esiste, inoltre, una forza di dispersione ottica associata alla forza di gradiente (scoperta da James C. Maxwell alla fine del XIX sec., che causa la scia di polvere nella coda delle comete quando queste si avvicinano al Sole). Se la potenza del laser è troppo elevata, la forza di dispersione potrebbe essere superiore alla forza di gradiente, il che causerebbe la fuga della microsfera. Per ridurre la componente di dispersione e aumentare quella del gradiente vengono utilizzati obiettivi di grande apertura numerica e fasci di luce contropropaganti.
Nello schema sperimentale di base delle OT una molecola è unita a due microsfere (per es., attraverso legami biotina-streptavidina). Ciò richiede una modifica chimica delle estremità della molecola e il rivestimento chimico della superficie delle microsfere. Una microsfera rimane ferma sulla punta di una micropipetta, la cui posizione si regola con un cristallo piezoelettrico, mentre l'altra viene catturata con la trappola ottica. Agendo sul cristallo piezoelettrico si può muovere la micropipetta effettuando distensioni. Ciò permette di misurare le proprietà elastiche della molecola con le curve forza-estensione (CFE), sulle quali viene rappresentata la forza che agisce sulla microsfera nel pozzo in funzione della distanza tra le due microsfere, che è uguale alla distensione molecolare (fig. 3). Le OT misurano forze tra lo 0,1 pN e i 200 pN con una risoluzione in forza dell'ordine di decimi di pN. Quest'ordine di grandezza della forza è ideale per lo studio di interazioni deboli nella biofisica molecolare. Attualmente, la risoluzione spaziale delle OT può raggiungere vari Ångstrom, il che la rende una delle tecniche più promettenti degli SME. Le OT sono state usate per lo più nella manipolazione di acidi nucleici e nei motori molecolari.
Pinze magnetiche
Una delle tecniche più versatili è l'utilizzazione delle pinze magnetiche. Il principio base di questo strumento è l'uso di un gradiente di campo magnetico per poter applicare delle forze su una microsfera magnetica, F=−μgradB. Uno dei maggiori vantaggi delle MT è che esse permettono di controllare la forza, mentre lo svantaggio è dovuto alla bassa risoluzione associata alla scarsa rigidità della trappola. Lo schema sperimentale di base delle MT è come quello delle tecniche descritte in precedenza. Per la manipolazione, si unisce chimicamente un'estremità della molecola a una microsfera magnetica e l'altra a una superficie di vetro fissata a una piattaforma mobile. Le molecole si allungano, muovendo la piattaforma con un piezoelettrico. Le fluttuazioni trasversali nella posizione della microsfera 〈Δx2〉 sono misurate con una camera CCD e determinano il valore della forza che agisce sulla molecola attraverso il principio di equipartizione, F = kBTL/〈Δx2〉, dove L è l'allungamento molecolare. Inoltre, le MT permettono di applicare una coppia di forze col fine di avvolgere molecole come il DNA intorno al suo scheletro (backbone). Le MT sono state applicate ampiamente per la misura delle proprietà elastiche e di torsione delle molecole di DNA.
Fluorescenza di singola molecola
La SMF si basa sulla rilevazione della luce emessa da molecole fluorescenti (fluorofori) legate con la molecola che si vuole studiare. Il vantaggio della fluorescenza risiede nella sua elevata risoluzione temporale (solitamente μs, talvolta anche ps) e nella grande sensibilità nell'individuazione di fotoni (fino a 103 fotoni al secondo) usando fotodiodi a valanga o fotomoltiplicatori. Una delle grandi applicazioni della SMF è la già citata FRET. L'efficienza di Forster, E, è data dalla formula E=E0/[1+(R/R0)6], dove R è la distanza tra il fluoroforo donatore (verde) e quello accettore (rosso), E0 è una costante e R0 è una distanza caratteristica (solitamente nell'ordine di vari nm). I valori E0 e R0 dipendono dagli spettri di emissione/assorbimento dei fluorofori e dalla relativa orientazione dei dipoli elettrici di ambedue le molecole. L'efficienza di Forster stabilisce una regola spettroscopica per la determinazione delle distanze. Quando una molecola subisce una variazione conformazionale, il valore E varia in seguito al cambio della distanza tra i fluorofori, il che facilita la determinazione del cambio di struttura molecolare. La SMF presenta anche alcuni inconvenienti, come la difficoltà chimica che comporta l'unire fluorofori a punti specifici della molecola, l'incognita dell'orientamento dipolare dei fluorofori e la degradazione degli stessi dopo la reazione chimica con altre molecole (photobleaching). La SMF è stata impiegata nello studio del trasporto molecolare, del ripiegamento di proteine e dei cambi conformazionali associati a reazioni enzimatiche.
Applicazioni degli SME
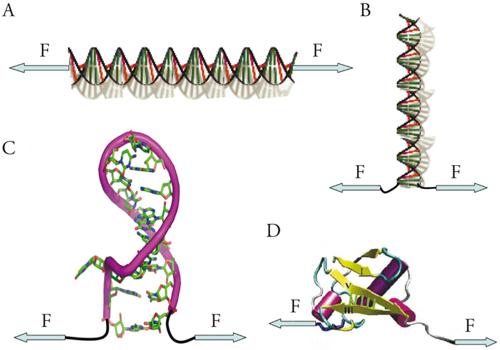
I blocchi base fondamentali della materia biologica sono gli acidi nucleici (come il DNA e l'RNA) e le proteine. Gli acidi nucleici hanno il compito di mantenere e regolare il flusso di informazione genetica durante i processi di duplicazione e di trascrizione. Tra le proteine si evidenziano, tra molti altri complessi molecolari, gli enzimi, catalizzatori delle reazioni chimiche all'interno della cellula. Sebbene la funzione primordiale degli acidi nucleici sia il mantenimento del genoma, nei primi anni Ottanta del Novecento si scoprì che l'RNA può presentare un'attività catalitica simile alle proteine. L'RNA, dunque, è una molecola con funzioni biologiche a cavallo tra il DNA e le proteine. Gli SME sono stati impiegati nello studio di una grande varietà di sistemi biomolecolari che spaziano dagli acidi nucleici alle proteine (fig. 4). Sono anche stati applicati alla ricerca su teorie del non-equilibrio in fisica statistica.
Acidi nucleici
La scoperta della struttura del DNA è stata il punto di partenza dello sviluppo della biologia molecolare. Il DNA è una doppia catena di polinucleotidi formati da zuccheri, generata dalla condensazione dell'acido fosforico con un gruppo idrossido del desossiribosio. Le due catene che configurano la molecola di DNA sono allineate in modo antiparallelo con legami a idrogeno tra basi complementari, formando le coppie di Watson-Crick (Guanina con Citosina e Adenina con Timina). Le proprietà elastiche della molecola di DNA sottoposta a una forza meccanica possono essere investigate in SME impiegando una qualsiasi delle tecniche descritte (fig. 4A). Negli esperimenti con MT è possibile applicare un momento sulla molecola di DNA e studiare le sue proprietà elastiche e di torsione simultaneamente. Uno dei risultati più importanti che emerge dallo studio della molecola di DNA è l'esistenza di un gran numero di transizioni strutturali in presenza di forza e/o di momento. Ne risulta un ricco e complesso diagramma di fasi.
I primi stiramenti di singole molecole di DNA sono stati effettuati da Laura Finzi, Steven Smith e Carlos Bustamante nel 1992 all'Istituto di Biologia Molecolare dell'Università dell'Oregon, quando visualizzarono molecole di DNA del batteriofago lambda usando AFM in presenza di forze magnetiche e idrodinamiche. Nel 1995 furono adoperate delle OT per allungare singole molecole di DNA. La risposta elastica della molecola può essere descritta adeguatamente con il modello di worm-like chain, noto anche in teoria dei polimeri come modello di Porod e Kratky, che descrive la deformazione di una catena di monomeri caratterizzata da una rigidità di curvatura. Le curve di forza-deformazione (CFE) dimostrano che il DNA ha una lunghezza di persistenza di circa 50 nm (la lunghezza di persistenza è legata alla rigidità della molecola e misura la distanza fino alla quale la molecola mantiene un determinato orientamento in presenza di fluttuazioni termiche). A 65 pN si osserva una transizione strutturale nella quale l'elica che conforma la molecola di DNA si srotola passando dalla normale struttura B a una struttura a scala nella quale le basi si inclinano, orientandosi in modo cooperativo lungo l'asse della forza. In questa struttura (denominata S in luogo di stretched) la lunghezza della molecola di DNA è 1,7 volte la sua lunghezza originale. La transizione di sovrastiramento tra le fasi B e S si mostra come un plateaux della forza nelle CFE, che ricorda le transizioni di primo ordine (fig. 3C). L'importanza biologica di questa transizione strutturale, così come di altre che si ottengono in presenza di avvolgimento/allungamento, è ancora sconosciuta.
In altri tipi di esperimenti è possibile staccare (unzipping) le due catene della molecola di DNA allungandole dalla stessa estremità della molecola (fig. 4B). In questi esperimenti i legami Watson-Crick, che mantengono unite le basi per i legami a idrogeno, si spezzano ed è possibile misurare le forze intramolecolari che mantengono stabile la struttura della molecola di DNA. Queste forze, dell'ordine dei 15 pN, permettono di staccare segmenti di doppia catena per alcune decine di paia di basi; lo schema di rottura dipende dalla specifica sequenza dei segmenti (specialmente se questi contengono prevalentemente legami di tipo GC o AT). Forse, in un futuro prossimo, questi esperimenti permetteranno di sequenziare le molecole di DNA per forze. Come il DNA, anche l'RNA forma una catena di polinucleotidi stabilizzata da legami a idrogeno tra basi nitrogenate. Tuttavia, esistono differenze strutturali di rilievo tra le due molecole: (a) l'RNA è composto da una catena semplice con la tendenza a formare strutture a forma di forcina (hairpin) mentre il DNA è composto da una doppia catena; (b) l'RNA contiene Uracile invece di Timina; (c) il ribosio dell'RNA contiene un gruppo OH altamente reattivo, capace di stabilizzare strutture ripiegate attraverso legami specifici con cationi bivalenti (per es., magnesio e calcio).
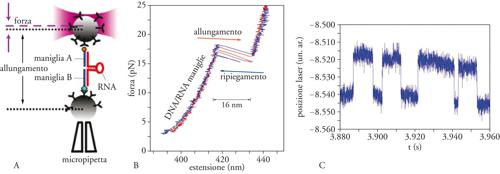
L'RNA forma una grande varietà di strutture secondarie (forcine, steli, anelli, biforcazioni, triplette, rigonfiamenti) e terziarie (biforcazioni, pseudonodi, kissing loops). La chimica dell'RNA è più semplice di quella delle proteine (4 nucleotidi nell'RNA contro i 20 amminoacidi delle proteine), il che lo rende un interessante modello per la comprensione della dinamica di ripiegamento delle biomolecole. Con gli SME, inoltre, è possibile studiare le forze che stabilizzano la struttura della molecola di RNA (fig. 3C). Lo schema sperimentale è simile a quello impiegato per allungare molecole di DNA, salvo per il fatto che le forcine che formano la molecola di RNA generalmente sono piccole e si estendono al massimo fino a varie decine di nanometri. Siccome le microsfere hanno un diametro nell'ordine dei micrometri e le forcine di RNA sono così piccole, bisogna sintetizzare un complesso molecolare che includa polimeri che apportino sufficiente spazio tra le due microsfere per poter stabilire la connessione molecolare. Ciò si ottiene unendo le molecole di RNA a maniglie formate da ibridi di DNA/RNA, che facilitano la manipolazione della molecola di RNA inserita al loro interno (fig. 5A). Nelle CFE ottenute durante l'allungamento delle molecole di RNA si osserva dapprima la risposta elastica delle maniglie e poi lo spiegamento della molecola di RNA (fig. 5B). Quest'ultimo è caratterizzato da un salto nella forza e da un allungamento del complesso molecolare.
La rottura dei legami che stabilizzano la struttura nativa della molecola di RNA avviene a valori di forze di circa 15 pN, simili ai valori della forza per i quali avviene il distacco della molecola di DNA. Durante il processo di rilassamento la molecola di RNA torna a ripiegarsi nella sua conformazione iniziale, mostrando un aumento di forza e una diminuzione dell'estensione molecolare. Il valore della forza, per la quale la forcina si ripiega/allunga, varia ogni volta, perché il processo di rottura è stocastico. Un'altra caratteristica importante della reazione in forza ripiegamento/allungamento è la presenza di isteresi: il valore medio della forza alla quale la molecola si allunga è maggiore del valore medio della forza alla quale la molecola si ripiega nella sua conformazione nativa. Queste misure permettono, usando il principio termodinamico ΔG=Wrev, di valutare l'energia libera di formazione, ΔG, per strutture di RNA, a partire dal lavoro meccanico richiesto per allungare la molecola durante un processo reversibile, Wrev. Il lavoro è dato dall'area delimitata dalla CFE, valutata tra il punto iniziale nel quale la molecola inizia ad allungarsi e quello finale quando la molecola è completamente allungata. A questo valore di lavoro bisogna sottrarre il contributo della deformazione elastica delle maniglie e quello dovuto all'allungamento della molecola di RNA, che, in assenza di forza, non rimane allungata bensì si ripiega come un gomitolo disordinato.
Inoltre, è possibile eseguire esperimenti di transizione, detti anche di salto, mantenendo fissa la posizione della micropipetta rispetto alla trappola (modalità passiva), oppure mantenendo costante il valore della forza attraverso un sistema di retroalimentazione (modalità a forza costante) fig. 5C. Si osserva, quindi, come la molecola salti dallo stato ripiegato a quello allungato per una determinata posizione della micropipetta o per un valore definito della forza applicata. In tutti e due i casi, i tempi di permanenza della molecola in ogni stato forniscono una misura diretta delle velocità di reazione a forza. Le teorie fenomenologiche (teorie di Kramers-Bell) descrivono la cinetica delle reazioni in sistemi con doppio stato in presenza di una forza meccanica. Queste teorie stabiliscono un rapporto tra le velocità di reazione e i parametri che caratterizzano le proprietà geometriche dell'andamento dell'energia libera lungo le coordinate di reazione, che in questo caso corrisponde all'estensione molecolare. Il comportamento di molte molecole di RNA è descritto bene dal modello del doppio stato, anche se in realtà il processo di ripiegamento è molto più complesso. Negli esperimenti di salto si riscontrano spesso stati intermedi di vita media rilevante e anche nei cicli di distensione si osserva la formazione di stati mal ripiegati. Tutto ciò suggerisce che il profilo di energia libera sottostante alla dinamica del ripiegamento delle molecole di RNA è instabile, caratterizzato da molti minimi locali. Le molecole, dunque, presentano una cinetica di ripiegamento più lenta di quella riscontrata nelle proteine.
Proteine
L'importanza delle proteine in biologia ha stimolato un gran numero di SME. Due sono i processi cruciali nei quali intervengono le proteine: (a) quelli che non richiedono attività biologica o consumo di ATP come fonte di energia chimica; (b) quelli che richiedono un consumo di ATP.
La molecola di ATP rappresenta la moneta di scambio energetico a livello cellulare. È composta da adenosina (Adenina+ribosio) e tre fosfati inorganici. L'ATP si idrolizza con una molecola d'acqua liberando un fosfato e producendo una quantità di energia equivalente a 7kBT. Nella prima categoria sono incluse le interazioni intramolecolari (per es., il ripiegamento delle proteine) e quelle intermolecolari (per es., le interazioni proteina-proteina, proteine-acidi nucleici o tra proteine e altre molecole). La seconda categoria di processi include i motori molecolari e gli enzimi. La maggior parte degli SME su proteine si è concentrata sullo studio delle interazioni intramolecolari delle proteine, delle interazioni intermolecolari di tipo ligando-recettore e, in particolar modo, sullo studio di motori molecolari.
Le interazioni proteina-proteina sono molto importanti per i processi di regolazione genica, trasmissione di segnali chimici all'interno della cellula e altri processi che si svolgono durante lo sviluppo embrionario. Lo studio delle interazioni donatore-accettore è realizzato con l'AFM. In questi esperimenti la punta dell'AFM e il substrato sono ricoperti con molecole di tipo donatore (ligando) o accettore, che possono interagire specificamente in base al meccanismo toppa-chiave. Le connessioni molecolari si stabiliscono con l'avvicinarsi della punta dell'AFM al substrato. Se si separa la punta dell'AFM dalla superficie, si ottiene la rottura del legame donatore-accettore, che ha luogo con la caduta brusca del valore della forza. Se l'esperimento è ripetuto diverse volte, si può ottenere la distribuzione delle forze di rottura. In generale questa distribuzione dipende dalla velocità di separazione della punta dal substrato. In particolare, la forza media di rottura aumenta in proporzione logaritmica rispetto alla velocità di separazione della punta.
Esperimenti simili sono stati eseguiti con l'AFM per studiare le interazioni intramolecolari attraverso l'allungamento meccanico delle proteine. Per esempio, la titina è una molecola dall'alto peso molecolare responsabile della risposta elastica delle fibre muscolari. La titina è composta da una sequenza ripetuta di diversi moduli proteici, come la immunoglobulina e la fibronectina. La distribuzione eterogenea di questi moduli ha portato alla sintesi di poliproteine attraverso l'applicazione di tecniche di ingegneria molecolare. Questi sono complessi molecolari contenenti un unico modulo proteico ripetuto lungo la poliproteina. Esperimenti eseguiti dai gruppi di Hermann E. Gaub, all'Università di Monaco di Baviera, e Julio M. Fernández, presso la Columbia University di New York, mostrano un segnale di forza a forma di dente di sega corrispondente all'allungamento dei diversi moduli (fig. 6). Il vantaggio delle poliproteine è che esse permettono di studiare nel dettaglio le proprietà meccanico-chimiche di un particolare modulo, evitando inoltre la presenza di interazioni non-specifiche tra substrato e modulo proteico. Quest'ultimo aspetto è particolarmente rilevante per lo studio del ripiegamento di proteine, un processo nel quale le proteine si ripiegano correttamente nella loro struttura nativa biologicamente attiva. Gli SME permettono di seguire in tempo reale il processo di ripiegamento della proteina, per esempio utilizzando la tecnica SMF o la FRET. Gli SME sono anche stati applicati allo studio della condensazione di acidi nucleici. Il DNA all'interno del nucleo cellulare è compattato in uno spazio di dimensioni tra le 10.000 e 100.000 volte inferiori alla lunghezza della molecola. Ciò è reso possibile grazie all'associazione del DNA con dei complessi proteici denominati istoni. Questi contengono gruppi di amminoacidi che si protonano in condizioni di pH fisiologiche (pH tra 6 e 7). Il DNA, invece, presenta un'alta carica elettrostatica negativa per via della ionizzazione delle catene dei fosfati. L'attrazione negativa tra gli istoni protonati e il DNA causa l'avvolgimento del DNA intorno agli istoni, formando una struttura chiamata nucleosoma, che rappresenta il livello più basso di compattazione del DNA. Alla fibra di nucleosomi (anche detta fibra da 10 nm) seguono altre strutture più complesse, nelle quali i nucleosomi si sovrappongono uno sull'altro. Con le OT è possibile studiare in tempo reale l'assemblaggio di una molecola di DNA e gli istoni. È anche possibile allungare le matrici di nucleosomi (nucleosomal array) e studiare il modo in cui i nucleosomi si distaccano dalla struttura man mano che la forza applicata aumenta. Altri esperimenti hanno studiato la condensazione di DNA con molecole polimeriche di dimensioni simili agli istoni. Questi sistemi sono più semplici di quello DNA-istone e forniscono modelli per la comprensione dell'aggregazione tra acidi nucleici e proteine.
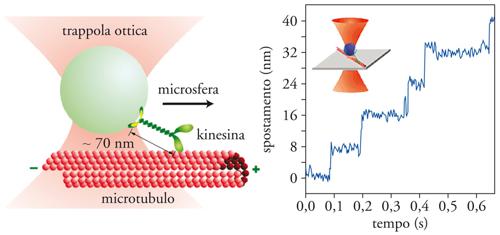
Infine, menzioniamo una delle tematiche più presenti tra gli SME: lo studio dei motori molecolari, cioè degli enzimi che si muovono lungo binari molecolari trasformando l'energia chimica ottenuta dall'idrolisi dell'ATP in lavoro meccanico utile. Per esempio, la kinesina è una molecola che si muove lungo microtubuli cellulari e trasporta le vescicole da una zona all'altra della cellula. A ogni ciclo meccanico-chimico la kinesina consuma una molecola di ATP e compie un passo lungo il microtubulo. I motori molecolari sono caratterizzati dalla processività, cioè dal numero di cicli compiuti prima di lasciare la traccia. Le processività variano in modo consistente, da 1 (come nel caso della kinesina) fino a vari milioni (per la DNA-polimerasi). Si ritiene che i motori molecolari funzionino come rettificatori browniani, utilizzando l'energia ottenuta nell'idrolisi dell'ATP in ADP per eseguire un movimento che viene poi rettificato attraverso il cambio conformazionale risultante dalla liberazione dell'ADP. I motori molecolari stupiscono per la loro enorme efficienza: la maggior parte dell'energia ottenuta dall'idrolisi di una molecola di ATP (circa 20 kBT) viene usata come lavoro utile. Per esempio, la kinesina adopera in modo efficiente circa il 60% dell'energia liberata nell'idrolisi dell'ATP, mentre nel caso del motore rotatorio F1-ATP, la bomba di protoni del mitocondrio, raggiunge un'efficienza del 90%. Si ignora in che modo i motori molecolari abbiano raggiunto tali gradi di efficienza nel corso della loro evoluzione.
Gli esperimenti sui motori molecolari si servono per lo più di OT. La procedura consiste nel legare il motore (per es., la kinesina) a una microsfera intrappolata nel potenziale ottico. Man mano che la kinesina avanza, esercita una trazione sulla microsfera e quindi una forza che si può misurare (fig. 7). È possibile mantenere una forza costante implementando un meccanismo di retroalimentazione della forza tale che mantenga la kinesina in costante movimento. Alternativamente, si può eseguire l'esperimento in modalità passi va, dove il motore avanza fintanto che la forza che agisce su di esso non è sufficientemente grande da fermarlo. Gli esperimenti realizzati da Steven M. Block e i suoi collaboratori all'Università di Stanford in California mostrano che la kinesina si muove a passi di 8 nm e a una velocità media di 2 μm al secondo. Esperimenti simili sono stati realizzati con gli enzimi che partecipano nella regolazione del genoma in processi come la duplicazione e la trascrizione. Le polimerasi di DNA e RNA si muovono lungo il DNA con il compito di trasmettere l'informazione genetica a una nuova generazione cellulare (replicazione), oppure di trascrivere il DNA in RNA-messaggero per la codifica dei geni (trascrizione) e la successiva sintesi di proteine nel ribosoma (traduzione). In questo caso la distanza tra basi consecutive lungo il DNA (3 Å) pone a dura prova i limiti strumentali delle OT. Lo sviluppo di strumenti ultrastabili ad alta risoluzione spaziale rappresenta dunque un passo importante per poter caratterizzare la cinetica associata al movimento di questi enzimi.
Menzioniamo anche lo studio dei motori incaricati di impacchettare il DNA di batteriofagi nel virione durante un ciclo di infezione virale. Un motore la cui struttura molecolare è ben nota è il batteriofago Φ29, studiato dall'équipe di Bustamante all'Università di Berkeley in California utilizzando OT: può generare forze fino a 60 pN, il che lo rende uno dei motori più potenti attualmente conosciuti. Infine, menzioniamo lo studio delle topoisomerasi e girasi, motori capaci di generare o rilassare la tensione torsionale del DNA e di grande importanza nella regolazione e nel mantenimento del genoma. Con le MT si possono introdurre o ridurre giri di elica della molecola di DNA e osservare l'azione diretta di questi enzimi nella topologia del DNA. Molti di questi esperimenti sono stati eseguiti dall'équipe di David Bensimon e Vincent Croquette del Laboratoire de Physique Statistique dell'École Normale Supérieure di Parigi.
Teorie del non-equilibrio in fisica statistica
Gli SME offrono la possibilità di utilizzare biomolecole come sistemi modello per lo studio di teorie di fisica statistica. La capacità degli SME di misurare energie dell'ordine di vari kBT permette di esaminare la condotta termodinamica di piccoli sistemi molto lontani dall'equilibrio. In effetti, la maggior parte dei processi biomolecolari si verifica in condizioni di grande densità molecolare (molecular crowding) ed è nettamente irreversibile. Lo studio dei processi termodinamici in piccoli sistemi riceve attenzione da anni, ma solo ora è finalmente possibile eseguire esperimenti ragionevolmente precisi sulle biomolecole. Negli ultimi anni è stata sviluppata un'importante attività teorica nei cosiddetti teoremi di fluttuazione. Questi teoremi forniscono relazioni che quantificano le probabilità di assorbimento dell'energia del bagno termico, paragonate con la facilità di dispersione della stessa quantità di energia verso il bagno termico, per sistemi termodinamici lontani dall'equilibrio. L'interesse di queste ricerche nella biofisica molecolare consiste nel fatto che la maggior parte delle reazioni molecolari si manifesta lontano dall'equilibrio, aprendo quindi la porta allo studio sull'importanza delle fluttuazioni estreme nel comportamento della materia biologica. Questo campo, attualmente in auge, lascia intravedere quesiti affascinanti, affrontabili in un futuro prossimo sia con un approccio teorico sia sperimentale.
Bibliografia
Bai 1999: Bai, Chunli e altri, Single molecule physics and chemistry, "Proceedings of the National Academy of Sciences", 96, 1999, pp. 11075-11076.
Bao 2002: Bao, Gang, Mechanics of biomolecules, "Journal of the mechanics and physics of solids", 50, 2002, pp. 2237-2274.
Bustamante 2000: Bustamante, Carlos - Macosko, Jed C. - Wuite, Gijs J.L., Grabbing the cat by the tail: manipulating molecules one by one, "Nature reviews. Molecular cell biology", 1, 2000, pp. 130-136.
Bustamante 2000: Bustamante, Carlos e altri, Single-molecule studies of DNA mechanics, "Current opinion in structural biology", 10, 2000, pp. 279-285.
Bustamante 2005: Bustamante, Carlos - Liphardt, Jan - Ritort, Felix, The nonequilibrium thermodynamics of smallsystems, "Physics today", 58, 2005, pp. 43-48.
Chatenay 2004: Multiple aspects of DNA and RNA: from biophysics to bioinformatics, edited by Didier Chatenay e altri, Berlin, Springer, 2004.
Cocco 2002: Cocco, Simona - Marko, John F. - Monasson, Rémi, Theoretical models for single-molecule DNA and RNA experiments: from elasticity to unzipping, "Comptes rendus physique", 3, 2002, pp. 569-584.
Fisher 1999: Fisher, Thomas e altri, The study of protein mechanics with the atomic force microscope, "Trends in biochemical sciences", 24, 1999, pp. 379-384.
Flyvbjerg 2002: Physics of biomolecules and cells, edited by Henrik Flyvbjerg e altri, Berlin, Springer, 2002.
Frontiers in chemistry: single molecules, "Science", 283, 1999, pp. 1593-1804.
Haran 2003: Haran, Gilad, Single-molecule fluorescence spectroscopy of biomolecular folding, "Journal of physics: condensed matter", 15, 2003, pp. R1291-R1317.
Ritort 2003: Ritort, Felix, Work fluctuations, transient violations of the second law and free-energy recovery methods, "Séminaire Poincaré", 2, 2003, pp. 193-226.
Strick 2001: Strick, Terence R. e altri, The manipulation of single biomolecules, "Physics today", 54, 2001, pp. 46-51.
Strick 2003: Strick, Terence R. e altri, Stretching of macromolecules and proteins, "Reports on progress in physics", 66, 2003, pp. 1-45.
Wang 2001: Wang, Kuan - Forbes, Jeffrey G. - Jin, Albert J., Single molecule measurements of titin elasticity, "Progress in biophysics and molecular biology", 77, 2001, pp. 1-44.