Citogenetica
Citogenetica
La citogenetica è una disciplina della biologia piuttosto giovane. È nata infatti con la riscoperta delle leggi di Mendel, avvenuta nel 1900, che fece constatare la stretta correlazione esistente fra queste leggi e il comportamento dei cromosomi nel corso dei fenomeni citologici legati alla divisione cellulare e al ciclo riproduttivo sessuale degli organismi eucarioti. La sua prima formulazione dottrinale è rappresentata dalla 'teoria cromosomica dell'eredità di Sutton-Boveri', esposta nel 1902-1903, con la quale i cromosomi sono identificati come la base fisica dell'eredità mendeliana e viene chiarito che, sebbene si rendano visibili solo durante la mitosi o la meiosi, essi sono comunque sempre presenti nel nucleo interfasico dove mantengono la loro individualità strutturale e la loro continuità genetica. Si riconosce anche che nelle cellule somatiche originate dallo zigote vi è un insieme di cromosomi ‒ il corredo o complemento cromosomico ‒ composto da coppie di cromosomi morfologicamente simili fra loro ‒ i cromosomi omologhi ‒, il cui numero totale è uguale al numero complessivo dei cromosomi presenti nei gameti dai quali lo zigote ha preso origine.
Nei gameti si ritrova un solo omologo della coppia e il loro insieme costituisce l'assetto cromosomico, l'entità che esprime l'intero e completo patrimonio ereditario della specie alla quale l'organismo appartiene e che per il numero, le dimensioni e la morfologia dei cromosomi dei quali si compone risulta anche essere un elemento caratteristico della specie. Il corredo cromosomico dei gameti, che consiste di un solo assetto cromosomico, è detto 'aploide' (n), mentre diploide (2n) è il corredo delle cellule originate dallo zigote che, con la fecondazione, ha visto l'unione del corredo del gamete maschile con quello del gamete femminile. Con questa teoria sono chiaramente delineati i fondamenti scientifici della citogenetica: lo studio dei cromosomi, intesi come supporto fisico dei fattori dell'eredità mendeliana, e quello dell'assetto cromosomico, ossia la modalità secondo la quale il materiale genetico della specie si organizza.
Il cammino della citogenetica è proseguito nel XX sec. da un lato avvalendosi del microscopio ottico come strumento d'indagine pressoché esclusivo e dall'altro mantenendo una impostazione dottrinale che valorizzava lo studio dei cromosomi piuttosto che il loro assetto complessivo. Nella trattatistica più diffusa, la citogenetica è definita come disciplina della biologia ‒ a cavallo fra citologia e genetica ‒ che studia l'ereditarietà in funzione delle strutture cellulari che hanno un diretto rapporto con i fenomeni dell'ereditarietà biologica. Essa viene distinta dalla 'cariologia', che studia le medesime strutture cellulari svincolate però dai loro rapporti con l'ereditarietà.
Con l'avvento delle tecnologie di biologia molecolare, dell'informatica e della nanotecnologia, che è stato inoltre accompagnato dal sequenziamento di interi genomi degli eucarioti, la citogenetica si è oggi liberata dal suo rapporto quasi esclusivo con la microscopia ottica e la citologia ed è divenuta una disciplina della genomica rivolta alla organizzazione morfo-strutturale dei genomi.
Mappe cromosomiche e cariotipo
A brevissima distanza di tempo dalla presentazione della teoria cromosomica dell'eredità, Thomas H. Morgan e i suoi studenti-collaboratori, utilizzando incroci di mutanti del comune moscerino della frutta (Drosophila melanogaster), dimostrarono che i geni, come sono oggi chiamati i fattori dell'eredità mendeliana, sono allocati sui cromosomi secondo una ordinata disposizione lineare e che ogni gene occupa sul cromosoma una propria precisa posizione, il locus genico. Dimostrarono anche che i geni posti sullo stesso cromosoma vengono ereditati insieme o in associazione e che ciò porta a identificare alcuni gruppi di associazione dei geni (o di linkage) il cui numero è identico a quello dei cromosomi che compongono l'assetto cromosomico della specie alla quale l'organismo appartiene. Mediante la valutazione dei tassi di ricombinazione relativi a geni che appartengono al medesimo gruppo di associazione, essi stimarono le distanze che separano un gene dall'altro e così, dopo aver elaborato graficamente le posizioni reciproche dei vari geni su un cromosoma con le corrispondenti distanze, riuscirono a disegnare 'mappe genetiche' dell'assetto cromosomico della drosofila.
La fortunata presenza di cromosomi politenici, un tipo di cromosomi dalle dimensioni enormemente grandi, nelle cellule delle ghiandole salivari di alcuni Ditteri, e in particolare della stessa D. melanogaster, ha successivamente consentito di reinterpretare in termini citologici i dati delle mappe genetiche. Su questi cromosomi era infatti possibile riconoscere un pattern trasversale di bande, positive alla reazione di Feulgen e dunque ricche in DNA, alternate a bande negative, che corrispondeva esattamente a quello proposto dalle mappe genetiche e che vedeva i geni mendeliani posizionarsi esattamente a livello delle bande Feulgen-positive. Le mappe cromosomiche citologiche e genetiche ottenute nella drosofila offrono una prima significativa evidenza dell'integrazione strutturale nell'organizzazione dell'assetto cromosomico, dagli alti livelli citologici rappresentati dai cromosomi, sino a quelli più bassi inerenti la posizione dei locus genici lungo i cromosomi.
Le favorevoli condizioni sperimentali offerte dalla drosofila non si ritrovarono nella generalità degli eucarioti; si dovettero perciò attendere diversi anni per confermare risultati di così grande potenza e significatività per il corpus dottrinale della citogenetica. Per tutta la prima metà del XX sec. gli studi citogenetici si rivelarono piuttosto complessi e di difficile interpretazione, come dimostra il fatto che per conoscere l'esatto numero dei cromosomi del corredo diploide della nostra specie si dovette aspettare il 1956. Questa situazione si modificò con il progresso delle tecniche di coltura in vitro delle cellule, il trattamento delle colture con colchicina e soprattutto con l'esposizione delle metafasi a una soluzione ipotonica prima della loro fissazione. Importante è stata anche una nuova fissazione dei preparati metafasici e la microfotografia per l'analisi delle relative piastre cromosomiche. Queste innovazioni metodologiche si sono tradotte in una procedura tecnica, l'analisi citogenetica, facilmente applicabile per lo studio citologico microscopico del complemento cromosomico degli eucarioti.
Questo studio, esteso alla generalità delle piante e degli animali, ha portato alla definizione del cariotipo, ovvero degli aspetti citologico-microscopici specie-specifici del complemento cromosomico di popolazioni cellulari, clonalmente fra loro collegate e riferibili a un individuo della specie. Lo studio del cariotipo in questi organismi ha confermato l'universalità della costanza dell'organizzazione del materiale genetico secondo assetti cromosomici specie-specifici, ed evidenziato come questi assetti siano invece variati enormemente durante l'evoluzione, con modalità assai differenti a seconda dei gruppi di eucarioti. Il cariotipo può consistere in un numero talora bassissimo di cromosomi, come le 2 coppie dello scorpione, talora molto elevato, fino a numeri superiori ai 220 cromosomi (tab. 1). Questi studi hanno contemporaneamente rilevato che, mentre in alcuni gruppi tassonomici il cariotipo si è mantenuto sostanzialmente inalterato attraverso lunghi stadi dell'evoluzione, in altri gruppi esso ha invece subito marcati cambiamenti, anche in specie relativamente vicine. Queste osservazioni hanno fatto concludere che l'assetto cromosomico, che esprime il complesso genetico della specie, ha assunto la configurazione morfo-strutturale del cariotipo che è variata nel corso dell'evoluzione al pari di qualsiasi altra caratteristica del fenotipo.
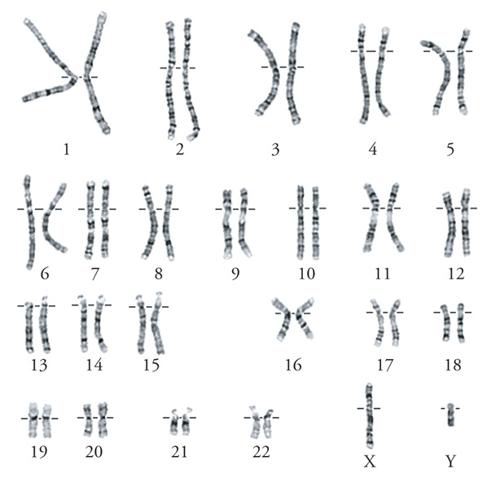
Per le specie animali o vegetali con sessi separati, lo studio del cariotipo permette di definire, in modo indipendente dal fenotipo, il genere sessuale di individui, ma anche di tessuti, linee cellulari o singole cellule. In queste specie, infatti, una delle coppie del complemento cromosomico diploide non manifesta la consueta omologia morfologica in entrambi i sessi, ma in uno solo dei due. I cromosomi di questa coppia sono detti 'eterocromosomi' o 'cromosomi sessuali', gli altri sono detti autosomi. Il cromosoma sessuale che mantiene l'omologia è chiamato X se il sesso dell'individuo è femminile, Z se il sesso è maschile. Per il cromosoma sessuale che non mantiene l'omologia si parla invece di cromosoma Y se il sesso dell'individuo è maschile, di W se è femminile. Il cariotipo delle cellule somatiche della specie umana consiste, com'è noto, di 46 cromosomi con 22 coppie di autosomi e una coppia di cromosomi sessuali, XX nella femmina e XY nel maschio.
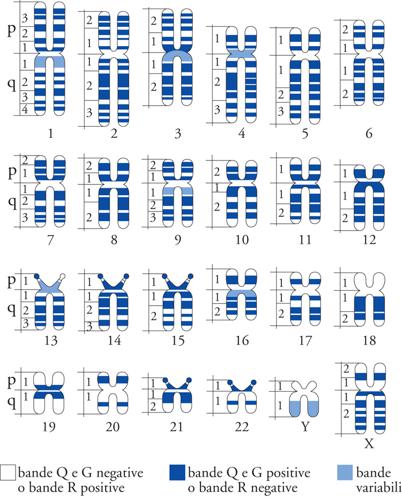
Agli inizi degli anni Settanta, l'introduzione delle cosiddette 'tecniche di bandeggiamento cromosomico' consentì di inserire fra le caratteristiche specie-specifiche del cariotipo sino ad allora considerate, quali la costanza del numero, delle dimensioni e della morfologia dei cromosomi, anche quelle relative alla struttura/composizione dei cromosomi. Al primo metodo, detto bandeggiamento Q (in quanto le metafasi erano colorate con mostarda di quinacrina), si aggiunsero diversi altri tipi di bandeggiamento, come le bande G, o le bande R, ecc., i quali, a seguito di determinati trattamenti dei preparati e colorazioni dei cromosomi metafasici, evidenziano un pattern trasversale di bande positive alternate a bande negative in una sequenza caratteristica e specifica di ciascuna coppia di omologhi, che permette non solo l'identificazione di tutti i cromosomi di un cariotipo, ma anche la caratterizzazione di ogni loro tratto. Sulla base della sequenza delle bande è inoltre possibile una rappresentazione dei cromosomi del cariotipo di una data specie con un ideogramma che, quale codice a bande, consente il suo studio con mezzi informatici (fig. 2). Basandosi esclusivamente sul contenuto in DNA dei singoli cromosomi, con la citometria a flusso è possibile ottenere il 'cariotipo a flusso', con il quale l'assetto cromosomico viene caratterizzato su base biochimico-quantitativa. Il cariotipo in questo caso non è più legato a un approccio morfo-strutturale di microscopia ottica, bensì alla composizione e distribuzione del DNA fra i cromosomi.
Il grado di risoluzione spaziale dell'analisi citogenetica, dato dalla sequenza delle bande, varia secondo il grado di allungamento dei cromosomi, le caratteristiche genetiche e il contenuto in DNA del loro genoma. Nel cariotipo umano, per esempio, sui compatti cromosomici metafasici si contano circa 350 bande per genoma aploide, mentre esse giungono fino a 1000 nei cromosomi prometafasici (fig. 3). Poiché il genoma umano consiste di circa 3 miliardi di paia di basi (pb), si può concludere che la risoluzione massima delle analisi citogenetiche con questi procedimenti citologici è dell'ordine di 3 milioni pb in termini di basi e di 30/50 in termini di geni, posto il loro numero totale intorno a 30.000/50.000. Le mappe citologiche ottenute con i metodi di bandeggiamento hanno quindi una risoluzione genetica ancora lontana da quella delle mappe citologiche dei cromosomi politenici. La microscopia elettronica non ha evidenziato nei cromosomi metafasici una specifica organizzazione subcromosomica che potesse essere correlata al bandeggiamento trasversale dei cromosomi. Questa organizzazione è invece evidente nei cromosomi meiotici pachitenici e in quelli a spazzola degli ovociti degli anfibi. Si prospetta così che tutti gli eucarioti possiedano, come dimostrato nella drosofila, una organizzazione morfo-strutturale specie-specifica del materiale genetico, che si estende dagli alti livelli citologici microscopici a quelli inferiori submicroscopici e sovramolecolari.
Variazioni del cariotipo
All'interno delle popolazioni di una data specie si possono trovare individui nei quali il cariotipo presenta alcune variazioni rispetto a quello tipico della specie di appartenenza. Le variazioni permanenti del cariotipo, capaci o meno di esercitare un'influenza sul fenotipo, sono dette 'anomalie cromosomiche'. Esse possono essere di tipo numerico, qualora si associno a variazioni di numero dei cromosomi del cariotipo, oppure di tipo strutturale, quando non comportano variazioni nel numero ma solo nella struttura di uno o più cromosomi. Sono anomalie cromosomiche numeriche le aploidie, quando il complemento cromosomico si compone di un solo assetto cromosomico, le poliploidie, quando il complemento cromosomico si compone invece di tre o più assetti cromosomici, e le aneuploidie, quando nel corredo diploide vi sono cromosomi in eccesso o in difetto rispetto al numero diploide caratteristico della specie. Sono invece anomalie cromosomiche strutturali: le delezioni, nelle quali si ha la perdita nel cariotipo di un segmento di cromosoma; le duplicazioni, quando il segmento di cromosoma è invece duplicato; le traslocazioni, quando si ha scambio di segmenti fra due o più cromosomi; le inserzioni, quando un segmento di cromosoma si inserisce all'interno di un cromosoma senza scambio reciproco di materiale.
Le anomalie del cariotipo, oltre che trovarsi in individui all'interno della popolazione di una data specie e risultare quindi intrapecifiche, possono interessare solo una parte dei tessuti o delle cellule di uno stesso individuo. Sono queste le anomalie cromosomiche intraindividuali, che si verificano dopo il concepimento in cellule ancora in grado di dividersi e che, conseguentemente, trasmettono clonalmente l'anomalia a tutte le cellule che derivano da esse per mitosi. Se l'evento è precoce nello sviluppo embrionale, l'individuo è 'a mosaico', possiede cioè organi e tessuti nei quali sono presenti da due a più popolazioni di cellule con differente cariotipo, una delle quali a cariotipo normale. Se l'evento avviene invece a sviluppo embrionale avanzato, e cioè dopo che si è completata l'organogenesi, l'anomalia cromosomica è confinata a un unico organo, tessuto o popolazione cellulare. Sono anomalie germinali quelle presenti nelle cellule della linea germinale e che si ritrovano quindi nei gameti (spermatozoi o cellula uovo); non hanno effetto sul fenotipo, possono essere trasmesse alla prole e interferire con la riproduzione.
Sono anomalie somatiche quelle che interessano organi, tessuti o popolazioni cellulari del corpo ma non cellule della linea germinale. Queste anomalie, pur potendo avere effetto funzionale sul fenotipo, non vengono trasmesse alla discendenza. Sono in particolare anomalie somatiche 'acquisite' quelle che si verificano nel corso del normale ricambio cellulare in un solo tessuto, come per esempio quelle che avvengono nel corso della carcinogenesi. Le anomalie somatiche possono essere letali, quando l'organismo che ne è colpito muore prima di aver raggiunto l'età riproduttiva o, raggiuntala, non si riproduce; subletali, patogene o neutre a seconda del coinvolgimento che hanno sull'individuo. Lo studio del cariotipo ha così una vasta ricaduta applicativa nei vari ambiti della biologia e della medicina.
Nell'uomo il riscontro di anomalie cromosomiche quali causa di malattia ha aperto un nuovo campo della medicina umana, quello della citogenetica clinica. Essa ha avuto inizio nel 1958, con la dimostrazione che la trisomia 21 è la causa della sindrome di Down. L'anno seguente è stata la volta della trisomia 18, causa della sindrome di Edwards, della trisomia 13, causa della sindrome di Patau, della sindrome di Klinefelter con un cromosoma X in eccesso in un soggetto di sesso maschile, della sindrome di Turner con un unico cromosoma X in un soggetto di sesso femminile. Le anomalie cromosomiche sono presenti nello 0,5% dei neonati, sono una delle principali cause di morte prenatale o di patologie congenite-malformative e sono all'origine di un numero notevole di condizioni cliniche con differente quadro clinico e gravità. Più del 50% degli aborti spontanei del primo trimestre di gravidanza è associato a un alterato numero o struttura dei cromosomi.
L'analisi citogenetica rappresenta oggi un importante test genetico. Nella medicina postnatale riguarda soggetti con sospetta sindrome cromosomica, genitori e familiari di soggetti con anomalie cromosomiche, riscontro di ritardo mentale e/o difetti congeniti, ritardo nella crescita, neonati nati morti, genitori di soggetti malformati o con sospetta sindrome cromosomica deceduti senza diagnosi, coppie con aborti spontanei ripetuti, infertilità maschile, femmine con assenza o interruzione del ciclo mestruale, fecondazione assistita, sindromi mendeliane da geni contigui o da instabilità cromosomica. Nella medicina prenatale l'analisi citogenica si effettua (nel corso del primo trimestre mediante villocentesi oppure mediante amniocentesi durante il secondo trimestre) nelle gravidanze a elevato rischio di anomalie cromosomiche del feto: età materna uguale o maggiore di 35 anni, figlio affetto da errore di numero dei cromosomi, anomalie del feto evidenziate in ecografia, indicazioni derivanti da test biochimici (altri test ecc.), aborti spontanei ripetuti. In oncologia lo studio citogenetico è utile per la diagnosi, la prognosi e la scelta della terapia più adeguata e ha lo scopo di identificare anomalie cromosomiche specifiche dei vari tipi istologici di tumori solidi o ematologici.
Cariotipo a colori e cariotipo ancestrale
Il complesso degli avanzamenti che coinvolgono la biologia moderna, in particolare quelli correlati alle tecnologie del DNA ricombinante, hanno interessato anche la citogenetica, facendola entrare nell'era molecolare. A dare inizio a questa fase è stata la FISH (Fluorescence in situ hybridization), una tecnologia basata sulla ibridazione in situ dei preparati citologici con sonde fluorescenti di DNA che possono consistere in genomi completi, interi cromosomi, parti di essi o singoli loro tratti, fino a qualsivoglia segmento genomico (inserito in vettori quali cosmidi, PACS, BACS e YACS) o sequenze di DNA, ripetute o singole. Con questa tecnica l'analisi citogenetica è rivoluzionata, in quanto viene svincolata dal suo rapporto, quasi esclusivo, con la morfo-struttura del cariotipo, per rapportarsi anche con le sequenze del DNA che sono alla sua base. Con la FISH non è nemmeno indispensabile, come per le tecniche precedenti, disporre di un preparato cromosomico: essa può essere infatti applicata a nuclei interfasici, sezioni di tessuto e a singole cellule. Con la Fiber-FISH possono anche essere utilizzate fibre distese di cromatina.
La risoluzione del mappaggio fisico su cromosomi metafasici altamente condensati è dell'ordine di 5Mbp. Un aumento della risoluzione fino a 100 kbp si può ottenere utilizzando nuclei interfasici e infine un ulteriore progresso in termini di risoluzione (5÷500 kbp) si ha con la Fiber-FISH. L'approccio morfologico-strutturale di studio dell'assetto cromosomico, che si era arrestato a una risoluzione di diverse megabasi, giunge con la FISH sino alla mappatura fisica dei geni e quindi alla stessa integrazione delle mappe citologiche e genetiche che si erano ottenute sulla drosofila. Sostenute da innovazioni tecnologiche di biologia molecolare, ottiche, informatiche e microelettroniche, a cavallo del passaggio del secondo millennio si è avuta una vera esplosione di successive implementazioni della tecnica di ibridazione in situ con sonde fluorescenti di DNA. Con le tecniche del painting cromosomico multicolore, come la M-FISH (Multiplex-FISH) o la SKY (Spectral karyotyping) con la MCB (Multicolor chromosome banding), che impiegano, come sonde, genoteche genomiche o cromosomiche marcate con diversi fluorocromi o con una combinazione degli stessi, si ottiene la 'cariotipizzazione a colori', cioè un'analisi del cariotipo mediante simultaneo e specifico riconoscimento in differente colore di ogni singolo cromosoma del cariotipo o di parte di esso con risoluzioni non inferiori a 2 Mbp. Lo sviluppo delle procedure per l'ibridazione di sonde multiple, ciascuna segnata con un colore diverso, per marcare metafasi disseminate e nuclei interfasici, ha avuto uno straordinario effetto sull'applicazione della citogenetica nella ricerca clinica e di base. Sono ora disponibili approcci multicolore che permettono di analizzare persino complessi riarrangiamenti cromosomici, come frequentemente si osservano nei tumori solidi. Essi possono essere applicati sia a metafasi disseminate sia a nuclei interfasici.
Mappe genomiche fisiche e cariotipo digitale
Un ulteriore decisivo sviluppo della cariotipizzazione a colori si è avuto con la ibridazione genomica comparativa (CGH, Comparative genomic hybridization). La CGH si basa su una ibridazione in situ che sfrutta la competizione tra due campioni di DNA genomico marcati diversamente e ibridati su un vetrino con metafasi normali appartenenti a un determinato cariotipo per identificare regioni, tratti o singole bande del cariotipo, che nei due genomi in comparazione risultano diverse per sequenza o quantità. La CGH costituisce la chiave per l'analisi comparativa degli assetti cromosomici più diversi con una risoluzione fino a 3 Mbp. Si può anche procedere con l'analisi citogenetica disponendo del solo DNA in quantità minimali, puro o anche con inquinamento batterico. Di queste tecniche si avvale oggi la citogenetica per analizzare le variazioni interindividuali del cariotipo e soprattutto quelle intraindividuali che si hanno nei tumori, o per impostare una citogenetica intraspecifica evoluzionistica in grado di verificare la conservazione o le variazioni strutturali in cariotipi di specie collocate in una linea evolutiva e, addirittura, riconoscere la collocazione di tratti di cromosomi umani in varie specie di Mammiferi, non solo in scimmie e proscimmie (cosa ovvia per l'indiscussa parentela) ma anche in specie evolutivamente lontane. La ricerca del cariotipo ancestrale per le varie classi dei viventi fino a giungere a quello originario degli eucarioti rappresenta uno degli obiettivi della citogenetica attuale.
Il completo sequenziamento di genomi di numerosi eucarioti, compreso quello umano del quale conosciamo oggi la sequenza, accurata al 99,99%, per il 99% della sua estensione, ha dato alla citogenetica l'ultima definitiva spinta perché essa abbandonasse la microscopia ottica come esclusivo campo d'indagine e per l'apertura dei suoi orizzonti tematici e metodologici agli altri campi dell'ereditarietà biologica. La citogenetica si è inserita a pieno titolo nella genomica, la nuova disciplina della biologia che si occupa dello studio del genoma, il sistema informazionale degli esseri viventi, nella sua struttura e contenuto genetico, funzione e funzionamento, nonché nella sua dinamica evolutiva. Il sequenziamento di grandi genomi ha imposto di suddividere il materiale genetico in diversi frammenti, che sono stati poi ricollocati secondo l'ordine e la posizione che essi occupano nei cromosomi interi. Questi frammenti possono essere utilizzati quali sonde e sono oggi prontamente disponibili per la maggior parte delle regioni genomiche. Le sonde possono essere selezionate facilmente utilizzando le banche dati quali Ensembl Cytoview, NCBI Map-Viewer o UCSC genome browser. Anche le sonde per il painting cromosomico, che colorano l'intero cromosoma o regioni cromosomiche, sono ampiamente disponibili, soprattutto grazie allo sviluppo degli approcci universali di PCR (Polymerase chain reaction) che consentono l'amplificazione di grandi regioni di DNA.
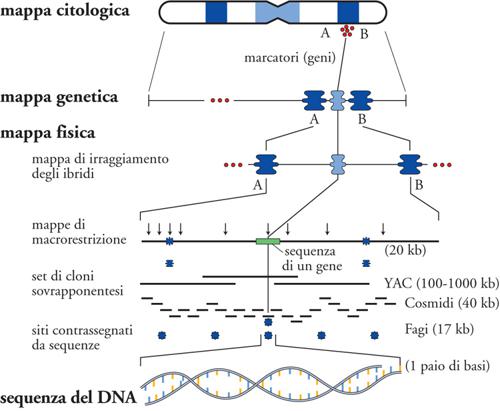
L'insieme dei tratti contigui di frammenti di DNA di un intero genoma costituisce la sua mappa fisica. Una mappa genomica fisica del genoma è l'allineamento dei tratti di DNA del genoma, che vede la distanza delle sequenze usate come marcatori espressa in chilobasi (kb). La mappatura fisica organizzata con contigui che coprano estese regioni cromosomiche viene ottenuta per un numero esponenzialmente crescente di genomi; la stessa sequenza completa di un dato genoma altro non è che la sua mappa fisica risolta alla singola base (Fig. 5). Per un numero esponenzialmente crescente di genomi è oggi possibile una integrazione fra mappe genetiche, citologiche e fisiche che fanno corrispondere quello che è visibile microscopicamente con le sue basi molecolari. La conoscenza di sequenze specifiche di un dato genoma consente di costruire frammenti di DNA a esso complementari e di utilizzare poi questi frammenti come sonde per ibridare in situ la corrispondente sequenza su di un preparato metafasico. È la citogenetica inversa che diviene una risorsa sempre più importante per la citogenetica e che è alla base del suo ultimo rivoluzionario approccio, l'array-CGH. Si tratta di una tecnologia più recente, che consiste nell'immobilizzare su un vetrino frammenti di diversa estensione di genoma, dai BAC/PAC fino a singoli nucleotidi, e di applicare su di essi la medesima strategia dell'ibridazione genomica comparativa. L'array-CGH permette di analizzare nell'intero genoma la perdita o l'acquisizione di materiale cromosomico a una risoluzione a livello del singolo nucleotide e di giungere conseguentemente al cosiddetto 'cariotipo digitale', in quanto tutto il processo di lettura è informatizzato.
La citogenetica del terzo millennio
Nella citogenetica attuale si combinano metodiche e tecnologie che le permettono di spaziare dallo studio di interi genomi fino all'identificazione di differenze a livello di singoli nucleotidi. Non essendovi più distinzione tra analisi citologica e analisi molecolare, è cambiata la natura della citogenetica, disciplina inizialmente a cavallo fra citologia e genetica, in quanto basata sulla microscopia ottica come suo esclusivo campo d'indagine. Un profondo rinnovamento della impostazione dottrinale della citogenetica tuttavia si attua oggi nei fatti dalla rivoluzione scientifica, che coinvolge il mondo delle scienze della vita e che ha posto il genoma a fondamento dei processi dell'ereditarietà biologica. Non è un caso che sia ritornato in auge un termine, quello di genoma, che era stato proposto negli anni Venti proprio per indicare l'assetto cromosomico in quanto aspetto citologico della totalità del patrimonio genetico. Per oltre mezzo secolo questo termine aveva avuto scarsa applicazione; l'impostazione scientifica dominante nella citogenetica del XX sec. riconosceva infatti ai cromosomi il rapporto più diretto con i caratteri ereditari e valorizzava pertanto queste strutture rispetto all'assetto cromosomico.
Genoma e assetto cromosomico attengono entrambi al medesimo soggetto scientifico, il patrimonio genetico della specie nella sua totalità e nelle sue caratteristiche specie-specifiche. Genoma e assetto cromosomico non sono però sinonimi. Mentre il genoma è il sistema informazionale proprio degli esseri viventi in tutte le sue caratteristiche, espressioni ed evoluzioni, l'assetto cromosomico rappresenta invece solo l'aspetto organizzativo morfo-strutturale specie-specifico del genoma. Questo, come tutti i sistemi informazionali stabili, necessita infatti di un supporto materiale per mantenere nel tempo il programma genetico scritto, e tale supporto, nella quasi totalità dei viventi, è costituito ancora dallo stesso DNA. L'assetto cromosomico è da intendersi quindi come la modalità specie-specifica secondo la quale il genoma si organizza negli alti livelli di complessità strutturale (citologici microscopici e submicroscopici e sovramolecolari), conseguenti al suo passaggio evolutivo dai procarioti agli eucarioti. In questo sta la coerenza semantica fra i due termini e questo giustifica anche il comune uso di genoma aploide in riferimento al genoma/assetto cromosomico.
È questa dunque la citogenetica del terzo millennio, un confluire e confondersi con tutte le differenti scienze interessate all'ereditarietà biologica, per assumere poi una sua più corretta posizione dottrinale. Non più disciplina della biologia, a cavallo fra citologia e genetica, che studia l'ereditarietà in funzione delle strutture cellulari che hanno un diretto rapporto con i fenomeni dell'ereditarietà biologica, bensì disciplina della genomica relativa all'organizzazione morfo-strutturale del genoma nelle situazioni di normalità, nella sua variabilità intraindividuale e interindividuale e nella sua dinamica evolutiva. Con questa veste si ricollega agli altri campi interessati all'ereditarietà biologica ed è coinvolta nelle ricadute applicative della genomica nei diversi ambiti delle scienze della vita, e in particolare in quello della medicina dell'uomo, come pure quelli finalizzati al miglioramento animale e vegetale.
Bibliografia
Barigozzi, Ghidoni 1976: Barigozzi, Claudio - Ghidoni, Achille, Cromosomi e meccanismi ereditari, Torino, UTET, 1976.
Garagna 2004: Mouse genetics after the mouse genome, edited by Silvia Garagna, Basel, Karger, 2004.
Gibson, Muse 2004: Gibson, Greg - Muse, Spencer V., Introduzione alla genomica, Bologna, Zanichelli, 2004 (ed. orig.: Primer in genome sciences, Sunderland, Sinauer, 2001).
Miller, Therman 2001: Miller, Orlando J. - Therman, Eeva, Human chromosomes, 4. ed., New York-London, Springer, 2001.
Schulz-Schaeffer 1980: Schulz-Schaeffer, Jurgen, Cytogenetics: plants, animals, humans, New York, Springer, 1980.
Speicher, Carter 2005: Speicher, Michael R. - Carter, Nigel P., The new cytogenetics: blurring the boundaries with molecular biology, "Nature reviews. Genetics", 6, 2005, pp. 782-792.
Swanson 1973: Swanson, Carl - Merz, Timothy - Young, William J., Citogenetica, Bologna, Zanichelli, 1973 (ed. orig.: Cytogenetics, New York, Prentice-Hall, 1967).
Sybenga 1972: Sybenga, Jacob, General cytogenetics, Amsterdam-London, North-Holland, 1972.
White 1973: White, Michael J.D., Animal cytology and evolution, 3. ed., Cambridge, Cambridge University Press, 1973 (ed. orig.: 1945).