Plasmidi
Plasmidi
I p. sono molecole di DNA di dimensioni ridotte, capaci di esistere come elementi genetici autonomi all'interno delle cellule. Essi sono molto diffusi fra i microrganismi, trovandosi in gran parte dei batteri (Procarioti), ma alcuni p. sono stati individuati in cellule più complesse come quelle dei lieviti (Eucarioti unicellulari). Il loro DNA è generalmente a doppia elica circolare, ma vi sono anche alcuni p. lineari, come quelli di batteri appartenenti ai generi Streptomyces e Borrelia. In maggioranza i p. sono circa 20 volte più piccoli dei cromosomi, ma è stata riscontrata grande diversità nelle loro dimensioni: da un migliaio a circa un milione di coppie di nucleotidi. Si conoscono migliaia di p. diversi: nel solo enterobatterio Escherichia coli ne sono stati scoperti diverse centinaia.
I p. sono capaci di replicarsi all'interno della cellula microbica in maniera indipendente rispetto al processo di replicazione del cromosoma; tuttavia il numero di copie di ogni p. all'interno di una cellula è costante. Alcuni di essi sono presenti in unica o poche copie (per es., il fattore di fertilità di Escherichia coli, di cui si parlerà diffusamente più avanti, è presente in due copie per cellula), altri possono raggiungere anche le 100 copie. Ciò è dovuto all'esistenza nella cellula di precisi meccanismi che impediscono, attraverso l'intervento di molecole regolatrici, sia l'eccessiva frequenza di replicazione, che porterebbe a un aumento del numero di copie, sia una più bassa velocità di replicazione, che porterebbe a una riduzione del numero di copie o persino alla comparsa nella popolazione di cellule prive di plasmide. Nel p. di Escherichia coli denominato ColE1, per es., il mantenimento del numero di copie avviene grazie a una proteina definita Rom, codificata dal p. stesso, che controlla la disponibilità di un piccolo RNA responsabile dell'inizio della replicazione del plasmide. All'interno di una stessa cellula possono coesistere p. di natura diversa, purché appartenenti a diversi 'gruppi di incompatibilità'. Un gruppo di incompatibilità (generalmente indicato con Inc) è costituito da p. incompatibili fra loro in quanto incapaci di esistere e quindi replicarsi all'interno della stessa cellula. Se con trasferimento genico (v. oltre) un p. entra in una cellula in cui ne è già presente un altro dello stesso gruppo Inc, i due p., per divisione cellulare, segregheranno in cellule figlie diverse. Ciò è reso possibile dallo stesso sistema di controllo del numero di copie citato sopra. Il repressore che regola il numero di copie 'percepisce' il p. estraneo dello stesso gruppo Inc come una copia aggiuntiva del p. endogeno e ne impedisce la replicazione.
Tutti i p. contengono i geni necessari per la loro replicazione e per il controllo del numero di copie e dell'incompatibilità con p. appartenenti allo stesso gruppo Inc. Alcuni posseggono anche geni che codificano per funzioni relative alla loro capacità di trasferirsi da una cellula a un'altra (v. oltre). Altri contengono geni che codificano per proteine che conferiscono alle cellule funzioni particolari, quali la patogenicità verso organismi superiori o verso altri microrganismi, oppure la capacità di detossificare metalli, degradare sostanze aromatiche complesse o conferire alla cellula resistenza a particolari sostanze tossiche. Tuttavia nella generalità dei casi i p. non esprimono funzioni indispensabili alla vita delle cellule, quali quelle legate alla replicazione del DNA, alla trascrizione dei geni, alla sintesi delle proteine o alle varie funzioni metaboliche che consentono alle cellule di accrescersi e duplicarsi. Infatti normalmente è possibile isolare cellule prive del loro p.; ciò avviene, anche se raramente, in natura, e può essere realizzato con più alta frequenza in laboratorio, incubando le cellule in presenza di inibitori della replicazione plasmidica quale l'acridina; queste cellule conservano la capacità di svilupparsi normalmente. Alcuni p. vengono definiti criptici in quanto non conferiscono alle cellule nessuna delle proprietà elencate sopra; è tuttavia possibile che una loro più accurata analisi porti all'identificazione di funzioni finora ignote e, in tal caso, essi non potranno più essere definiti tali.
Plasmidi coniugativi
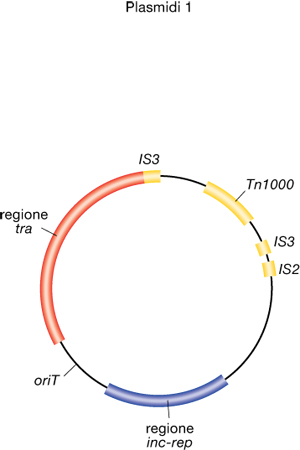
Alcuni p. vengono definiti coniugativi, in quanto possono essere trasmessi ad altre cellule mediante coniugazione, fenomeno in cui avviene un accoppiamento fra una cellula dotata di un certo p. coniugativo e un'altra cellula, compatibile con la precedente, nella quale non è presente (normalmente, ma non sempre, cellule della stessa specie microbica). Un esempio noto di p. coniugativo è il fattore di fertilità (fattore F) di Escherichia coli, costituito da circa 100.000 coppie di nucleotidi (fig. 1). Ceppi contenenti tale fattore (ceppi F+) vengono definiti maschi, ceppi che non lo contengono ma possono riceverlo per coniugazione (ceppi F−) vengono definiti femmine. Tali termini vanno riferiti esclusivamente al fenomeno di trasferimento genico, trattandosi di una sessualità tipica dei batteri non assimilabile alla più complessa sessualità propria degli Eucarioti. I ceppi F+ contengono sulla superficie cellulare due o tre copie di un'appendice, detta pilus sessuale o F-pilus, assente nei ceppi F− e responsabile dell'attrazione fra la cellula donatrice e la cellula ricevente. Si ritiene che il pilus attiri la cellula F− legandosi attraverso la propria estremità distale a un recettore che risulta presente sulla sua superficie e che, contraendosi, induca l'avvicinamento fra le due cellule che prelude all'accoppiamento. A dimostrazione del ruolo di tale appendice nella coniugazione, cellule che l'hanno persa in conseguenza, per es., di vigorosa agitazione di una provetta in cui è sospesa una coltura di un ceppo F+, non sono più in grado di trasferire il fattore di fertilità. Tali cellule, dotate di fattore di fertilità tuttavia incapaci di trasferirlo, sono definite fenocopie. L'accoppiamento tra due cellule inizia con una vera e propria fusione, con formazione di un ponte coniugativo attraverso il quale si pensa scorra il DNA plasmidico dal donatore al ricevente. Il trasferimento avviene mediante un meccanismo detto del cerchio rotante. Una proteina (TraI) crea un taglio su uno dei due filamenti del DNA plasmidico a livello del sito oriT interno al p., questi comincia a ruotare e il filamento lineare formatosi viene trasferito alla cellula F−. Mano a mano che il p. ruota, nella cellula donatrice avviene la sintesi del filamento che viene trasferito e nella cellula ricevente quella del filamento complementare a esso, in modo che alla fine del processo ambedue le cellule avranno una copia intera e circolare del p., costituita in entrambi i casi da un filamento preesistente e uno neoformato. Ne consegue che in seguito a un processo coniugativo la cellula maschio rimane tale, mentre la cellula femmina diventa maschio.
Sono stati descritti, anche se con minori dettagli, altri p. coniugativi, alcuni dei quali (come quelli appartenenti al gruppo di incompatibilità IncP) possono essere oggetto di trasferimento anche fra specie e generi diversi. I meccanismi di trasferimento nei batteri Gram negativi assomigliano molto a quello del fattore F di Escherichia coli descritto sopra, mentre nei batteri Gram positivi sono diversi e non sembrano riconducibili a un'unica tipologia. Un gruppo interessante di p. coniugativi sono i fattori col, presenti in ceppi di Escherichia coli e definiti colicinogeni in quanto responsabili della produzione di polipeptidi ad attività tossica contro altri batteri, le colicine (v. oltre). Le proprietà di trasferimento dei fattori col sono simili a quelle del fattore F e coinvolgono il ruolo di un pilus. In alcuni casi si tratta proprio dell'F-pilus e c'è incompatibilità tra il fattore F e col (è questo il caso di colV), cosa che induce a ritenere che il fattore col contenga informazioni genetiche proprie del fattore F. Tra i p. coniugativi vi sono anche alcuni fattori della classe R (fattori di resistenza, v. oltre), elementi genetici complessi che contengono una porzione deputata al trasferimento e alla produzione di un pilus. Il trasferimento di fattori R avviene anche fra specie diverse, contrariamente al fattore F, specifico per Escherichia coli.
Plasmidi integrativi
Alcuni p., oltre a esistere come elementi genetici autonomi, possono integrarsi nel cromosoma cellulare e vengono definiti plasmidi integrativi. Generalmente essi sono anche coniugativi e vengono tradizionalmente detti episomi. I fattori F, col e R appartengono al gruppo degli episomi. La presenza dei p. come elementi genetici autonomi nella cellula è uno stato molto stabile; tuttavia, con frequenza di un evento su circa 106, l'episoma si può integrare nel cromosoma e andare a costituire una condizione altrettanto stabile, in cui l'episoma integrato si replica in modo solidale al cromosoma, ritornando soltanto raramente allo stato autonomo. Tale fenomeno è stato studiato principalmente per il fattore F. L'integrazione non è casuale, ma avviene in siti specifici del DNA cromosomico, costituiti da piccole sequenze dette sequenze di inserzione o IS (Insertion Sequences), presenti anche nel fattore F (fig. 1). L'integrazione avviene quindi mediante ricombinazione omologa, cioè scambio fra sequenze uguali. In seguito all'inserzione, per effetto del meccanismo di ricombinazione, l'episoma sarà fiancheggiato da due sequenze IS uguali fra loro. Nella forma integrata il fattore F conserva le sue proprietà di trasferimento e quindi è in grado di coniugare con una cellula F−. Il trasferimento, come nel caso del fattore F non integrato, avviene mediante rottura di uno dei due filamenti cromosomici e trasferimento di un singolo filamento con simultanea sintesi dei due filamenti complementari nelle due cellule. Tale evento inizia con il trasferimento di DNA interno al fattore di fertilità a partire dal sito oriT e continua fino a separazione casuale delle due cellule. Ne consegue che: a) oltre al DNA plasmidico viene trasferito anche parte del cromosoma, a cominciare dalla porzione adiacente al punto di inserimento del fattore di fertilità; b) il trasferimento completo del fattore di fertilità, e quindi la trasformazione della femmina in maschio, avviene raramente, perché richiede il trasferimento dell'intero cromosoma. Pertanto, il maschio rimane tale e la femmina, pur avendo acquisito DNA del donatore, rimane femmina, salvo i rari casi in cui l'accoppiamento sia durato tanto a lungo (nel caso di Escherichia coli circa 100 minuti) da consentire il trasferimento, con l'intero cromosoma, anche della porzione distale del fattore di fertilità. Bisogna anche aggiungere che il DNA trasferito dal maschio alla femmina sarà da quest'ultima acquisito come materiale genetico proprio soltanto se esso ricombina con l'omologo DNA del proprio cromosoma, in quanto frammenti non circolari di DNA non possono replicarsi all'interno della cellula e vengono quindi persi. I ceppi contenenti un p. coniugativo integrato nel cromosoma vengono definiti Hfr (High frequency of recombination), in quanto in grado di trasferire frammenti di DNA cromosomico ad alta frequenza, rispetto alla bassa frequenza di un ceppo F+. In quest'ultimo caso la capacità di trasferire, seppur a bassa frequenza, frammenti di DNA cromosomico, è con ogni probabilità dovuta alla presenza, in una popolazione di cellule F+, di una piccola quota (generalmente una su 106 o meno) di cellule in cui il p. si è integrato. In Escherichia coli esistono numerosi Hfr diversi, ognuno dei quali contiene il fattore F inserito in un sito particolare.
L'incrocio di ognuno di essi con un F− trasferirà frammenti di DNA cromosomico diversi per lunghezza e per sequenza nucleotidica e ciò dipenderà dalla posizione in cui è inserito il fattore di fertilità e dal suo orientamento (orario o antiorario). Questa caratteristica è stata applicata per decenni in genetica batterica per studiare la posizione relativa dei geni sul cromosoma e per trasferire mutazioni da un ceppo a un altro.
Principali gruppi di plasmidi
Mentre il fattore F, pur di dimensioni rispettabili (circa 100.000 coppie di nucleotidi), non contiene particolari informazioni genetiche se non quelle connesse alla sua replicazione e al suo trasferimento (fig. 1), altri p. codificano per un'ampia gamma di funzioni (v. tabella). Essi vengono talvolta definiti plasmidi fenotipici, dal momento che sono capaci di conferire alle cellule caratteristiche particolari (fenotipi). Molto importanti sono i p. R già citati, per la capacità di alcuni di essi di trasferirsi e di integrarsi nel cromosoma. Sono presenti in numerosi batteri, da Escherichia coli e diverse altre specie di enterobatteri fino ai generi Pseudomonas e Staphylococcus.
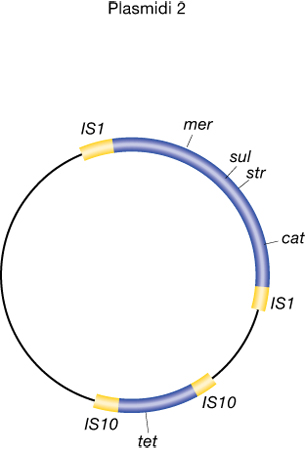
I p. R conferiscono alle cellule resistenza ad antibiotici oppure ad altre sostanze tossiche, codificando proteine che li modificano o li degradano, rendendoli innocui o impedendone l'ingresso nella cellula. Tali p. sono spesso coniugativi e quindi nel trasferirsi conferiscono alla cellula ricevente la resistenza agli antibiotici da essi codificata. Inoltre, assai di frequente i geni responsabili della resistenza sono situati all'interno di elementi genetici trasponibili, capaci cioè essi stessi di migrare dal p. al cromosoma o da un p. a un altro, aumentando ulteriormente la capacità di trasferimento della funzione. Tale proprietà crea seri problemi da un punto di vista epidemiologico, perché causa una diffusione delle resistenze, rendendo nel tempo inefficaci antibiotici comunemente usati negli ospedali e nelle terapie veterinarie. Uno dei più noti p. R è l'R100, un elemento genetico di ca. 100.000 coppie di nucleotidi, contenente geni per la resistenza al mercurio e a numerosi antibiotici (fra questi vi sono la streptomicina, il cloranfenicolo e la tetraciclina) e capace di trasferirsi per coniugazione fra diversi generi di batteri (fig. 2).
Altro interessante gruppo di p., anch'essi spesso coniugativi, sono i fattori colicinogeni, vale a dire produttori di colicine, polipeptidi ad attività antimicrobica con più alto peso molecolare rispetto agli antibiotici. Le colicine sono dette tali in quanto prodotte da ceppi di Escherichia coli. Oggi si tende a utilizzare il più generale termine di batteriocine, in quanto sono stati individuati molti batteri produttori di siffatti polipeptidi, spesso ma non sempre codificati da plasmidi. Abbiamo così la subtilisina (prodotta da Bacillus subtilis), le piocine (da Pseudomonas aeruginosa), le megacine (da Bacillus megaterium), le influenzacine (da Haemophilus influenzae). Le batteriocine agiscono generalmente contro specie strettamente correlate. I meccanismi d'azione sono molto diversificati e variano dalla formazione di canali sulle membrane delle cellule bersaglio alla degradazione del loro DNA o RNA. Normalmente un batterio colicinogeno è immune dall'azione tossica della batteriocina prodotta, grazie a specifici fattori proteici protettivi. La scoperta di batteriocine prodotte da microrganismi utilizzati nelle fermentazioni alimentari (quali i batteri lattici) ha stimolato molto interesse verso questi polipeptidi in quanto potenziali sostanze ad attività antimicrobica contro l'infezione degli alimenti a opera di batteri patogeni. Fra esse la più conosciuta è la nisina, già utilizzata da diversi anni per prevenire lo sviluppo di diversi patogeni umani nei prodotti lattiero-caseari.
Alcuni p., prevalentemente presenti in Pseudomonas, codificano per enzimi coinvolti nella degradazione di sostanze aromatiche quali toluene, naftalene e stirene. Si tratta di p. spesso coniugativi e di grosse dimensioni (fino a 500.000 coppie di nucleotidi), come i p. CAM e OCT, responsabili rispettivamente della degradazione biologica della canfora e dell'ottano. Molti rivestono grande interesse biotecnologico e sono perciò oggetto di approfonditi studi finalizzati all'uso dei batteri che li contengono in processi di detossificazione di siti contaminati o di sottoprodotti industriali tossici. Molti p. codificano per funzioni che sono responsabili della patogenicità batterica, e sono denominati fattori di virulenza. Alcuni contengono l'informazione genetica per la sintesi di tossine enteropatogene e di proteine di superficie responsabili dell'adesione delle cellule batteriche all'epitelio intestinale. È questo il caso di diversi ceppi di Escherichia coli e di Shigella. - Di origine plasmidica sono anche la tossina esfoliativa di Staphylococcus aureus, che causa la cosiddetta sindrome della cute ustionata, la tossina tetanica di Clostridium tetani e molte altre. In ceppi di Agrobacterium sono presenti p. che, in seguito a infezione di piante dicotiledoni a opera del batterio, trasferiscono parte del proprio DNA (il T-DNA) nelle cellule della pianta, inducendo una complessa serie di fenomeni che causa nella pianta un tumore denominato galla del colletto.
I plasmidi nell'ingegneria genetica
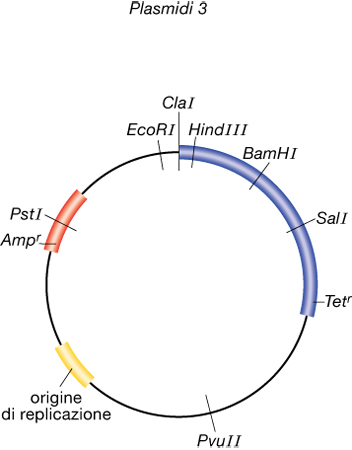
È possibile separare e purificare facilmente i p. rispetto al cromosoma mediante centrifugazione degli estratti cellulari in gradiente di densità, tecnica che trae profitto dalla netta differenza di avvolgimento e quindi di densità fra i due tipi di DNA. Ciò ha consentito negli ultimi decenni non solo di studiare a fondo i p. e le loro funzioni, ma anche di utilizzarli come vettori per il trasferimento genico programmato in laboratorio. Infatti, grazie agli enzimi di restrizione, che tagliano la doppia elica del DNA in punti ben definiti, è possibile tagliare un p. in un solo punto e in tal modo linearizzarlo. Aggiungendo poi frammenti di DNA estranei al p. in presenza di una ligasi (enzima che catalizza la formazione di legami fra molecole di DNA), si possono costruire p. cosiddetti chimerici mediante ricombinazione in vitro. Quando si reinserisce il p. nelle cellule mediante un processo di trasformazione (inserimento diretto di DNA in cellule opportunamente rese permeabili), esso, nel replicarsi, perpetuerà anche le funzioni geniche codificate dal DNA estraneo in esso inserite. Tali esperimenti, detti di clonaggio genico, consentono di produrre grandi quantità di DNA di interesse per vari tipi di studio, come di studiare i meccanismi di espressione di geni ignoti e di isolare ceppi batterici aventi proprietà nuove codificate da p. chimerici al fine di produrre quantità elevate di molecole di interesse farmaceutico oppure commerciale. Un p., per essere un buon vettore di clonaggio, deve essere presente nelle cellule con un alto numero di copie, deve essere relativamente piccolo per poter essere facilmente manipolato e deve codificare per funzioni quali la resistenza a uno o più antibiotici, in modo da poterne seguire il destino nelle cellule mediante selezione in presenza di antibiotici. Il primo importante vettore di clonaggio, fra i più versatili, è stato pBR322, un p. multicopie (circa 20 per cellula) di poco più di 4000 coppie di nucleotidi, ottenuto in laboratorio per successive ricombinazioni in vitro da altri plasmidi. Esso contiene geni per la resistenza a due antibiotici e vari siti unici di restrizione per il clonaggio di geni (fig. 3). La quantità di DNA che può essere inserito in un p. è limitata. P. ricombinanti contenenti quantità eccessive di DNA tendono a non riprodursi o a riarrangiarsi nelle cellule perdendo parte del materiale genetico. Allo scopo di clonare grossi frammenti di DNA sono stati costruiti in laboratorio derivati dei p., detti cosmidi, che contengono la regione cos del batteriofago lambda (un virus che infetta Escherichia coli) corrispondente alle estremità coesive del batteriofago. Tali costrutti consentono di inserire in modo stabile nella molecola ricombinante quantità elevate di DNA estraneo (fino a 40.000 coppie di nucleotidi). Essi vengono trasferiti in Escherichia coli a seguito di impacchettamento nel virus e infezione virale delle cellule batteriche, evitando così la trasformazione che, rispetto all'infezione fagica, è meno efficiente. Un'altra classe di vettori di clonaggio sono i vettori shuttle, che possono replicarsi in due organismi diversi grazie al fatto che in essi sono stati inseriti frammenti di DNA che consentono la replicazione del p. in entrambi. Il più noto è quello che può replicarsi in Escherichia coli e nel lievito Saccharomyces cerevisiae: esso contiene due origini di replicazione, ognuna attiva in uno dei due organismi e due geni per la selezione specifica (i marcatori ampicillina-resistenza per il batterio e leu per il lievito).
bibliografia
J.D. Watson, T.A. Baker, S.P. Bell et al., Biologia molecolare del gene, Bologna 1965, 20055.
M. Polsinelli, M. De Felice, A. Galizzi et al., Microbiologia, Torino 1993.
L.M. Prescott, J.P. Harley, D.A. Klein, Microbiologia, Bologna 1995.
B. Lewin, Il gene 6, Bologna 1999.
M.T. Madigan, J.M. Martinko, J. Parker, Brock biologia dei microrganismi, 2 voll., Milano 2003.