Le basi genetiche del cancro
Le basi genetiche del cancro
Dall'inizio degli anni Ottanta le ricerche sull'origine del cancro nell'uomo sono state principalmente volte all'identificazione e all'analisi funzionale degli oncogeni e dei geni soppressori tumorali. Queste ricerche hanno fornito dati decisivi a favore della base genetica del cancro, permettendo di formulare teorie sulla sua insorgenza e progressione, sulla sua origine monoclonale, sull'espansione clonale di popolazioni di cellule tumorali e sui meccanismi cromosomici degli eventi mutazionali. L'identificazione delle funzioni di questi geni ha ampliato le nostre conoscenze sulle alterazioni specifiche del cancro nella regolazione della crescita e della divisione cellulare, nonché sui meccanismi con cui le cellule tumorali interagiscono con le cellule normali. Nella pratica clinica si sono diffusi metodi diagnostici e prognostici, risultati tangibili di queste scoperte; si stanno allestendo appropriate strategie di intervento soprattutto nei riguardi di famiglie che presentano storie di predisposizione genetica al cancro e si stanno attivamente perseguendo terapie mirate a specifiche lesioni genetiche.
Il cancro come malattia genetica
Il cancro si manifesta in un' ampia varietà di tessuti in molte e differenti forme, e in tutte sono presenti alterazioni genetiche. L'osservazione che sia la perdita sia l'acquisizione di informazione genetica nelle cellule possano contribuire direttamente all'insorgenza del cancro fu effettuata per la prima volta circa un secolo fa, sebbene non fosse stata ampiamente accettata. Tuttavia, dall'inizio degli anni Ottanta gli studi di epidemiologia dei tumori, di cito genetica e di genetica molecolare delle popolazioni umane hanno fornito una grande quantità di evidenze a supporto di questa ipotesi.
L'epidemiologia genetica del cancro, spesso condotta nell'ambito di studi sulle famiglie, è in grado di fornire una prima valutazione del grado di associazione tra mutazioni ereditate e predisposizione al cancro. Questi studi hanno mostrato che il cancro si verifica con frequenze assai maggiori, o tende a 'raggrupparsi', in certe famiglie rispetto ad altre. Ciò è stato dimostrato per molti tumori comuni, come quelli della mammella, delle ovaie, del colon, della pro stata, del cervello e del polmone (Sidebottom et al., 1995). Il tipo di distribuzione dell'aumento di rischio, nell'ambito di queste famiglie, indica che il 'raggruppamento' è dovuto a una predisposizione ereditaria al cancro, piuttosto che alla comune esposizione a danni provocati da agenti ambientali. Questo punto è illustrato da due esempi ben documentati relativi al cancro della mammella e della prostata. In un ampio studio sul cancro familiare della prostata, gli aumenti di rischio relativi ai maschi con uno, due o tre parenti colpiti dalla malattia sono risultati rispettivamente di 2,2, 4,9 e 10,9. Il rischio aumentava ulteriormente (di 10÷11 volte) in individui con due parenti affetti, di cui uno diagnosticato prima dei 40 anni di età. Anche gli studi effettuati sul cancro della mammella, in individui con uno o più parenti di primo grado colpiti dalla malattia, hanno evidenziato un aumento di rischio, rispettivamente di 1,8 e 3,3 volte. Come per il cancro della prostata, un ulteriore incremento del rischio di cancro della mammella si verificava se per uno dei parenti la malattia era stata diagnosticata in età precoce (trenta o quaranta anni). Queste scoperte indicano che l'entità del rischio è funzione del numero di parenti affetti e che è effettivamente maggiore in quegli individui con più parenti nei quali la malattia si manifesta in età precoce. Tali evidenze sono a favore dell'ipotesi di una trasmissione ereditaria di un difetto che predispone al cancro.
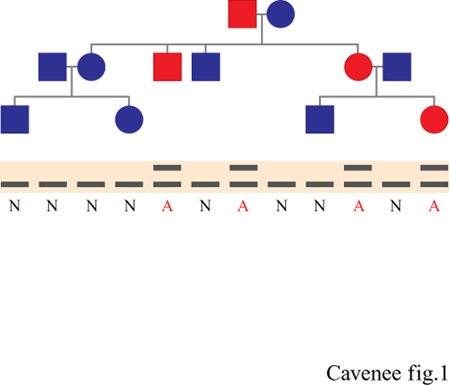
L'alta frequenza con cui alcuni tipi di tumore si manifestano nell' ambito di certe famiglie suggerisce la presenza di una componente ereditabile nell'eziologia del cancro, ma di per sé non offre alcun indizio sulle modalità con cui il carattere genetico della malattia viene trasmesso. La modalità di trasmissione del carattere canceroso nell'ambito di ciascuna di queste famiglie (per un ipotetico 'pedigree della malattia' vedi figura 1, in alto) si può dedurre confrontando i dati relativi alle singole famiglie con quelli previsti dai diversi modelli di trasmissione genica della malattia. Il modello che meglio spiega la maggior parte dei 'raggruppamenti' familiari di predisposizione al cancro, esemplificato nella famiglia presentata in figura (fig. 1), è detto eredità autosomica dominante (Sidebottom et al., 1995). In questo modello, l'ereditarietà della malattia non è legata al sesso; può essere trasmessa da entrambi i genitori e implica la trasmissione di uno o più geni, la cui presenza è sufficiente a causare l'insorgere della malattia. Questa modalità di trasmissione autosomica dominante prevede che la malattia insorgerà, in media, nel 50% della prole di un genitore affetto. Da un punto di vista epidemiologico, considerazioni sulla frequenza del 'raggruppamento' familiare di predisposizione al cancro, secondo il modello della trasmissione autosomica dominante, portano a concludere che il 5 ÷10% di tutti i tipi di cancro studiati è attribuibile all' ereditari età di uno o più geni dominanti; tutti gli altri tipi di cancro vengono detti sporadici.
Oltre al raggruppamento familiare di alcuni tipi comuni di cancro, vi sono due ulteriori osservazioni cliniche a supporto dell'esistenza, per il cancro, di un'eziologia genetica. Per prima cosa, alcuni individui e le loro famiglie presentano una trasmissione autosomica dominante di predisposizione non solo per un singolo tipo, come descritto precedentemente, ma per diversi tipi di cancro che si manifestano indipendentemente in differenti organi e tessuti del corpo. La seconda osservazione è che in individui che presentano difetti di sviluppo in differenti organi si manifestano spesso anche rare forme tumorali specifiche. Con un ragionamento statistico si può dedurre che l'insorgenza di più tumori indipendenti o la frequente associazione di difetti nello sviluppo con tumori che di per sé sono molto rari nella popolazione generale, sono così improbabili da suggerire una correlazione eziologica.
Gli argomenti statistici e i modelli di trasmissione derivati dagli studi di epidemiologia dei tumori offrono una base teorica su cui condurre la ricerca del gene (o dei geni) della malattia attraverso l'analisi genetica dell' associazione (linkage). Le tecniche di citogenetica e di genetica molecolare vengono utilizzate per identificare un'associazione statisticamente significativa tra il fenotipo della malattia e uno specifico difetto genetico (v. figura 1). Questa ricerca inizia con l'analisi al microscopio dei cromosomi delle cellule normali e tumorali di individui con una predisposizione ereditaria al cancro.
l cromosomi in metafase di cellule che vanno incontro alla mito si vengono colorati in modo che ciascuno di essi assuma una caratteristica serie di bande chiare e scure, una sorta di codice a barre per identificare ogni singolo cromosoma. L'osservazione delle alterazioni riproducibili rispetto al normale bandeggio cromosomico in tutte le cellule di individui affetti da cancro, e che sono assenti in parenti non malati, circoscrive la ricerca del gene della malattia alla regione cromosomica anomala. Purtroppo, le regioni cromosomiche identificate con le tecniche cito genetiche sono quasi tutte così grandi da contenere centinaia o anche migliaia di geni, di cui solo uno o pochi sono i veri responsabili del fenotipo canceroso. Gli strumenti della genetica molecolare, e in particolare la combinazione della tecnica di ibridazione nota come Southern blot e della reazione a catena della polimerasi (PCR, Polymerase Chain Reaction), vengono perciò utilizzati per ridurre la regione cromosomica sospetta a un segmento di DNA contenente solo pochi geni che possono essere analizzati direttamente per evidenziare i possibili difetti negli individui colpiti dalla malattia. Il procedimento che va dall'analisi epidemiologica alla citogenetica e alla genetica molecolare ha portato finora all'isolamento di 16 geni che, quando trasmessi nelle forme mutate, predispongono gli individui portatori allo sviluppo di certe forme tumorali.
Il retinoblastoma: un prototipo di cancro ereditario e sporadico
L'apparente trasmissione dominante di caratteri cancerosi descritta nel precedente paragrafo è paradossale alla luce di tre osservazioni: l) cellule ibride ottenute sperimentalmente dalla fusione di cellule tumorali fortemente maligne con cellule normali non presentano, in genere, proprietà tumorigene, e ciò suggerisce che il fenotipo normale sia dominante in presenza di mutazioni tumorigene; inoltre, nei pochi casi in cui una cellula ibrida diventa tumorigena, essa ha perduto cromosomi specifici originariamente forniti dalla cellula normale, e ciò implica che il fattore responsabile del fenotipo tumorale non è l'acquisizione di un carattere canceroso dominante, ma una perdita cromosomica specifica; 2) se una singola mutazione fosse di per sé sufficiente a provocare un tumore, ci dovremmo aspettare che famiglie che segregano forme autosomiche dominanti di cancro non presentino tessuto normale nell'organo malato; questa previsione è in aperta contraddizione con la descrizione clinica di tumori che presentano lesioni focali circondate da tessuto normale e funzionante dello stesso organo; 3) analisi epidemiologiche di forme sporadiche e familiari di diversi tipi di cancro umano, in contrasto con la trasmissione autosomica dominante, hanno indicato che la conversione di una cellula normale in cellula tumorale richiede una serie di eventi (Knudson, 1971).
Questi paradossi sono stati alla fine risolti attraverso lo studio del retinoblastoma, una forma tumorale che è servita da prototipo per la comprensione della genetica della grande maggioranza dei tumori ereditari e sporadici nell'uomo. Il retinoblastoma è un tumore relativamente raro (1 ogni 20.000 nascite) che colpisce i bambini e che si manifesta sia in forma ereditaria dominante autosomica sia in forma sporadica. Sulla base di soli dati statistici derivanti dall' epidemiologia e da osservazioni cliniche, sono state tratte importanti conclusioni circa la natura degli eventi che conducono alla formazione di questo tumore (Knudson, 1971). Per prima cosa, la mutazione ereditata non è da sola sufficiente a indurre la malattia, dato che vi sono almeno 10⁷ cellule (retinoblasti), ciascuna portatrice della mutazione ereditata, che potrebbero dare origine alla neoplasia (retinoblastoma), mentre in media si formano solo 3 tumori indipendenti in ogni individuo affetto. Ciò ha suggerito anche che a livello genetico le mutazioni che causano il retinoblastoma possano essere recessive, piuttosto che dominanti come suggerirebbe la modalità di trasmissione. È stato proposto che i tumori ereditari originino da una prima mutazione a livello della linea germinale, seguita da una seconda mutazione in una cellula somatica. La frequenza con cui si verificano mutazioni somatiche è simile sia nei tumori sporadici sia in quelli ereditari, sebbene i primi richiedano due mutazioni somatiche, ciascuna nello stesso retinoblasto, per la formazione del tumore. In pieno accordo con ciò è l'osservazione che i casi ereditari si verificano in genere in un'età più precoce, sono spesso bilaterali e sviluppano tumori multipli, mentre i casi sporadici sono sempre unilaterali e sviluppano singoli tumori. Poiché, nei casi ereditari, vi è una bassa possibilità che una seconda mutazione somatica non si verifichi mai, circa il 5% dei portatori non sviluppa alcun tumore. La natura dei due bersagli delle mutazioni nel genoma era sconosciuta al tempo di queste osservazioni, ma le tecniche di cito genetica e genetica molecolare hanno risposto a questo quesito e hanno offerto un approccio generale allo studio di altri tipi di cancro umano.
L'analisi del bandeggio cromosomico in pazienti affetti da retinoblastomi ereditari e sporadici ha rivelato una delezione del cromosoma l3q14 (cromosoma 13, braccio q o lungo, banda uno-quattro), suggerendo che il gene per il retinoblastorna (Rb) è localizzato in qualche parte di questa regione. Il DNA estratto dalle cellule di tumori ereditari è stato poi analizzato con sonde di DNA clonato (DNA markers) che potevano distinguere le due copie, o alleli, del cromosoma 13 in ciascuna cellula. È stato trovato che, in tumori di individui affetti, la regione contenente il gene sospetto Rb sul cromosoma 13 era presente solo in una forma mutata. Questa conversione da uno stato di eterozigosi a uno stato di omozigosi per la mutazione è stata chiamata perdita di eterozigosi (LOH, Loss of Heterozygosity) e costituirebbe il secondo evento necessario per la formazione di tumori nei casi ereditari (Cavenee et al., 1983). Inoltre, sul cromosoma l3q14 si verificava LOH anche nei casi di retinoblastoma sporadico. Questi dati hanno fornito un supporto notevole all'ipotesi secondo la quale la formazione del tumore potrebbe verificarsi in seguito allo 'smascheramento' di un difetto genetico recessivo. La scoperta che, nell'uomo, l'LOH si verifica in altre forme tumorali ereditarie e nella maggior parte dei tumori sporadici (Rodriguez et al., 1994) ha dato il via, contemporaneamente, alla genetica delle cellule cancerose somatiche, accoppiata alla genetica del cancro ereditario. È stato, infine, isolato il gene responsabile del retinoblastoma: le sue funzioni sono state determinate grazie all'identificazione della regione del cromosoma l3q14 con LOH più consistente nel DNA tumorale. L'ipotesi dei due eventi (two hit hypothesis) si è rivelata valida a livello genico per i retinoblastomi ereditari e sporadici ma, come si vedrà nel prossimo paragrafo, si presenta altrettanto valida anche per spiegare la formazione di quasi tutti gli altri tipi di tumore, sia ereditario sia sporadico.
Altri geni soppressori tumorali
Geni identificati dalla predisposizione familiare
Tramite l'analisi dell'associazione e gli studi di LOH nel DNA delle cellule tumorali, sono stati identificati 16 geni che, se ereditati in forme mutanti, causano la predisposizione verso specifici tipi di cancro. In maniera simile al retinoblastoma, ciascuna di queste predisposizioni al cancro viene trasmessa come carattere autosomico dominante e, a livello genico, tutte eccetto una (oncogene RET) sono dovute a un allele mutante recessivo ereditato che viene smascherato in una cellula somatica da una mutazione successiva o dalla perdita della rimanente copia normale. Questi geni sono detti soppressori tumorali, in quanto la perdita delle loro normali funzioni di controllo della crescita porta direttamente o indirettamente alla formazione del tumore. Considerando solamente i tumori ereditari, il numero totale di potenziali geni soppressori tumorali umani è stato stimato in circa 50 (tab. Ia, b), dato che questo è il numero di differenti tipi di tumori ereditari descritti mediante studi epidemiologici e clinici (Mulvihill, 1977).
Il tumore di Wilms del rene è stato il secondo tumore embrionale dei bambini nella cui eziologia è stato riscontrato il coinvolgimento dell'LOH. Questo particolare tipo di tumore può manifestarsi da solo o in associazione con uno dei diversi disordini genetici nello sviluppo, tra cui l'aniridia sporadica o le sindromi W AGR, di Denys-Drash o di Beckwith-Wiedemann. A differenza del retinoblastoma, almeno tre diversi geni possono predisporre al tumore di Wilms, solo uno dei quali, WT1, è stato clonato (Sidebottom et al., 1995).
Nonostante le comuni modalità di trasmissione che si riscontrano tra i tumori ereditari, la percentuale di portatori del gene che, di fatto, sviluppa la malattia varia a seconda dei diversi tipi di cancro. Per esempio, il 90-95% di individui che ereditano mutazioni nel gene Rb sviluppano il retinoblastoma (Knudson, 1971). Il 5 ÷10% dei portatori che non sviluppano il retinoblastoma sembra riflettere la piccola possibilità, statisticamente ben definita, che non avvenga una seconda mutazione in una cellula somatica. Al contrario, la trasmissione ereditaria del tumore di Wilms mostra una penetranza incompleta essendo non penetrante in alcune generazioni, ma altamente penetrante nelle generazioni successive. Ciò potrebbe essere dovuto a modificazioni epigenetiche del carattere della malattia, dato che nei tumori di Wilms sporadici ed ereditari vengono perduti preferenzialmente gli alleli materni. Individui con mutazioni BRCAl hanno una penetranza specifica del tumore sia per il cancro della mammella (80%) sia per quello ovarico (40%). La penetranza della malattia in individui con mutazioni BRCAl è modificata da un secondo gene, HRAS1. l portatori di BRCAl che portano un allele HRSAl che è raro nella popolazione generale hanno probabilità 2,1 volte maggiori di sviluppare il cancro ovarico rispetto a quelli in cui l' allele HRSAl è quello più comune. Il significato funzionale di questo allele raro è ancora sconosciuto, ma questa è la prima osservazione che mostra l'effetto di un gene modificatore sulla penetranza di una sindrome di cancro ereditaria.
Sebbene l'inattivazione di molti geni soppressori di tumori richieda una mutazione che causa perdita funzionale in entrambe le copie del gene normale, cellule eterozigoti per forme mutanti di altri geni soppressori tumorali, come p53 o WT1, possono avere un vantaggio nella crescita rispetto a cellule normali per un effetto dominante negativo. La proteina p53 è attiva in forma tetramerica ed è resa inattiva dall'incorporazione di uno o più monomeri dip53 mutante che ha conservato la capacità di legarsi con altre subunità e formare oligomeri. Similmente, nel tumore di Wilms una mutazione della proteina WT l, che risulta alterata per il legame con il DNA, contrasta l'attività della normale WTl formando oligomeri con questa. In tal modo, cellule eterozigoti per i mutanti p53 o WTl possono avere un vantaggio nella crescita, se vengono espressi sia il gene mutante sia quello normale. Oltre alla perdita di funzione o alla attività dominante negativa, p53 può acquisire nuove proprietà, mediante mutazioni di acquisto di funzione che determinano un aumento delle proprietà tumorigene in cellule mancanti di p53 normale.
Il gene RET, che è coinvolto nella neoplasia endocrina multipla di tipo 2 (MEN2, Multiple Endocrine Neoplasia type 2) e nel carcinoma familiare midollare tiroideo (FMTC, Familial Medullary Thyroid Carcinoma) è, inoltre, l'unico gene che predispone al cancro noto per essere un soppressore tumorale (Mak e Ponder, 1996). MEN2 e FMTC vengono trasmessi in maniera autosomica dominante, ma la trasmissione uniparentale di un solo gene RET mutato può essere sufficiente a provocare la malattia, anche in presenza di un allele RET normale. L'analisi funzionale delle proteine RET mutate ha rivelato un'attivazione costitutiva dell'attività intrinseca della tirosinachinasi e un'alterata specificità peptide-substrato. In tal modo il gene RET sembra seguire un modello di oncogene dominante, piuttosto che una perdita della funzione di soppressore. È, comunque, alquanto paradossale che i tumori associati al gene RET siano di natura focale, come avviene per i tumori ereditari associati a geni soppressori, invece di coinvolgere tutte le cellule dei tessuti endocrini colpiti, come ci si potrebbe attendere da un allele mutante dominante. È dunque possibile che sia necessaria una seconda alterazione genetica o epigenetica per la formazione del tumore, come viene ulteriormente suggerito dalla presenza di mutazioni sia somatiche sia germinali del gene RETnei tumori di numerosi individui affetti da FMTC. Alterazioni del gene RET sono coinvolte anche nei tumori sporadici, inclusi i carcinomi midollari surrenali e della tiroide. Un'alta incidenza di alterazioni del gene RET è stata osservata nei tumori della tiroide di bambini della Bielorussia, nella ex Unione Sovietica, diversi anni dopo l'incidente nucleare di Cernobyl del 1986, suggerendo così la possibilità di alterazioni del gene RET indotte dalle radiazioni.
Diversi geni soppressori tumorali, identificati grazie allo studio della predisposizione ereditaria al cancro, sono coinvolti anche in uno spettro piuttosto ampio di tumori sporadici. Ciò è ben rappresentato da tre geni, Rb, p53 e p16/ CDKN2. A causa del numero limitato di forme tumorali trovate in pazienti con retinoblastoma ereditario, è stato piuttosto sorprendente scoprire che il gene del retinoblastoma è alterato in molti tumori sporadici di individui adulti come nel cancro del polmone, della mammella, della prostata, del pancreas, del cervello, nei sarcomi dei tessuti ossei e molli, nelle leucemie e nei linfomi (Sidebottom et al., 1995). Per molti di questi tumori sporadici, l'LOH del gene Rb sembra essere coinvolta nella progressione del tumore piuttosto che nel suo stadio iniziale. Le mutazioni del gene p53 nella linea germinale predispongono a molti tipi di tumori nella sindrome di Li-Fraumeni, oppure possono predisporre a un singolo tipo di tumore (per esempio, tumori cerebrali). Le mutazioni del gene p53 nella linea somatica costituiscono l'alterazione genetica più frequentemente osservata nei tumori sporadici; si verificano infatti in circa il 50÷60% di tutti i tumori esaminati, inclusi quelli della mammella, del colon, del fegato, del polmone e del cervello. Le mutazioni del gene p53 insorgono negli stadi precoci dei tumori del cervello e del polmone, ma assai più tardi nel tumore del colon (Fearon e Vogelstein, 1990), suggerendo che la mutazione di p53 può essere coinvolta sia nello stadio iniziale sia nella progressione dei tumori. Alterazioni del gene p16, presenti nel melanoma familiare, sono frequenti anche in tumori sporadici, per esempio quelli del pancreas, della vescica, della testa e del collo, delle cellule renali, del polmone, della mammella, della pro stata e del cervello (Pollock et al., 1996). È probabile che la diversità delle forme tumorali che coinvolgono mutazioni nei geni Rb, p53 o p16 sia dovuta all'espressione ubiquitaria di questi geni nelle cellule normali. l geni BRCAl e BRCA2 per i tumori ereditari, rispettivamente della mammella e di mammella e ovaie, non sono in genere mutati nei tumori sporadici della mammella, costituendo in tal modo due importanti eccezioni al doppio coinvolgimento di geni sia in tumori ereditari sia sporadici. Per altri geni soppressori tumorali, il coinvolgimento esclusivo in uno specifico tipo di tumore (per esempio, il tumore di Wilms) può essere spiegato da un'espressione limitata nel tempo e nel tessuto (per esempio, l'espressione di WTl durante lo sviluppo del rene e nei tessuti delle gonadi). In alcuni casi, la modalità di espressione di un gene soppressore tumorale nei tessuti normali può fornire indizi sul tipo di tumore che può essere innescato dalla sua mutazione.
Oltre alla segregazione con la predisposizione al cancro, la perdita di funzione di geni soppressori tumorali può essere verificata sperimentalmente mediante tecniche di transfezione e delezione. Il gene Rb normale inibisce la crescita delle cellule tumorali se inserito, tramite transfezione, in cellule tumorali in coltura mancanti di copie funzionali di Rb. Questa osservazione è accompagnata anche dalla soppressione di altre caratteristiche maligne di queste cellule, come la perdita dell'inibizione da contatto e la capacità di generare tumori in topi immunodepressi. Topi che presentano un knock out mirato di una copia del gene Rb mostrano una grande predisposizione a sviluppare tumori, anche se non sviluppano retinoblastomi. Non è ancora chiaro se ciò dipenda dal minor numero di retinoblasti presenti nei topi (Knudson, 1993) o, semplicemente, sia correlato a differenze interspecifiche.
A livello molecolare si hanno conoscenze approfondite sulla normale funzione della proteina Rb nella regolazione della crescita cellulare (Hunter e Pine s, 1994). Rb è una fosfoproteina nucleare di 110 kDa che nella forma non fosforilata lega il fattore di trascrizione E2F e inibisce la progressione del ciclo cellulare nel punto di controllo (checkpoint) G1/S. L'interazione con complessi di ciclina Dl-CDK4 (chinasi ciclina-dipendente 4) o di ciclina DlCDK6 inattiva Rb mediante fosforilazione, promuovendo così la transizione attraverso la fase G l del ciclo cellulare. l regolatori di Rb sono a loro volta regolati, aggiungendo in questo modo livelli ulteriori di complessità al controllo del ciclo cellulare, mediato da Rb. Almeno due inibitori di CDK, p15 e p16, possono legarsi e inibire CDK4 e CDK6. Mutazioni del gene p16 nella linea germinale sono responsabili della predisposizione ereditaria al melanoma (v. tabella la, b), fornendo così, inaspettatamente, un meccanismo comune per l'origine di due neoplasie ereditarie molto differenti. Nei tumori sporadici l'alterazione genetica o epigenetica di uno di almeno sei di questi componenti della via di Rb può eliminare l'inibizione della crescita cellulare di Rb e contribuire al carattere maligno, sia tramite una perdita di funzione (Rb, p16, p15) sia attraverso un'acquisizione di funzione/sovrapproduzione (CDK4, CDK6, ciclina DI), sottolineando così l'importanza di questa via nel cancro umano. Rb inibisce la funzione della proteina ABL e interagisce anche con diverse proteine scoperte recentemente, RBPl, RBP2, BRGI e RlZ; tuttavia non sono ancora stati identificati i ruoli specifici di queste interazioni nelle cellule normali e cancerose.
È facile immaginare come la perdita di alcuni geni soppressori, come quelli che regolano negativamente il ciclo cellulare (Rb, p16, p53) o quelli che inibiscono i segnali positivi di crescita (NF l, APC), contribuisca direttamente allo sviluppo di tumori. La perdita di altri geni, come i quattro geni MSH2, MSH1, PMSl e PMS2 (Fearon, 1995) coinvolti nella riparazione di errati appaiamenti di basi del DNA (MMR, Mismatch Repair), richiede una spiegazione indiretta e dunque più complessa per rendere conto della perdita del controllo della crescita e della predisposizione al cancro. Una possibilità è che una riparazione inadeguata del DNA porti a un incremento della frequenza di mutazione per condurre, infine, alla mutazione di un altro gene (o di altri geni) che esercita un effetto diretto sulla crescita o sulla divisione cellulare. Un potenziale gene effettore, che codifica il recettore di tipo II per il fattore trasformante di crescita β (TGF-β RII), è mutato nel suo tratto di poliadenina nel 90% dei tumori colon-rettali in coltura che mostrano instabilità genomica; in alcuni di essi è presente un gene MMR mutato. L'effetto di inibizione della crescita da parte di TGF-β, che richiede recettori sia di tipo I sia di tipo II, è abolito nella maggior parte dei tumori colon-rettali RER. Questi dati suggeriscono che le mutazioni di TGF-B RII potrebbero ricoprire un ruolo nel mediare l'effetto soppressore dei geni per la riparazione del DNA. Non è ancora chiaro se il tipo di mutazioni di TGF-β RII identificate in questo studio siano sufficienti a produrre un tale effetto o se il gene TGF-f3 RII sia un bersaglio critico, dato che diversi altri siti del genoma in queste cellule risultano mutati con frequenze anche più alte. Nonostante ciò, è chiaro che l'alterazione genetica critica che predispone a questi tipi di tumore del colon è la perdita di funzione tramite mutazione ereditaria e LOH di un gene soppressore tumorale con attività di riparazione MMR del DNA.
Geni identificati grazie all'instabilità cromosomica
Difetti nella riparazione del DNA sono implicati anche nella forte predisposizione al cancro che accompagna quattro sindromi di instabilità cromosomica (tab. 2). A differenza di altre forme di predisposizione al cancro, queste sindromi sono trasmesse in forma autosomica recessiva e sono pertanto assai rare: circa 1 caso su 300.000 per l'atassia-teleangectasia (AT), ossia da dieci a quindici volte meno del retinoblastoma. Fino a oggi, sono stati donati il gene responsabile dell'atassia-telangectasia, sei dei sette geni responsabili dei differenti gruppi di complementazione dello xeroderma pigmento so (XP) (Sidebottom et al., 1995) e uno dei tre geni responsabili della sindrome di Fanconi (FA).
Un'aumentata predisposizione al cancro, in individui omozigoti per un gene mutato, è drammaticamente evidente nei pazienti affetti da AT e XP. Rispetto alla popolazione generale, i pazienti affetti da AT manifestano un aumento del rischio di sviluppare leucemia o linfoma, rispettivamente di 70 e 250 volte. L'incidenza del carcinoma basale e di quello dell' epitelio a cellule squamo se nei pazienti con XP è ben 2000 volte maggiore rispetto a gruppi della stessa età della popolazione generale (Sidebottom et al., 1995). Pur essendo gli individui omozigoti rari, il numero di eterozigoti è ovviamente molto più grande. Diversi studi epidemiologici suggeriscono che gli eterozigoti per il gene dell'AT, che si stima comprendano l'1% della popolazione generale, potrebbero essere predisposti a sviluppare il cancro con un incremento di rischio generale di tre-quattro volte e fino a cinque volte nel caso dei tumori della mammella nelle donne eterozigoti. Dato che la sensibilità degli eterozigoti per AT alle radiazioni è intermedia tra quella dei pazienti omozigoti e degli individui non affetti, l'impiego della mammografia per diagnosticare il tumore della mammella è assolutamente da evitare negli eterozigoti per AT. È stato suggerito, ma non provato, che la sensibilità alle radiazioni e la predisposizione al cancro negli eterozigoti per AT potrebbe dipendere dal do saggio genico (un solo gene AT normale). Il recente donaggio del gene AT permetterà di verificare queste interessanti scoperte epidemiologiche a livello molecolare. Un'analisi preliminare del gene AT in 38 tumori della mammella sporadici scelti a caso, e nelle corrispondenti cellule normali del sangue, non ha rivelato un aumentato numero di eterozigoti per AT.
Geni identificati nei tumori sporadici
Oltre all'identificazione di geni soppressori tumorali tramite i difetti genetici ereditati, diversi geni candidati sono stati isolati attraverso studi della LOH nei tumori sporadici (tab. 3). Il numero sempre crescente di marcatori del DNA disponibili e lo sviluppo di analisi PCR multipla, cioè su più regioni contemporaneamente, basata sulla fluorescenza e assistita da computer, ha fortemente aumentato le possibilità applicative dell'analisi dell'LOH. Nuove metodiche di esplorazione del genoma, come RLGS (Restriction Landmark Genomic Scanning) e RDA (Represantational Difference Analysis), potranno essere d'aiuto nell'identificazione di questi geni. Dati sulle sequenze provenienti dal progetto "genoma umano" faciliteranno senz'altro questi studi.
Meccanismi cromosomici responsabili dell'inattivazione dei geni soppressori tumorali
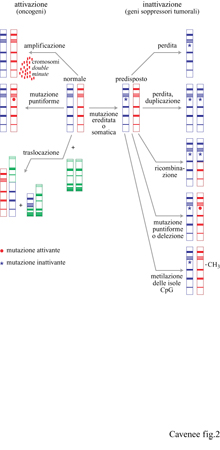
Come ricordato nell'introduzione, nei tumori umani sono stati osservati sia acquisizione sia perdita di informazione genetica. A livello genico le perdite riflettono l'inattivazione di geni soppressori tumorali. La perdita cromosomica è la più comune modificazione genetica osservata nella maggioranza dei tumori umani (Rodriguez et al., 1994), con l'eccezione delle leucemie e dei linfomi. Lo smascheramento di alleli tumorali recessivi tramite LOH è stato discusso nel contesto dei tumori sporadici ed ereditari. l meccanismi con cui avviene l'LOH (Cavenee et al., 1983) sono schematizzati in figura (fig. 2). Esempi di questi meccanismi sono stati osservati per la prima volta nel retinoblastoma sporadico ed ereditario, e in seguito praticamente in tutti i tipi di cancro umano.
Per alcuni geni soppressori tumorali operano tutti e cinque i meccanismi, sebbene uno di essi possa essere predominante. l primi due meccanismi elencati, perdita cromosomica e perdita/duplicazione, risultano nell'LOH per tutti i loei di un particolare cromosoma e nel conseguimento dell'omozigosi per la mutazione iniziale. l tumori cerebrali costituiscono un esempio di perdita cromosomica: la monosomia del cromosoma 10 è frequentemente associata alla progressione della forma tumorale più maligna, il glioblastorna. Un esempio di perdita cromosomica, seguita da duplicazione del cromosoma mutante, è quella del cromosoma 3 nel melanoma uveale. In contrasto con questi due meccanismi, la mutazione seguita da ricombinazione somatica risulta nell'LOH solo per una porzione del cromosoma che comprende la regione contenente il gene mutato. L'identificazione delle regioni del cromosoma che costituiscono siti di ricombinazione o delezione comuni a molti tumori spesso porta alla localizzazione cromosomica dei geni soppressori tumorali e viene detta mappatura per delezione. Il gene p16/CDKN2 è stato identificato attraverso l'uso estensivo di questa tecnica sul cromosoma 9p21. In molti e diversi tipi di tumore, la delezione omozigote è un meccanismo comune di inattivazione del gene p16/CDKN2 e del gene p19 sovrapposto (Pollock et al., 1996). Le mutazioni puntiformi, principali responsabili dell'inattivazione del gene p53 (Hollstein et al., 1996), vengono identificate con tecniche di genetica molecolare e sequenziamento diretto dei nucleotidi. Come suggerito dalla figura 2, mutazioni puntiformi separate su entrambi gli alleli potrebbero inattivare completamente la normale funzione del gene soppressore senza LOH, sebbene la frequenza di questo evento sia sconosciuta. La natura e la frequenza delle mutazioni in centinaia o forse migliaia di tumori sono state catalogate per diversi geni e sono reperibili in varie pubblicazioni (per esempio, per il gene P 16/CDKN2, Pollock et al., 1996; per il gene Rb, Lohmann et al., 1996; per il gene p53, Beroud et al., 1996) o su Internet (BRCAl, Breast Caneer lnformation Consortium, BlC). La catalogazione delle mutazioni ha fornito una molteplicità di evidenze sulla presenza di 'punti caldi' (hot spots) per le mutazioni all'interno dei geni, come i codoni 175, 248 e 273 nel gene p53, che possono render conto di quasi un quarto di tutte le mutazioni di questo gene, sia considerando tutti i tipi di tumore (Hollstein et al., 1996) sia solo tipi particolari (per esempio, il tumore del cervello, Bögler et al., 1995). In molti geni del cancro, punti caldi per le mutazioni spesso indicano domini funzionali critici della proteina codificata; per esempio, quelli nel gene p53 riguardano domini proteici essenziali per l'interazione col DNA.
La metilazione delle isole CpG è forse il meccanismo d'inattivazione dei geni soppressori tumorali meno studiato. Le isole CpG sono brevi tratti di DNA ricchi di dinucleotidi CpG e sono presenti all'estremità 5' di circa il 50÷60% di tutti i geni noti. Le isole CpG si mantengono in uno stato demetilato in tutti i tessuti normali ma, con un meccanismo non ancora noto, quelle all'estremità 5' di diversi geni soppressori tumorali diventano metilate nelle cellule tumorali, parallelamente alla perdita dell'espressione dei geni. Queste 'mutazioni' non modificano la sequenza nucleotidica, ma influenzano l'espressione genica e sono perciò considerate epigenetiche. Il cosiddetto silenziamento epigenetieo anomalo, tramite metilazione delle isole CpG nelle cellule tumorali, è stato descritto per i geni VHL, Rb,p16/CDKN4 ed E-eaderina (Jones, 1996). Poiché molti geni soppressori tumorali sono inattivati in prevalenza mediante un solo meccanismo, è possibile che esistano geni soppressori inattivati prevalentemente tramite metilazione delle isole CpG. Questi geni sarebbero sfuggiti all'analisi genetica convenzionale del DNA dei tumori. Recentemente, un gene candidato soppressore tumorale, HIC-l (Hypermetilated In Cancer), è stato identificato analizzando una particolare regione cromosomica che presenta un'alterata metilazione delle isole CpG; si è visto che questo gene è alterato con alta frequenza negli stadi precoci della progressione dei tumori cerebrali.
Oncogeni
La scoperta degli oncogeni dominanti ha in realtà preceduto di circa un decennio quella dei geni soppressori, ed è stata realizzata grazie allo studio dei retro virus trasformanti (Bishop, 1995). L'idea che i virus stessi fossero in grado di provocare il cancro, o che l'identificazione dei geni trasformanti codificati da questi virus potesse portare all'identificazione di analoghi geni tumorali endogeni nell'uomo, si è rivelata fondata. Un numero limitato di tumori è fortemente associato a specifiche infezioni virali. Essi comprendono illinfoma di Burkitt e il carcinoma naso-faringeo associati al virus di Epstein-Barr, il tumore del fegato associato al virus dell'epatite B, il tumore della cervice uterina al papillomavirus e la leucemia delle cellule T al relativo virus. Degli oltre 20 oncogeni identificati grazie all'omologia con i geni trasformanti dei retro virus, solo per pochi (KRAS, HRAS, ERBB, ERBB2, MYC, NMYC) è stato dimostrato un coinvolgimento nel cancro umano. Attualmente, più di cento geni sono stati catalogati come oncogeni, anche se ciò dipende probabilmente dalla inclusione di tutti i geni con capacità trasformante dominante, indipendentemente dall'effettivo coinvolgimento nel cancro umano (Bishop, 1995). La comprensione dei meccanismi cromosomici che attivano gli oncogeni ha portato all'identificazione della maggior parte di tali geni, noti per il loro coinvolgimento nelle neoplasie umane.
I protooncogeni cellulari, geni che normalmente hanno un ruolo regolatore positivo nella crescita, possono essere modificati in forme oncogeniche mediante amplificazione genica, mutazioni punti formi o traslocazioni reciproche (v. figura 2). L'amplificazione genica è definita come l'incremento numerico (che può essere di oltre cento volte in alcune cellule cancerose) di segmenti di DNA compresi tra 100 kb e 2÷3 Mb e può presentarsi come una delle due seguenti anomalie citogenetiche: come regioni che si colorano in modo omogeneo (HSR, Homogeneously Staining Regions) quando fanno parte di un cromosoma, oppure sotto forma di minicromosomi soprannumerari, detti double minutes (DM), quando sono extracromosomici. Le regioni amplificate, dette ampliconi, contengono spesso diversi geni e nella maggioranza dei casi, ma non sempre, vi è una corrispondente sovraespressione del gene amplificato (o dei geni amplificati) che contribuisce all'aumentato potenziale di crescita e al carattere maligno delle cellule affette. Il gene amplificato può anche subire dei riarrangiamenti diventando un oncogene sovraespresso, costitutivamente attivo e deregolato. L'amplificazione genica è un evento che si osserva essenzialmente nelle fasi tardive dello sviluppo del tumore ed è correlata più verosimilmente alla progressione, piuttosto che allo stadio iniziale del tumore stesso (Rodriguez et al., 1994).
L'attivazione degli oncogeni per mezzo di mutazioni puntiformi produce in genere un segnale positivo di crescita che è attivo in modo costitutivo, come nel caso delle mutazioni del gene RAS. Il gene RAS è importante nella trasduzione del segnale da vari recettori della superficie cellulare, inclusi quelli per i fattori di crescita (Bos et al., 1995). Il segnale trasdotto da RAS, coadiuvato da una serie di proteine regolatrici e adattatrici, alla fine raggiunge il nucleo e attiva l'espressione della crescita. Mutazioni puntiformi nel gene RAS, presenti nel 25% di tutti i tumori umani e fino al 90% in certi tipi di tumori (per esempio, nei carcinomi del pancreas), comportano una continua trasduzione del segnale via RAS, anche in assenza di segnali extracellulari. Le mutazioni puntiformi di RAS sono altamente specifiche e sono state trovate quasi tutte nei codoni 12, 13 o 61. Un elenco parziale degli oncogeni attivati per amplificazione genica o per mutazioni puntiformi, che comprende quelli precedentemente discussi, è presentato in tabella (tab. 4). Questi oncogeni possono essere suddivisi in cinque gruppi in base alle loro funzioni note e alla localizzazione cellulare e comprendono fattori di crescita extracellulari, recettori transmembrana dei fattori di crescita, trasduttori intracellulari di segnali e fattori nucleari di regolazione del ciclo cellulare e di trascrizione. Indipendentemente dal meccanismo di attivazione, questi oncogeni agiscono in maniera dominante per promuovere il carattere maligno.
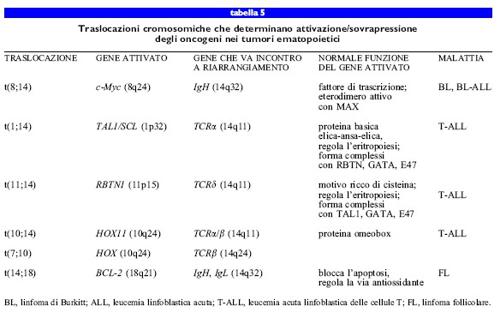
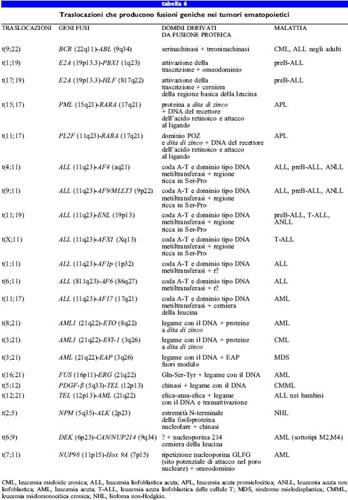
Un terzo meccanismo che porta all'attivazione degli oncogeni è la traslocazione cromosomica (v. figura 2), che costituisce il meccanismo eziologico primario delle leucemie e dei linfomi (Rabbitts, 1994). La traslocazione implica due rotture cromosomiche, in genere su due cromosomi diversi, seguite da scambio reciproco e riunificazione di un segmento di un cromosoma con quello di un altro. Le inversioni cromosomiche sono meno comuni e coinvolgono eventi in un singolo cromosoma, come rottura, inversione e riattacco. Le due principali conseguenze delle inversioni e delle traslocazioni sono: l) la giustapposizione di un gene che codifica un'immunoglobulina o il recettore della cellula T con un protooncogene, in modo da attivare quest'ultimo e fado esprimere in maniera aumentata e non regolata; 2) una fusione genica conseguente alla riunificazione, che codifica una proteina trasformante chimerica, nel caso che ambedue i punti di rottura cromosomica siano situati all'interno di introni.
Nella leucemia e nei linfomi, il frequente coinvolgimento dei geni per le catene pesanti e leggere delle immunoglobuline (rispettivamente 19H e 19L) e dei geni per i recettori delle cellule T nell'attivazione dei protooncogeni sembra riflettere aberrazioni nel processo naturale con cui questi geni sono riarrangiati nelle cellule ematopoietiche, per generare i geni per i recettori degli antigeni. La prima traslocazione, caratterizzata dal punto di vista molecolare, consiste in una giustapposizione del protooncogene Myc, già noto per essere attivato mediante amplificazione genica nei tumori solidi, con un gene IgH in una neoplasia maligna dei linfociti B, illinfoma di Burkitt (BL). La traslocazione di Myc e la conseguente sovraespressione si verificano nel 90% dei casi di BL. La sovraespressione del gene Myc (che codifica un fattore di trascrizione) altera l'equilibrio di una serie di fattori di trascrizione che coinvolge almeno altri tre fattori, MAD, MAX e Mxi-l. La sovraespressione di Myc sposta l'equilibrio verso un eccesso di dimeri MycMAX, necessari per l'attività dell'oncogene. Altri geni con diversi ruoli nella regolazione dello sviluppo (HOXI I), nel differenziamento (TALI /SCL) e ne II ' apoptosi (BCL- 2) sono attivati o sovraespressi per mezzo di simili eventi di traslocazione (tab. 5).
Relativamente all'attivazione dei protooncogeni, la fusione genica e l'espressione di proteine chimeriche potrebbero essere le conseguenze più comuni degli eventi di traslocazione (tab. 6). Queste traslocazioni reciproche producono due potenziali fusioni, entrambe le quali potrebbero essere espresse, ma in genere nelle cellule tumorali si ritrova solo un trascritto di fusione. Nella stessa cellula possono anche essere presenti i trascritti dei geni normali di ciascun omologo non coinvolto nella traslocazione, cosicchè le proteine chimeriche possono contribuire al carattere maligno mediante un meccanismo dominante negativo. Nelle cellule della leucemia mieloide acuta (AML, Acute Myelogenous Leukemia) l'inibizione dominante negativa del recettore normale dell'acido retinoico (RARa), causata dalla fusione PLZF-RARa, e quella della proteina normale AML (CBFA2, una subunità del fattore di trascrizione CBF) da parte delle proteine di fusione AMLl/ MDSl o AML/EAP, sono state dimostrate in vitro. Tuttavia, nel 50% delle leucemie linfocitiche acute con traslocazioni di ALL-I, il gene ALL-I (detto anche MLL o Hrx) si fonde con sé stesso lasciando la cellula affetta senza nessun gene ALL- I normale, con un fenomeno che ricorda l'inattivazione dei geni soppressori tumorali. Le cellule staminali embrionali (ES, Embrionic Stem) con un doppio knock out del gene ALL- I mostrano colonie con due fenotipi, simili alle cellule ematopoietiche anormali dei pazienti leucemici e crescono più velocemente delle cellule parentali o delle cellule ES in cui è stato inattivato un solo gene ALL, suggerendo che la perdita della funzionalità del gene ALL possa contribuire al carattere maligno (Fidanza et al., 1996). Da questi studi emerge la possibilità che ALL possa agire come gene soppressore tumorale piuttosto che come oncogene dominante.
Le fusioni geniche identificate nelle neoplasie maligne delle cellule ematopoietiche implicano, in genere, dei fattori di trascrizione; tuttavia sono state anche identificate fusioni con proteine che legano RNA (TLS/FUS), con domini della tirosinachinasi (ABL), omeodomini (HOX9A7) e proteine dei pori nucleari (NUP98, CAN/NUP2l4). Il primo prodotto di fusione caratterizzato dal punto di vista molecolare è stato BCR-ABL, presente nella maggior parte delle leucemie mielogene croniche (CML) e spesso anche nelle leucemie linfoblastiche acute (ALL). Questo rappresenta anche il primo caso di traslocazione reciproca caratterizzato dal punto di vista cito genetico: il cromosoma 22 risultante è stato denominato cromosoma Philadelphia (Rabbitts, 1994). Il gene ABL, l'omologo umano dell'oncogene virale che causa la leucemia murina di Abelson, codifica una tirosinachinasi citoplasmatica, mentre BCR codifica una proteina con funzioni ignote. La conseguenza principale della fusione BCR-ABL è una aumentata e non regolata attività tirosinachinasica; quindi, si tratta probabilmente di una mutazione del tipo acquisto di funzione. Topi transgenici BCR-ABL e topi normali irradiati che hanno subito trapianto di midollo spinale con cellule che esprimono BCR-ABL sviluppano neoplasie maligne e ciò indica che BCR-ABL agisce in maniera dominante nel provocare lo sviluppo del carattere maligno.
Traslocazioni che generano proteine di fusione si verificano anche nei tumori solidi. Questi studi sono stati focalizzati sui sarcomi in quanto, in questi tumori, le anomalie cito genetiche sono ben caratterizzate. La prima di queste fusioni geniche riguarda il gene RET nel carcinoma papillare tiroideo (PTC, Papillary Thyroid Carcinoma). Il dominio tirosinachinasi del gene RET è fuso con uno di tre geni differenti la cui funzione è ancora sconosciuta o scarsamente caratterizzata. Nel carcinoma papillare tiroideo sono state trovate tutte e tre le fusioni di RET. Il gene del sarcoma di Ewing (EWS) può fondersi con uno di quattro partner di traslocazione tra cui WTI, il gene soppressore tumorale responsabile di alcuni casi di tumore di Wilms. A differenza delle traslocazioni di RET, ciascuna delle quattro traslocazioni cui EWS può andare incontro è stata osservata in un tipo diverso di tumore (tab. 7). Non è ancora chiaro se ciascuna traslocazione di EWS sia specifica di un tipo particolare di tumore.
Progressione maligna di cellule premaligne
Il modello di evoluzione clonale
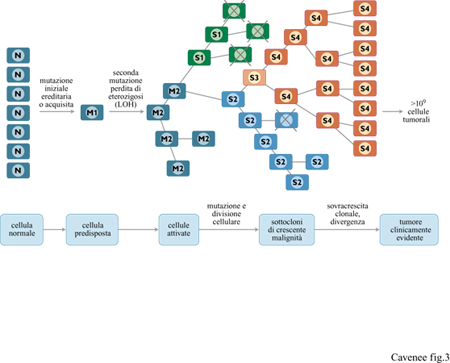
Finora abbiamo parlato di mutazioni singole nei tumori. È chiaro, tuttavia, che lo sviluppo del cancro richiede molte tappe: il processo attraverso il quale da una singola cellula normale si forma un tumore clinicamente evidente è detto evoluzione clonale ed è riassunto in figura (fig. 3). In questo modello genetico (Nowell, 1976) l'inizio del cancro ha luogo a partire da una mutazione ereditaria o somatica in una singola cellula ed è seguito dalla LOH, in accordo con l'ipotesi dei due eventi, formulata da A.G. Knudson. In questo modello, le prime cellule tumorali sono molto probabilmente normali secondo criteri morfologici e biochimici, anche se hanno acquisito un vantaggio proliferativo rispetto alle cellule adiacenti normali. Il clone tumorale iniziale continua a dividersi producendo cellule tumorali geneticamente identiche. Occasionalmente, la divisione cellulare sarebbe accompagnata da una mutazione addizionale; molte di queste mutazioni sarebbero letali, mentre altre non conferirebbero alcun vantaggio selettivo. l rari casi in cui vi è un vantaggio positivo nella crescita rappresentano le basi molecolari attraverso cui le cellule tumorali evolvono verso un aumentato carattere maligno. Il numero di queste mutazioni vantaggiose, inizialmente rare, aumenta con la divisione cellulare, talora abbastanza rapidamente, in concomitanza con un riscontro clinico di un passaggio netto da una crescita benigna a un tumore a carattere fortemente maligno. Nelle popolazioni umane, l'accumulo dei difetti genetici è tempo-dipendente, in accordo con l'aumento del cancro con l'età.
Le alterazioni genetiche che conferiscono un vantaggio di crescita possono essere di diversa natura. Le mutazioni possono modificare molecole che regolano la crescita normale (per esempio geni soppressori tumorali), permettere la crescita di cellule tumorali in modo indipendente dagli ormoni o indurre, mediante effetti locali, dei cambiamenti nella crescita circoscritti. Cellule con alterazioni genetiche o epigenetiche che conferiscono resistenza a sostanze chimiche o alle radiazioni potrebbero anche essere favorite dalle pressioni selettive che provengono dalle terapie anticancro. In uno stadio qualsiasi della progressione del tumore, differenze nell'ambiente locale possono stimolare l'insorgenza di subcloni varianti, il più 'adatto' dei quali supererà alla fine gli altri, divenendo la popolazione dominante del tumore in evoluzione. Si può anche verificare una divergenza di più subcloni lungo differenti vie di progressione, contribuendo in tal modo all'eterogeneità genetica osservata in alcuni tumori. Perciò, l'evoluzione clonale delle popolazioni di cellule tumorali è un processo a più tappe, trainato dall' acquisizione continua e progressiva di vantaggi genetici di crescita e di modificazioni epigenetiche in un tipo cellulare che una volta possedeva un genotipo e un fenotipo normali.
Evidenze a favore del modello
L'origine monoclonale della maggioranza dei tumori umani è stata verificata con differenti marcatori genetici o epigenetici. Nelle neoplasie ereditarie il tumore origina da una cellula eterozigote predisposta in cui si verifica una seconda mutazione nella restante copia normale del gene. Con pochissime eccezioni (gene RET) tutte le cellule dei tumori ereditari sono omozigoti per il gene soppressore tumorale mutante e dunque devono derivare da un'unica o al massimo da poche cellule. La dimostrazione dell'origine unicellulare della maggior parte dei tumori sporadici è stata più ardua, perché non si conosceva quali fossero i difetti iniziali, e neanche se essi fossero genetici o epigenetici (o entrambe le possibilità).
Mediante analisi citogenetiche, inizialmente è stato dimostrato che nella leucemia mieloide cronica (CML, Chronic Myelogenous Leukemia) il 100% delle cellule tumorali presentava la stessa specifica anomalia dei cromosomi 9 e 22 e ciò indicava che questa alterazione genetica era avvenuta nella cellula tumorale iniziale. l geni interessati, BCR e ABL, sono stati isolati, rispettivamente sul cromosoma 9 e sul cromosoma 22, e la presenza della mutazione BCR-ABL è stata dimostrata in tutte le cellule, a riprova di un'origine unicellulare della CML a livello genico. Studi sulle modalità di inattivazione del cromosoma X in cellule normali e affette da CML dello stesso paziente hanno fornito ulteriori prove dell'origine clonale o unicellulare della CML. Nelle cellule delle femmine, uno dei due cromosomi X viene inattivato in maniera casuale ed ereditaria. Mentre i tessuti normali sono costituiti da un mosaico di cellule, il tessuto tumorale, se di origine clonale, dovrebbe avere lo stesso cromosoma X inattivato in tutte le cellule. L'espressione dei geni polimorfici del cromosoma X, come quello per la glucosio-6-fosfatodeidrogenasi (G6PD), permette di verificare queste possibilità. Le cellule CML di individui polimorfici esprimono solo un tipo di G6PD, indicando che tutte le cellule CML devono essersi originate per proliferazione di una singola cellula. L'impiego di questo tipo di indagini sull'origine clonale delle cellule tumorali, sfruttando l'inattivazione del cromosoma X, ha permesso di estendere queste conclusioni alla maggioranza dei tumori sporadici comuni.
Con l'analisi di campioni tumorali ottenuti sequenzialmente dallo stesso individuo, o di campioni ottenuti sincronicamente, contenenti componenti distinguibili di differenti stadi della progressione, è stata dimostrata la parallela progressione clinica e genetica dei tumori umani, ipotizzata nel modello di evoluzione clonale. Tumori che necessitano di rimozione chirurgica, ma che in seguito si ripresentano in una forma ancor più maligna nello stesso sito, come i tumori cerebrali, permettono l'analisi sequenziale delle alterazioni genetiche. Coppie di tumori cerebrali dello stesso paziente, uno a basso e uno ad alto grado di malignità, sono stati esaminati per quanto riguarda la mutazione nel gene soppressore tumorale p53, in modo da permettere una valutazione quantitativa dell'allele mutante rispetto a quello normale in ciascun tumore (Sidransky et al., 1992). I tumori a bassa malignità avevano solo poche cellule con mutazioni specifiche del gene p53, mentre i tumori altamente maligni erano composti quasi esclusivamente di cellule contenenti p53 mutante. La mutazione specifica (un cambiamento di un singolo nucleotide) era la stessa in entrambi i tumori di ciascuna coppia; ciò ha portato alla conclusione che una rara cellula mutante, nel tumore a bassa malignità, era andata incontro a un'espansione clonale tale da costituire il tipo cellulare predominante nel tumore altamente maligno. Un singolo tumore cerebrale, che presentava componenti sia a basso sia ad alto grado di malignità, è stato dissezionato e analizzato in maniera simile. Nella porzione a bassa malignità, il 60% delle cellule conteneva un gene p53 mutante e uno normale e il 40% delle cellule aveva due differenti geni p53 mutanti, mentre la componente ad alto grado di malignità era composta interamente da cellule con ambedue gli alleli mutanti. Perciò, la progressione clinica e istologica dei tumori cerebrali era associata all'espansione clonale di cellule che avevano precedentemente acquisito una mutazione nel gene p53 che aveva a sua volta fornito loro un vantaggio selettivo nella crescita.
L'accumulo sequenziale di difetti genetici è stato dimostrato anche in diversi altri studi. Per esempio, con l'analisi cito genetica di un neurofibrosarcoma prelevato tre volte nell' arco di cinque mesi sono state osservate mutazioni cumulative. Il primo campione, ottenuto da biopsia con ago aspirato, presentava un i(1)q10 in sovrannumero, come unica anomalia cito genetica; alcune cellule avevano perduto anche il cromosoma 18. Tre settimane dopo, un secondo campione, ottenuto per resezione chirurgica, mostrava un cariotipo predominante con i(1)q10, monosomia del cromosoma 18 e trisomia del cromosoma 21. Dopo 5 mesi è stato asportato un grosso tumore ricorrente nel cui cariotipo complesso erano presenti alcune, ma non tutte, le anomalie trovate nei primi due campioni, insieme a diverse nuove aberrazioni. La divergenza genetica e l'alto grado di anomalie citogenetiche riscontrate durante l'evoluzione di questo tumore si osservano anche in molti altri tipi di neoplasia e sono particolarmente evidenti nei tumori microdissezionati che mostrano instabilità genomica.
Per studiare i vari aspetti del modello di evoluzione clonaIe proposto, quando per motivi clinici era difficile disporre di campioni sequenziali, sono stati utilizzati anche campioni ottenuti contemporaneamente dallo stesso tumore, contenenti componenti distinte con differenti gradi di malignità. Il dubbio che campioni sincroni appartengano a un continuum di progressione maligna è stato chiarito, per quanto riguarda diversi tipi di tumore, come quelli della mammella, del polmone, del colon e dell 'uvea, attraverso la dimostrazione di alterazioni genetiche specifiche condivise dai vari campioni. Per esempio, in una metaplasia squamosa (un'anomalia non tumorale dei bronchi) adiacente, anche se fisicamente separata, a un carcinoma a cellule squamose, era presente una piccola frazione di cellule contenenti una specifica mutazione di p53 identica alla mutazione che si osserva nelle cellule del carcinoma: ciò dimostra, chiaramente, una discendenza nella progressione maligna. Analogamente, la progressione del carcinoma del dotto in situ (DClS, Ductal Carcinoma In Situ) a cancro della mammella, infiltrato e metastatico, è stata dimostrata identificando sette regioni cromosomiche che, in entrambi i tipi tumorali, presentavano LOH. Tuttavia, in questi stessi individui, foci di DClS adiacenti mostravano un tipo diverso di perdita allelica negli stessi loci cromosomici, indicando una divergenza genetica durante l'evoluzione clonale di DClS a tumore invasivo della mammella. In vari pazienti con cancro del colon sono stati osservati contemporaneamente adenoma benigno di grado elevato e carcinoma maligno ed è stata usata la tecnica di tipizzazione allelica per dimostrare l'avvenuta LOH in siti cromosomici vicini o all'interno di tre geni soppressori tumorali, il gene APC sul cromosoma 5q, il gene DCC sul18q e il gene p53 sul17p (Boland et al., 1995). L'espansione clonale di cellule con LOH nel cromosoma 5q avviene precocemente negli adenomi benigni e quella di cellule con LOH nel cromosoma l7q ha luogo durante la transizione da grandi adenomi a carcinoma. La displasia grave, che precede il carcinoma, è caratterizzata da un alto grado di eterogeneità genetica, indicando una divergenza della progressione tumorale a questo stadio. L'LOH del l8q non è associata in maniera preferenziale ad alcuno degli stadi patologici.
Un esempio recente di melanoma dell'uvea con una componente pigmentata e una non pigmentata illustra molto bene il parallelismo tra progressione genetica e istologica. Le due componenti sono facilmente separabili e patologicamente distinte, con la porzione pigmentata composta di cellule epitelioidi di forma regolare e quella non pigmentata contenente un insieme più eterogeneo di "grandi cellule epitelioidi e molte forme bizzarre e multinucleate". L'analisi cito genetica ha evidenziato cinque anomalie nelle cellule pigmentate e almeno undici nelle cellule tumorali non pigmentate. Tre delle undici alterazioni sono comuni a entrambe le componenti; una quarta anomalia, la monosomia del cromosoma 3, è stata trovata solo nelle cellule pigmentate con l'analisi citogenetica, ma l'analisi genetica a livello molecolare ha evidenziato LOH nel cromosoma 3 delle cellule non pigmentate. Ciò indica che la disomia del cromosoma 3 nelle cellule non pigmentate è derivata da monosomia seguita da duplicazione. Sia l'istopatologia sia l'analisi genetica hanno suggerito decisamente che le cellule tumorali non pigmentate pleomorfiche derivano dall'espansione clonale e dalla successiva divergenza di cellule pigmentate omogenee.
È interessante notare che condizioni descritte come 'non tumorali', come la metaplasia squamosa del polmone, l'adenoma benigno del colon, la cheratosi actinica della pelle e la presenza di cellule anomale della mucosa nel cancro della testa e del collo, contengano una frazione di cellule con mutazioni genetiche. È possibile che si tratti, in realtà, di cellule tumorali a uno stadio molto precoce dell'evoluzione del tumore. Queste osservazioni sono di importanza decisiva, poiché si presenta la possibilità di utilizzare marcatori genetici per identificare cellule potenzialmente maligne a uno stadio della loro progressione molto più precoce di quanto non fosse possibile in precedenza.
Esempi di evoluzione clonale: cooperazione di geni soppressori tumorali e oncogeni
Una comprensione accurata ed esauriente dell'eziologia genetica del cancro umano necessita chiaramente dell'integrazione di una serie di anomalie che coinvolgono sia gli oncogeni sia i geni soppressori tumorali. Per una varietà di tumori, tra cui quelli del cervello, del colon, del polmone, della cervice uterina, della mammella e della pro stata, si sta tentando di individuare questo tipo di progressione genetica integrata. Lo studio approfondito della progressione istopatologica dei tumori cerebrali e del colon ha fornito una base per classificare queste anomalie genetiche ed è servito da prototipo per analisi analoghe in altri tipi di tumori. Questi due tipi di neoplasia sono paradigmatici anche per quanto riguarda l'analisi della progressione tumorale che si ottiene con campioni sequenziali nel caso del tumore del cervello e per l'analisi della progressione tumorale, limitata necessariamente a componenti dissezionabili, di campioni ottenuti sincronicamente nel caso del tumore del colon.
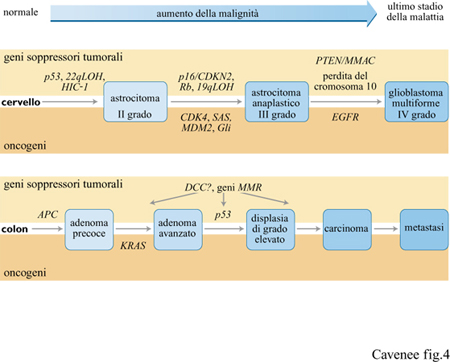
Nella progressione maligna dei gliomi, la forma più comune di tumore cerebrale primario, sono stati distinti tre stadi istopatologici (fig. 4), accompagnati parallelamente da tre serie cumulative di alterazioni genetiche specifiche (Sidebottom et al., 1995). Dall'esame di tumori primari e ricorrenti dello stesso paziente e di tumori di vario grado di malignità di individui differenti, si evidenzia un accumulo specifico e ordinato di alterazioni di geni soppressori tumorali e di oncogeni. Le alterazioni genetiche che si verificano precocemente durante la progressione maligna, come la mutazione di p53, l'LOH di 22q e la metilazione di HIC- I, sono riscontrabili in tutti gli stadi del glioma (lI-IV), mentre eventi genetici tardivi (per esempio, perdita di pI 6/CDKN2 o amplificazione di CDK4) sono presenti solo negli stadi più maligni (lll, IV) o quasi esclusivamente nella fase finale (stadio IV) della malattia (per esempio, monosomia del cromosoma 10). Ciascuna di queste anomalie si verifica con frequenze alte, ma non sempre, indicando la possibilità di vie alternative che coinvolgano una mutazione o un'altra. Per esempio, la mutazione di p53 si verifica approssimativamente nel 30% di tutti i gliomi con gravità di diverso grado (Bögler et al., 1995), l'amplificazione del gene EGFR nel 40% e la monosomia del cromosoma lO nel 60÷95% dei gliomi di IV grado. A differenza dell'alterazione di un singolo gene, la frequenza di inattivazione di una particolare via regolatrice della crescita, che può verificarsi attraverso tappe multiple, è vicina al 100%. Per esempio, l'abolizione della via di regolazione negativa controllata dal gene Rb è il risultato, in quasi tutti i gliomi molto gravi, di uno solo di vari possibili eventi, tra cui la perdita di Rb, la delezione di p16/CDK4 o l'amplificazione di CDK4 (e meno frequentemente di CDK6). Analogamente, la via di inibizione della crescita cellulare che coinvolge p53 è inattivata, in alcuni gliomi, dalla mutazione di p53 e, in altri, dall' amplificazione/sovraespressione del gene MDM2 e della proteina, da esso codificata, che si lega e inattiva il dominio di attivazione della trascrizione di p53. Perciò, per la progressione di alcuni tumori potrebbe essere necessaria l'inattivazione di una particolare via regolatrice della crescita.
Anche la progressione maligna del tumore del colon coinvolge un accumulo di alterazioni negli oncogeni e nei geni soppressori tumorali (v. figura 4), dimostrata mediante l'analisi di campioni di differenti gradi di malignità ottenuti sincronicamente da singoli individui e dal confronto di tumori di molti pazienti (Fearon, 1995; Boland et al., 1995). Numerosi esempi di identiche mutazioni genetiche negli adenomi (stadi precoci) e nei carcinomi (stadi avanzati) dello stesso paziente con cancro del colon avvalorano l'uso di campioni ottenuti sincronicamente nello studio della progressione delle neoplasie maligne. Tre di queste alterazioni genetiche si manifestano con alta frequenza e con una certa specificità di stadio: la mutazione o la LOH del gene soppressore tumorale APC nel 50% degli adenomi precoci, la mutazione punti forme del codone 12 o 13 dell'oncogene KRAS nel 50% dei casi di adenomi di grandezza inferiore a 1 cm e la LOH del gene p53 nel 75% dei carcinomi del colon. È anche frequente l'inattivazione del gene DCC, ma non è un evento stadio-specifico.
Nella progressione genetica sia dei tumori del colon sia del cervello, e della maggior parte degli altri tumori studiati finora, le alterazioni più precoci riscontrabili riguardano geni soppressori tumorali, mentre il passaggio a stadi con carattere più maligno è dovuto all'effetto combinato di attivazione degli oncogeni e di inattivazione dei geni soppressori tumorali.
Effetti delle mutazioni sul fenotipo
La base genetica del cancro è dimostrata dalla consistente associazione tra mutazione, ereditata o acquisita, di geni soppressori tumorali o di oncogeni, con forme o sindromi cancerose specifiche. Nel caso delle sindromi da cancro, un singolo gene mutante può predisporre gli individui a più tipi di tumore, ma l'analisi clinica nei confronti di più generazioni di famiglie numerose indica che, mentre alcuni tipi di tumore possono essere condivisi dalle famiglie più colpite, la manifestazione di altri varia da una famiglia all'altra. La ricerca sull'origine di questa variabilità, attraverso analisi dettagliate delle mutazioni in diversi di questi geni, ha rivelato importanti correlazioni tra tipi particolari di tumore, osservati in ciascuna famiglia, e tipo e localizzazione della mutazione, all'interno del gene responsabile. Descriveremo ora due esempi di queste correlazioni, le mutazioni del gene RET con la neoplasia endocrina multipla di tipo 2 (MEN2) e le mutazioni del gene VHL con il morbo di von Hippel-Lindau.
MEN2 si manifesta con tre varianti clinicamente definite, MEN2A, MEN2B e FMTC, che hanno in comune la predisposizione verso tumori delle cellule 'C' della tiroide (carcinomi midollari tiroidei o MTC) (Mak e Ponder, 1996). Queste sindromi differiscono in quanto MTC è l'unico tumore tipicamente presente nelle famiglie FMTC, mentre sia i pazienti MEN2A sia quelli MEN2B possono sviluppare tumori della ghiandola surrenale (feocromocitomi) e solo i pazienti con MEN2A sviluppano iperplasia della paratiroide. Uno spettro molto ristretto di mutazioni nel gene RET causa MEN2A, MEN2B e FMTC e le mutazioni sono specifiche per ciascuna di queste sindromi. In MEN2A 1'87% delle mutazioni avviene a livello di un singolo residuo di cisteina nel codone 634 e il 93% in una delle cinque cisteine del dominio extracellulare di RET. Inoltre, la manifestazione dell'iperplasia della paratiroide, specifica per MEN2A, è associata a una mutazione da cisteina ad arginina nel codone 918, in corrispondenza del sito del dominio tirosinachinasico di RET, deputato al riconoscimento del substrato. Molte delle mutazioni RET in FMTC sono simili a quelle in MEN2A (5 cisteine) ma sono distribuite più uniformemente. Sono state osservate anche alcune famiglie con mutazioni apparentemente specifiche di FMTC nei codoni 768 e 804 di RET.
Sia la localizzazione sia il tipo di mutazioni del gene VHL mostrano forti correlazioni col fenotipo della malattia VHL (Sidebottom et al., 1995). VHL è stata originariamente descritta come una singola entità clinica in cui tutte le famiglie hanno una predisposizione costante agli angiomi della retina e agli emangioblastomi del sistema nervoso centrale, ma anche una predisposizione al carcinoma delle cellule renali (RCC, Renal Cell Carcinoma) e ai feocromocitomi che è variabile tra le famiglie VHL. Analizzando più generazioni di una famiglia numerosa, mutazioni da T a C nel nucleotide 505 erano correlate all'assenza completa di RCC, ma alla presenza del feocromocitoma nel 54% degli individui colpiti. In una seconda famiglia numerosa è stata osservata una relazione inversa, con mutazioni da T a C nel nucleotide 686, in cui il 41 % degli individui colpiti aveva RCC, ma solo l'1% aveva feocromocitoma. Una terza famiglia con una mutazione da C a T nel nucleotide 712 presentava tutti e quattro i possibili tumori, con frequenze comprese tra il 32 e il 65% degli individui colpiti. Considerazioni sul tipo di mutazione (cambiamento di senso o missense, non-senso o non-sense, microdelezione/inserzione, grande delezione) rispetto al fenotipo della malattia hanno permesso di evidenziare l'esistenza di una correlazione tra mutazioni del tipo cambiamento di senso e famiglie VHS che sviluppavano feocromocitoma, cioè il 96% di queste famiglie aveva mutazioni cambiamento di senso nel gene VHL.
Correlazioni tra la localizzazione di mutazioni specifiche in un particolare gene e tipi specifici di tumore sono state dimostrate anche per diversi altri geni, compresi p53 nei tumori sporadici e APC nei tumori ereditari del colon (Fearon, 1995). Questi dati, insieme allo studio dei geni VHL e RET, hanno stabilito un legame inscindibile tra fenotipo e difetti genetici.
Contrariamente agli studi descritti in precedenza, mutazioni identiche, trovate all'interno di famiglie e tra famiglie, potrebbero derivare da una singola mutazione avvenuta in un progenitore comune, potenzialmente distante (effetto del fondatore), piuttosto che da mutazioni indipendenti ma identiche. La presenza di tali mutazioni fondatrici ancestrali, all'interno di una popolazione umana di comune origine etnica o geografica, è stata descritta per MLHl e MSH2, geni di riparazione del DNA, in diverse famiglie HNPCC (Hereditary non polyposis colon cancer) e per il gene BRCA1, nel caso del tumore delle ovaie di tipo familiare. L'identificazione delle mutazioni comuni che derivano da un effetto del fondatore o da eventi indipendenti è di immenso valore poiché permette una determinazione rapida e tecnicamente semplice dei rischi di cancro prima che se ne manifestino i sintomi.
Applicazioni pratiche della genetica del cancro
L'identificazione degli oncogeni e dei geni soppressori tumorali coinvolti nei tumori sporadici ed ereditari ha stimolato lo sviluppo di un approccio molecolare alla diagnosi, alla progno si e al trattamento del cancro. Attualmente, il principale vantaggio apportato da queste ricerche al paziente affetto da cancro consiste nelle possibilità di previsione e diagnosi che, in alcuni casi, hanno portato a strategie d'intervento più razionali e tempestive. È anche possibile che una diagnosi più precoce del cancro, tramite l'analisi molecolare, possa portare, per alcuni tipi di tumore, a una più alta frequenza di successo nella cura con i trattamenti attualmente disponibili.
La predisposizione alla maggior parte dei tumori ereditari discussi in questo saggio può essere determinata grazie all'analisi genetica di individui presintomatici. Attualmente, queste analisi sono di routine negli ospedali e per alcuni tipi di tumore sono tecnicamente piuttosto semplici ed economiche. Per esempio il 90% degli individui FAP che ereditano un gene mutante APC può essere identificato attraverso uno dei due test molecolari effettuati sugli acidi nucleici in un piccolo campione di sangue (Powell et al., 1993). Questi test identificano le proteine APC mutanti o troncate con una tecnica di traduzione in vitro (saggio di traduzione della proteina), oppure individuano riduzioni alleliche endogene della trascrizione del gene APC. In molti casi l'identificazione iniziale di una mutazione APC in un individuo affetto permette poi un test definitivo per altri membri non sintomatici della stessa famiglia. Ciò risulta particolarmente utile quando una prima mutazione fondatrice è rappresentata in più generazioni di famiglie numerose, come è stato descritto recentemente per due famiglie HNPCC con mutazioni nel gene MSH2 o nel gene MLH1, e che risultavano identiche in tutti i membri affetti. Una volta identificate, queste mutazioni erano entrambe rilevabili con una singola reazione PCR. L'analisi presintomatica per le famiglie FAP e HNPCC permette una sorveglianza colonoscopica focalizzata su quegli individui che ereditano il gene mutante, aumentando in tal modo la probabilità di una diagnosi precoce e di una progno si più accurata. D'altra parte, per quanto riguarda il tumore del colon, questo tipo di esame minimizza la necessità di ripetere indagini invasive in individui non portatori e riduce l'ansia di sviluppare un cancro. Simili procedure genetiche, tecnicamente semplici, si possono effettuare per quei tumori ereditari con uno spettro di mutazioni molto ristretto, come il morbo di von Hippel-Landau e la neoplasia endocrina di tipo 2. Monitorare le mutazioni di altri geni responsabili di tumori ereditari, come il gene Rb nel retinoblastoma o il gene BRCAl nel tumore della mammella familiare, è assai più difficile perché i geni sono molto grandi e le mutazioni sono distribuite in parti estese del gene piuttosto che raggruppate o situate in punti caldi. L'analisi di sequenza del DNA, effettuata con le attuali metodologie, è insufficiente in questi casi, ma lo sviluppo di batterie di oligonucleotidi o di 'DNA chips' per un'analisi di sequenza rapida basata sull'ibridazione potrebbe alla fine portare a superare questa difficoltà.
Sebbene non si siano ancora affermati nella pratica medica di routine, sono in corso di sperimentazione alcuni metodi non invasivi per la diagnosi precoce di alcuni tumori sporadici. Due di questi metodi misurano le alterazioni di p53, che attualmente rappresentano l'alterazione genetica prevalente in tutti i tipi di tumori, come marcatori diretti o indiretti di cellule cancerose. Il primo metodo, il clonaggio fagico dei prodotti PCR di p53 (Sidransky, 1995), implica l'analisi diretta del gene p53 mediante amplificazione PCR di piccolissime quantità di DNA proveniente da cellule dei fluidi corporei. Un 'repertorio' (library) di prodotti PCR del gene p53 viene allestito in un batteriofago e poi selezionato per la presenza di geni p53 mutanti, mediante ibridazione con sonde oligonucleotidiche di tipo selvatico e mutante. Questo metodo può identificare un singolo gene mutante p53 su un fondo di 10.000 cellule normali. Cellule mutanti nel gene p53 sono state rilevate nell'urina di pazienti con tumore della vescica, in campioni di feci di pazienti con tumore del colon, nella saliva di pazienti con tumore della testa e del collo e nell' espettorato di pazienti con tumore al polmone. Di fatto, un anno prima della diagnosi clinica di cancro polmonare, in campioni di espettorato sono state trovate cellule mutanti nel gene p53, dimostrando il potenziale beneficio clinico di questo tipo di approccio molecolare per una diagnosi precoce del cancro.
Il secondo, indiretto approccio rileva la presenza di anticorpi p53 con il saggio ELlSA (Enzyme Linked Immuno Sorbent Assay, saggio basato sull'assorbimento del complesso immunitario a cui è legato un enzima), metodo in grado di rilevare anticorpi specifici o antigeni in un campione biologico (Lubin et al., 1995). Gli anticorpi p53 non sono normalmente presenti nel siero ma, in seguito a una mutazione, la proteina p53 può divenire più stabile e accumularsi nelle cellule tumorali causando, in una piccola percentuale di casi, la produzione di auto-anticorpi p53. Il valore di questo test diagnostico, potenzialmente routinario, è stato dimostrato dal ritrovamento di anticorpip53 (e dunque alterazione di p53) nel siero di due forti fumatori, diversi mesi prima della diagnosi di tumore polmonare. È importante sottolineare che il livello di anticorpi diminuiva parallelamente alla risposta al trattamento clinico e radiologico. In un altro caso, la remissione della malattia in seguito al trattamento era associata a un concomitante ritorno di aumentati livelli di anticorpi p53, indicando che questo saggio può essere utile per un monitoraggio continuo della risposta al trattamento da parte del paziente. Un recente studio multicentrico ha convalidato questa idea, mostrando una correlazione tra risposta del paziente, presenza e livello di anticorpi p53 (Lubin et al., 1995). Si comincia a utilizzare questo tipo di indagine anche per i tumori della mammella e del pancreas. L'adattamento dei metodi ELlSA e di clonaggio fagico, per il rilevamento di altre mutazioni genetiche, potrebbe significativamente ampliare la loro utilità.
Attualmente, sono in corso tentativi di correlare la sopravvivenza del paziente e la risposta al trattamento con la molteplicità di alterazioni genetiche riproducibili e specifiche nei tumori sporadici: questo potrebbe portare a una migliore stratificazione del campione dei pazienti (cioè a una migliore rappresentatività della popolazione del campione stesso) e a una più appropriata applicazione delle terapie. Per esempio, l'amplificazione del gene EGFR nei tumori al cervello è associata a un minore tempo di ricaduta. Questi studi non hanno ancora considerato la possibilità di un'ulteriore stratificazione su base genetica nel 20% dei tumori al cervello con amplificazione e riarrangiamento di EGFR, che nei sistemi sperimentali causa un marcato aumento del potere tumorigeno. Similmente sono state anche stabilite correlazioni dirette e quantitative tra alterazioni dell' espressione della ciclina E e l'aggressività del tumore della mammella. L'uso clinico di queste osservazioni genetiche è attualmente limitato a un ruolo ausiliario dell'istologia tumorale. Dunque, un obiettivo dell'analisi genetica dei tumori sporadici è quello di identificare e trattare più aggressivamente quei pazienti che ci si attende rispondano a terapie specifiche e sottoporre a trattamenti diversi quei pazienti che, potenzialmente, non rispondono.
Conclusioni
La soppressione della crescita di cellule tumorali maligne è stata realizzata in sistemi sperimentali mediante manipolazione genetica, per esempio tramite la sostituzione di geni soppressori tumorali, l'inibizione di oncogeni attivati, l'interferenza con processi di angiogenesi e metastasi e l'aumento dell'immunogenicità delle cellule tumorali. Per quanto riguarda la terapia genetica del cancro umano, essa deve superare diversi ostacoli (per esempio, deve essere identificato un metodo efficace e selettivo per l'introduzione di geni nelle cellule tumorali) prima che possa essere mantenuta la promessa di un miglioramento terapeutico mediante la medicina molecolare. l progressi recenti nello sviluppo di metodi per indirizzare molecole e cellule verso organi specifici, mediante peptidi selezionati in vivo, indicano che questi ostacoli saranno superati nel prossimo futuro (Pasqualini e Ruoslahti, 1996).
Ringraziamenti
Desideriamo ringraziare Frances Putkey per l'assistenza nella preparazione delle tabelle, Oliver Bögler e Frank B. Furnari per la revisione critica del manoscritto e Karen Arden per le utili discussioni. Joseph F. Costello è finanziato con un training grant (5T32CA09290) del National Institute of Health.
Bibliografia citata
BEROUD, C., VERDIER, F., SOUSSI, T. (1996) p53 gene mutation: software and database. Nucleic Acids Res., 24, 147-150.
BISHOP, lM. (1995) Cancer: the rise of the genetic paradigm. Genes and Dev., 9, 1309-1315.
BOGLER, O., HUANG, H-J. S., KLEIHUES, P., CAVENEE, W.K. (1995) The p53 gene and its role in human brain tumors. GUa, 15, 308-327.
BOLAND, C.R., SATO, J. APPELMAN, H.D., BRESALIER, R.S. , FEINBERG, AP. (1995) Microallelotyping defines the sequence and tempo of allelic losses at tumour suppressor gene loci during colorectal cancer progression. Nature Med., 1,902-9.
BOS, J.L. (1995) p2lras: an oncoprotein functioning in growth factor-induced signal transduction. Eur. J. Cancer, 31A, 1051-1054.
CAVENEE, W.K., DRYJA, T.P., PHILLIPS, R.A., BENEDICT, W.F., GODBOUT, R., GALLIE, B.L., MURPHREE, A.L., STRONG, L.C., WHITE, R.L. (1983) Expression of recessive alleles by chromosomal mechanisms in retinoblastoma. Nature, 305, 779-784.
FEARON, E.R. (1995) Molecular genetics of colorectal cancer. Annals of N.Y. Acad. of Sci., 768, 101-10.
FEARON, E.R., VOGELSTEIN, B. (1990) A genetic model for colorectal tumorigenesis. CelI, 61, 759-767.
FIDANZA, V., MELOTTI, P., YANO, T., NAKAMURA, T., BRADLEY, A., CANAANI, E., CALABVETTO, B., CROCE, C.M. (1996) Double knock-out of the ALL-I gene blocks hematopoietic differentiation in vitro. Cancer Res., 56, 1179-1183.
HOLLSTEIN, M., SHOMER, B., GREENBLATT, M., SOUSSI, T., HOVIG, E., MONTESANO, R., HARRIS, C.C. (1996) Somatic point mutations in the p53 gene of human tumors and cell lines. Nucleic Acids Res., 24, 141-6.
HUNTER, T., PINES, J. (1994) Cyclins and cancer II: cyclin D and CDK inhibitors come of age. CelI, 79, 573-582.
JONES, P.A. (1996) DNA methylation errors and cancer. Cancer Res., 56, 2463-2467.
KNUDSON, A.G. (1971) Mutation and cancer: statistical study of retinoblastoma. Proc. Natl. Acad. Sci. USA, 68, 820-823.
KNUDSON, A.G. (1993) Antioncogenes and human cancer. Proc. Natl. Acad. Sci. USA, 90, 10914-10921.
LOHMANN, D.R., BRANDT, B., HOPPING, W.E.P., HORSTHEMKE, B. (1996) The spectrum of RB I germ-line mutations in hereditary retinoblastoma. Am. J. Rum. Genet., 58, 940-949.
LUBIN, R., et al. (1995) Serum p53 antibodies as early markers of lung cancer. Nature Med., 1, 701-702.
MAK, Y.F., PONDER, B.A.J. (1996) RET oncogene. Curr. Op. in Genet. Dev., 6, 82-86.
MULVIHILL, J.J. (1977) Genetic repertory of human neoplasia. Genetics of human cancer, a c. di Mulvihill J.J., Miller R.W., Fraumeni ll, New York, Raven, pp. 137-143.
NOWELL, P.C. (1976) The clonal evolution of tumor cell populations. Science, 194, 23-28.
PASQUALINI, R., RUOSLAHTI, E. (1996) Organ targeting in vivo using phage display peptide libraries. Nature, 380, 364-366.
POLLOCK, P.M., PEARSON, lY., HAYWARD, N.K. (1996) Compilation of somatic mutations of the CDKN2 gene in human cancers: non-random distribution of base substitutions. Genes, Chromosomes and Cancer, 15,2, 77-88.
POWELL, S.M., PETERSEN, G.M., KRUSH, A.J., BOOKER, S., JEN, J., GIARDIELLO, F.M., HAMILTON, S.R., VOGELSTEIN, B., KINZLER, K.W. (1993) Molecular diagnosis of familial adenomatous polyposis. N. Engl. J. Med., 329, 1982-7.
RABBITTS, T.H. (1994) Chromosomal translocations in human cancer. Nature, 372, 143-9.
RODRIGUEZ, E., SREEKANTAIAH, C., CHAGANTI, R.S. (1994) Genetic changes in epithelial solid neoplasia. Cancer Res., 54, 3398-3406.
SIDEBOTTOM, E., PONDER, B.A.J., CAVENEE, W.K., SOLOMON, E. a c. di (1995) Genetics and cancer: a second look, Plainview, New York, Cold Spring Harbor Laboratory Press, pp. 219-232.
SIDRANSKY, D. (1995) Screening for clonal genetic alterations. Eur. J. Cancer, 31A, 1127-9.
SIDRANSKY, D., MIKKELSEN, T., SCHWECHEIMER, K., ROSENBLUM, M.L., CAVENEE, W.K., VOGELSTEIN, B. (1992) Clonal expansion of p53 mutant cells is associated with brain tumor progression. Nature, 355, 846-847.
Bibliografia generale
CAIRNS, J. (1975) Mutation selection and the natural history of cancer. Nature, 255, 197-200.
CAVENEE, W.K., WHITE, R. (1995) The genetic basis of cancer. Sci. Amer., 272, 72-79.
VARMUS, H., WEINBERG, R. Genes and the biology of cancer, New York, Scientific American Library, 1993.