La grande scienza. Biotecnologie
La grande scienza. Biotecnologie
Biotecnologie
Le biotecnologie sono generalmente definite come l'utilizzazione progettuale di sistemi biologici per ottenere beni e servizi. I sistemi biologici sono, su scala crescente di complessità, molecole semplici come metaboliti, molecole complesse come proteine e acidi nucleici, complessi sopramolecolari, organuli cellulari, cellule, tessuti, organi e organismi (sia animali sia vegetali). I beni e i servizi sono da intendere nel senso più ampio poiché le biotecnologie sono altamente pervasive, così da influenzare una vasta gamma di settori di applicazione: la biologia, la medicina umana e veterinaria, la zootecnia, l'agricoltura, l'ambiente e l'energia. Questo significa che una determinata tecnologia basata su sistemi biologici, o un insieme di tecnologie (come, per es., quelle del DNA ricombinante), possono rivoluzionare settori diversi comprendenti la salute umana e animale, la conservazione degli alimenti, il controllo degli ecosistemi e la protezione ambientale.
Tradizionalmente, la data di nascita delle biotecnologie innovative viene fissata intorno al 1970, a seguito delle scoperte dell'ingegneria genetica e delle tecnologie avanzate di colture di cellule in vitro. In effetti, le prime permisero di cominciare a trattare il DNA, già da tempo identificato come la struttura biologica responsabile della trasmissione dei caratteri ereditari, alla stregua di una molecola chimicamente e funzionalmente analizzabile e modificabile. Le seconde, invece, fornirono le metodologie di studio ideali per convalidare ipotesi di lavoro non ancora sufficientemente esplorate e anche per ottenere grandi quantità di cellule con caratteristiche predefinite, capaci di produrre quantità illimitate di sostanze chimiche pregiate.
In realtà, la grande rivoluzione biotecnologica consiste nell'avere globalmente impostato le scienze e le tecnologie della vita su basi meccanicistiche. Ciò significa studiare in maniera strategica e il più possibile sistematica tutti i processi biologici in termini molecolari. Da allora, discipline quali la genetica, l'immunologia, la farmacologia, la biologia cellulare e altre hanno superato l'approccio fenomenologico e descrittivo, entrando nel vivo dei meccanismi (relazioni di causa-effetto) che stanno alla base di questi processi. La biochimica e la biologia molecolare, istituzionalmente depositarie delle strategie di studio e delle metodologie utili a decifrare questi meccanismi, hanno identificato le regole necessarie a interpretare a livello deterministico i processi biologici e di conseguenza a ricostruirli in vitro: con ciò si è realizzato il duplice vantaggio di aumentare le nostre conoscenze e di ottenere prodotti utilizzabili per numerose applicazioni.
Caratteristiche delle biotecnologie innovative
Interdisciplinarità e multisettorialità
È abbastanza comune raffigurare le biotecnologie come una clessidra, immagine che in effetti ne fotografa efficacemente due delle principali caratteristiche: l'interdisciplinarità e la multisettorialità. La parte alta della clessidra sta a indicare che numerose discipline contribuiscono allo sviluppo e al perfezionamento di contenuti comuni. La chiave di lettura di questi contenuti, come ricordato, è l'attenzione ai meccanismi di sistemi e processi biologici. I meccanismi vengono esplorati rigorosamente a livello molecolare con il contributo di metodologie mutuate dalle singole discipline che quindi convergono in un continuum (la strozzatura centrale della clessidra). Da questo impasto multi- e interdisciplinare derivano, come raffigurato nella parte bassa della clessidra, gli orientamenti e le applicazioni delle biotecnologie in molteplici settori.
Pertanto, con le biotecnologie si è potuto superare il concetto di una scienza astratta, approdando invece a quello di una scienza completa la cui traiettoria parte da premesse anche esclusivamente teoriche, ma si perfeziona nell'interazione tra più discipline, viene a focalizzarsi in tecnologie ad alto valore aggiunto che poi, con l'intervento quasi sempre determinante delle industrie, sono applicabili alla soluzione di numerosi e diversi problemi. Questa realtà dimostra un importante requisito, quello della pluralità degli operatori in ambito biotecnologico ('le biotecnologie sono di tutti e di nessuno al tempo stesso'): non solo nei termini, mai sufficientemente sottolineati, della necessaria interazione tra esperti di diverse discipline, ma soprattutto nel trasferimento del patrimonio di nuove conoscenze e tecnologie a diversi settori che ne possono trarre vantaggi incalcolabili.
Per queste ragioni lo sviluppo ottimale della ricerca biotecnologica richiede un continuo e serrato dialogo tra gli operatori scientifici (parte alta della clessidra) e gli utenti delle ricerche stesse (parte bassa). Questo non solo perché i prodotti della ricerca devono essere analizzati (controllo di qualità) attraverso una continua verifica sul campo, ma soprattutto perché dall'impiego dei prodotti scaturiscono preziose informazioni su come ottenerli o eventualmente perfezionarli.
Le due anime delle biotecnologie
L'approccio interdisciplinare allo studio delle scienze e tecnologie della vita ha innescato un peculiare circolo virtuoso: la ricerca di base genera alta tecnologia e l'innovazione tecnologica alimenta la ricerca di base. In altri termini, il flusso di informazioni nella clessidra può e deve essere bidirezionale: dall'alto al basso si costruiscono sistemi idonei a generare processi e prodotti utili (beni e servizi). Dalla strozzatura centrale, che rappresenta la convergenza di idee, e anche dalla parte bassa della clessidra (per es., nuova biostrumentazione o nuove metodologie), scaturiscono valide opportunità per affrontare e risolvere i problemi sempre nuovi che nascono durante l'avanzamento della ricerca biologica. Infatti la scienza di base è spesso la prima ad avvantaggiarsi dei propri risultati, grazie all'abbattimento delle barriere tra le singole discipline e alla strategia unificante con cui viene studiato qualsiasi sistema biologico, per esempio la cellula.
Per questi motivi, è ormai abituale sentire parlare di due anime delle biotecnologie. La prima concepisce le biotecnologie come uno strumento vincente per fare ricerca di alto livello. La seconda interpreta le biotecnologie quale mezzo di produzione per ottenere da un lato prodotti già noti con processi ottimizzati, dall'altro prodotti nuovi con performance migliorate rispetto a quelle esistenti. Su questa divergente interpretazione si accendono spesso conflitti accademici, economici e giuridici, e in generale le tendenze oscillano variabilmente, per esempio nelle politiche di sviluppo dei diversi Paesi, privilegiando ora il sostegno alla ricerca libera (purché integrata), ora quello alla ricerca orientata e di sviluppo. Questa stessa variabilità dimostra quanto sia difficile scegliere strategie che si rivelino adeguate nel medio-lungo periodo. La ricerca biotecnologica ha costi estremamente variabili. In un contesto limitato quale quello di un'azienda medio-piccola, la produzione e la commercializzazione di prodotti ben definiti (anticorpi, kit diagnostici, reagenti chimici pregiati, utensili biologici, biosensori) possono comportare costi decisamente bassi, tali da configurarsi come attività di nicchia altamente competitive sui mercati internazionali. In un sistema più ampio come quello di un Paese che intenda dotarsi di strumenti e di risorse scientifiche e umane atte a sostenere ricerca, produzione e occupazione, le biotecnologie assumono necessariamente la connotazione di Big science, molto costosa ma non eliminabile. Si pensi da un lato alla strumentazione analitica necessaria per caratterizzare biomolecole ‒ diffrattometri a raggi X, spettroscopi NMR (nuclear magnetic resonance) ‒, dall'altro a sistemi di produzione industriale su larga scala (bioreattori e loro gestione). Di conseguenza, impostare e sostenere la ricerca richiede strategie lungimiranti e una valutazione molto attenta dei rapporti costi/benefici, prima di operare i relativi interventi.
Analoghe considerazioni valgono per industrie di grandi dimensioni che, pur puntando su segmenti precisi di mercato (per es., vaccini, farmaci modulatori di risposte biologiche, processi polivalenti come immobilizzazione di cellule), devono essere in grado di compiere l'intera traiettoria che va dall'idea brevettabile fino alla commercializzazione di un prodotto/processo. Le recenti e attuali fusioni di multinazionali volte a formare pochi colossi dell'industria delle biotecnologie nel mondo obbediscono a questa logica.
Il paradosso delle biotecnologie: progettualità/imprevedibilità
Il concetto di progettualità permea tutto il sistema delle biotecnologie, dal comparto dell'accademia a quello della produzione. Progettualità significa, infatti, pianificazione di un'attività sperimentale di grandi e piccole dimensioni, dalla costruzione di un programma di ricerca nazionale o internazionale con più operatori al disegno di un singolo progetto sperimentale. Progettualità è, tuttavia, anche quella di un'industria che deve preliminarmente operare scelte, sulle basi congiunte delle esigenze di mercato e delle proprie vocazioni, tracciando un business plan che sia il più ampio ma anche il più preciso possibile, prendendo in considerazione punti critici e opzioni alternative per superarli, prefigurando sviluppi tecnologici e possibili variazioni di mercato, valutando inoltre gruppi accademici consolidati o in fase di organizzazione con i quali interagire, districandosi con attenzione tra la miriade di brevetti e di rivendicazioni di proprietà intellettuali non sempre chiare.
La progettualità è dunque una caratteristica fondamentale del modo di fare ricerca biotecnologica in qualsiasi contesto, da programmi di dimensioni planetarie (per es., il sequenziamento del genoma umano) alle attività produttive di industrie di dimensioni medio-piccole. Con l'avvento delle nuove biotecnologie si è infatti superato storicamente il metodo empirico di una sperimentazione impostata sul principio di prova ed errore, ricorrendo invece a una programmazione precisa e rigorosa.
In realtà, le biotecnologie ci hanno ormai abituato a rapidi mutamenti di scenari a causa di un progresso discontinuo, 'a salti', della ricerca e delle relative tecnologie. Gli esempi al riguardo sono numerosi e riflettono in massima parte intuizioni impreviste, spesso di singoli scienziati o di piccoli gruppi di ricercatori. All'inizio dell'era delle nuove biotecnologie, nei primi anni Settanta, gli enzimi di restrizione erano considerati e studiati come sistemi di difesa dei microrganismi contro l'infezione da virus. Successivamente, Stanley Cohen e i suoi collaboratori (1973) introdussero gli enzimi di restrizione in operazioni sperimentali di 'taglia e cuci' del DNA, rendendo possibile la clonazione genica e l'avvio delle tecnologie di ricombinazione del DNA. Pochi anni dopo, fu una apparentemente semplice riflessione di Yuet Wai Kan e Andrée M. Dozy (1978) a permettere un importante salto di qualità, con l'utilizzazione di questi enzimi ‒ capaci di tagliare il DNA in corrispondenza di specifiche sequenze nucleotidiche ‒ come 'forbici molecolari' selettive per frammentare il DNA umano, sia normale sia mutato, e quindi per delinearne l'analisi strutturale. Nel caso specifico, gli enzimi di restrizione furono utilizzati per ottenere una mappatura del DNA da singoli individui ('mappe di restrizione') e per realizzare un nuovo e risolutivo strumento diagnostico per l'identificazione di malattie genetiche. Tuttavia, oltre all'impiego per la diagnosi di malattie, gli enzimi di restrizione applicati allo studio del genoma permisero anche un formidabile avanzamento della ricerca di base, integrando le metodologie avanzate sviluppate da Allan M. Maxam e Walter Gilbert (1977) e da Frederick Sanger per l'analisi di sequenze polinucleotidiche del DNA.
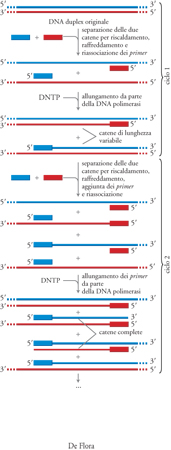
Sempre in questo contesto, un altro esempio ormai classico dello sviluppo della ricerca biotecnologica è la PCR (polymerase chain reaction) ideata da Kary Mullis nel 1983 e presentata nel 1985. Questa tecnologia, frutto combinato di metodologie di biochimica, biologia molecolare, microbiologia e chimica, avviò i procedimenti di amplificazione virtualmente infinita del DNA o di suoi tratti (Tav. I).
La PCR è ormai un paradigma dei vantaggi derivanti dalle biotecnologie. In primo luogo essa permette di produrre, con metodi alla portata di qualsiasi laboratorio, quantitativi elevati del DNA di interesse che diventa pertanto analizzabile in termini chimici e chimico-fisici esattamente come qualsiasi altra biomolecola; per tale motivo la PCR ha rappresentato uno strumento decisivo nel passaggio dall'esplorazione dell'universo delle proteine, più ampiamente analizzato nel trentennio precedente, a quella del DNA e degli acidi nucleici, il cui risultato più completo è il recente sequenziamento dei genomi di diversi organismi vegetali e animali (uomo incluso). L'universalità del DNA ne permette l'indefinita replicazione in vitro indipendentemente dall'organismo da cui esso viene estratto. Di conseguenza, la PCR trova applicazioni in molteplici settori: in medicina, per la soluzione di problemi medico-legali (identificazione di criminali da un capello o da minimi residui biologici), chirurgici (analisi dell'idoneità di donatori di organi da trapiantare in pazienti), genetici (riscontro di mutazioni responsabili di malattie genetiche e degenerative), infettivologici (identificazione di patogeni in fluidi biologici), oncologici (valutazione delle malattie minime residue, importante per il monitoraggio di progressione neoplastica); nel controllo di qualità di alimenti (valutazione della presenza di patogeni in cibi e derivati alimentari); nell'analisi dei suoli, mediante lo studio dei microorganismi ivi residenti; in zootecnia, per lo studio analitico della variabilità genetica di specie da allevamento; in botanica, per lo studio dell'individualità di piante comuni o rare e per l'individuazione di fitopatogeni. Soprattutto, in piena coerenza con il circolo virtuoso precedentemente sottolineato, la PCR è diventata uno strumento ineludibile nello studio dei processi biologici, entrando nell'equipaggiamento obbligato delle tecnologie di clonazione del DNA, degli studi di espressione genica e dei relativi fenomeni di regolazione, e contribuendo di conseguenza a ricollegare il mondo del DNA con quello delle proteine, delle quali ha permesso la produzione in grandi quantità e quindi l'analisi strutturale dettagliata.
Un altro esempio dell'imprevedibilità della ricerca biotecnologica è quello degli studi di Georges Köhler e César Milstein che, negli anni 1973-1975, portarono alla produzione di anticorpi monoclonali partendo da ricerche di immunologia di base. Da queste ricerche scaturì tuttavia uno strumento di lavoro che va ben oltre gli interessi dell'immunologia, poiché gli anticorpi monoclonali costituiscono reagenti molecolari aventi specificità assoluta e predefinita nei confronti di un determinato antigene e possono essere prodotti facilmente in grandi quantità. Da un lato gli anticorpi monoclonali hanno fatto avanzare di decenni la ricerca fondamentale, consentendo l'identificazione, la caratterizzazione e la localizzazione di proteine antigeniche note e non note, quindi la decifrazione di nuove strutture biologiche. In più, rispettando la regola della 'doppia personalità' delle biotecnologie, gli anticorpi monoclonali possono identificare e permettere l'analisi quantitativa non solo di antigeni, e quindi di proteine, utili per l'avanzamento delle conoscenze, ma anche di proteine aventi numerosi altri significati di tipo più propriamente applicativo: quello diagnostico, ormai dominante in medicina, che consente il monitoraggio di malattie genetiche, infettive, degenerative; quello di marcatori della qualità degli alimenti, di cui essi permettono un controllo preciso e affidabile; quello di impalcature per la possibile costruzione di nuovi catalizzatori da utilizzare sia a scopi conoscitivi sia per applicazioni in enzimologia (anticorpi catalitici, potenzialmente in grado di permettere reazioni chimiche inedite e quindi del massimo interesse in biochimica, ma anche nelle tecnologie industriali dei bioreattori e in quelle analitiche dei biosensori).
Le biotecnologie in biologia e medicina
Il settore generale di applicazione più maturo delle biotecnologie, sia nelle conoscenze sia nei risultati, è quello della biomedicina. Settori meno sviluppati, ma potenzialmente migliorabili in futuro dalla ricerca biotecnologica, riguardano l'agroalimentare, l'ambiente e l'energia.
Che la biomedicina abbia finora ottenuto i risultati più importanti e più sensazionali dalle nuove biotecnologie è una logica conseguenza del fatto che l'adozione di strumenti e sistemi biologici produce le ricadute più immediate e dirette proprio su questi sistemi. Per esempio, le strategie e le tecnologie della genetica, integrate dagli approcci meccanicistici derivanti dalla biochimica e dalla biologia molecolare, hanno portato a conoscenze avanzate sui geni (ne è evidente riprova il recente completamento della sequenza del genoma umano). Ciò significa non solo compilarne un catalogo e studiare le proprietà strutturali utili a definirne la fisiologia (espressione), ma anche scoprire mutazioni (silenti o associate a malattie), correlarle con patologie selezionate e aumentare di conseguenza le conoscenze su queste patologie. In aggiunta, il naturale perfezionamento degli approcci sperimentali (circolo virtuoso delle biotecnologie) fornisce strumenti sempre più precisi in termini di reagenti utili sia per acquisire informazioni avanzate sui sistemi biologici (e patologici), sia per sviluppare tecnologie idonee per la soluzione dei diversi problemi biologici e medici.
Il contributo delle biotecnologie al settore biomedico è stato ed è evidente nelle tre principali ramificazioni della medicina: la diagnosi, la prevenzione e la terapia.
Diagnosi e prevenzione
Probabilmente questo è il sottosettore nel quale sono stati ottenuti i risultati più concreti grazie alle seguenti tappe: la costruzione di reagenti biologici pregiati come i già ricordati anticorpi monoclonali e le sonde oligonucleotidiche; lo sviluppo di sistemi integrati nei quali questi reagenti sono opportunamente utilizzati (per es., biosensori e DNA micro arrays); la definizione di polimorfismi a livello sia di microorganismi patogeni sia dell'assetto genetico dei singoli individui; la recente identificazione di nuovi geni responsabili di malattie; lo studio sempre più accurato (anche perché continuamente assistito da nuovi strumenti biotecnologici) di malattie non solo genetiche, ma anche degenerative e infettive, con il conseguente chiarimento di sindromi apparentemente omogenee sul piano clinico ma in realtà basate su determinanti eziopatogenetici eterogenei. Queste e altre conquiste (per es., quelle della biostrumentazione) hanno portato a un affinamento della diagnosi che sarebbe stato impensabile anche solo pochi anni fa. Ne sono derivate metodologie diagnostico-analitiche utili a effettuare screening di intere popolazioni in tempi brevissimi e con affidabilità assoluta, salvo errori umani (per es., scambio di provette contenenti campioni biologici).
Pertanto il medico può attualmente disporre di nuovi e spesso infallibili strumenti per identificare una malattia e anche per valutarne l'evoluzione (monitoraggio). Poiché l'accertamento di una malattia è la premessa per impostare non soltanto le terapie più opportune, ma anche la prevenzione (isolare un paziente infettivo, evitare l'espressione clinica di un gene mutato, indurre protezione nei confronti di patogeni), le nuove biotecnologie stanno interessando con sempre maggiore successo anche il sottosettore della prevenzione.
Esistono numerosi modi di fare prevenzione, a seconda dell'eziologia delle malattie e della loro modalità di trasmissione e di manifestazione clinica. Le biotecnologie hanno migliorato in maniera decisiva la gestione di quelle malattie, come si è detto sempre più numerose, la cui diagnosi si basa su sistemi e tecnologie affidabili. L'identificazione di malattie molecolari (con accertamento delle mutazioni responsabili di patologie) ha portato in alcuni casi all'eradicazione di esse attraverso strategie di epidemiologia molecolare precedentemente non immaginabili. Ne è un clamoroso esempio l'insieme degli interventi diagnostici e conseguentemente preventivi che hanno portato al completo controllo di un vero e proprio flagello che ha colpito la Sardegna per secoli, quello della β-talassemia (l'anemia mediterranea). Ciò è stato possibile in seguito all'identificazione della stragrande prevalenza in Sardegna di una singola e definita mutazione (che colpisce il codon 39 nella sequenza del gene per la β-globina) nei portatori di questa emoglobinopatia quantitativa. Di conseguenza, è stato possibile progettare e costruire sonde oligonucleotidiche specifiche per quella mutazione, con le quali viene effettuata una diagnosi molecolare di β-talassemia sia sugli eterozigoti sia sul feto. Tali reagenti, se applicati quotidianamente sull'intera popolazione, identificano ormai il rischio di malattia su basi oggettive e non solo ricorrendo a indagini anamnestiche o allo studio di marcatori ematologici. L'introduzione di tali strumenti nei consultori genetici ha rappresentato la via maestra per eradicare di fatto la β-talassemia in Sardegna. Naturalmente un successo simile non avrebbe potuto essere conseguito in regioni a elevato polimorfismo genetico (più mutazioni responsabili di diverse sindromi talassemiche), come accade per esempio in Thailandia.
Un caposaldo della prevenzione noto fin dai tempi di Edward Jenner è quello delle vaccinazioni, intese come induzione di una risposta immunitaria (anticorpi neutralizzanti/protettivi) o contro specifici patogeni o contro strutture molecolari determinanti di malattie, per esempio di tumori. L'avanzamento della ricerca ha fornito strumenti altamente migliorativi nel settore delle vaccinazioni, realizzando vaccini ricombinanti, vaccini peptidici ottenuti per via sintetica, vaccini a DNA e vaccini anti-idiotipo.
Poiché la medicina preventiva è destinata nel tempo a prevalere sulla medicina curativa, lo studio dei nuovi vaccini rappresenta uno dei settori di maggior impegno delle biotecnologie. Per questo motivo la vaccinologia dimostra in maniera paradigmatica tutte quelle caratteristiche della ricerca biotecnologica che sono già state menzionate, in particolare: integrazione interdisciplinare; progettualità; circuiti tra ricerca di base, tecnologie abilitanti, conoscenza di malattie. Oltre a una progressiva 'molecolarizzazione' degli strumenti di vaccinazione, si registra da qualche tempo una crescente attenzione al potenziamento dell'attività anticorpopoietica attraverso nuovi adiuvanti e moderne tecnologie suggerite dagli avanzamenti dell'immunologia cellulare: adiuvanti comunemente utilizzati nell'immunizzazione di animali da laboratorio e ovviamente non adatti alla vaccinazione umana sono stati superati da sistemi integrati di citochine, il cui impiego presuppone peraltro un'approfondita conoscenza dei complessi circuiti intercellulari che determinano la risposta immunitaria.
Terapia
Negli ultimi decenni si sono registrate linee di tendenza del tutto innovative nella terapia di numerose malattie, essenzialmente attraverso tre tipologie d'intervento: (a) la produzione biotecnologica di farmaci tradizionali che in precedenza venivano ottenuti per via estrattiva o sintetica; (b) la produzione di farmaci nuovi; (c) le terapie cellulari.
a) Gli esempi più conosciuti di farmaci convenzionali ottenuti per via biotecnologica riguardano proteine ricombinanti dotate di attività farmacoterapeutica. Fino agli anni Ottanta la terapia sostitutiva del diabete insulino-dipendente e delle sue temibili complicanze era basata sulla somministrazione di insuline estratte da altre specie animali (soprattutto suini e bovini), simili ma non identiche all'ormone umano. I problemi legati a questa scelta obbligata riguardavano, da un lato, una limitata sicurezza del prodotto (contaminanti), dall'altro, una progressiva perdita di attività in vivo dovuta a produzione di anticorpi anti-insulina. La combinazione di tecnologie di biologia molecolare e di chimica, che permise l'ottenimento nei primi anni Ottanta dell'insulina umana ricombinante, ha permesso di estendere il trattamento sostitutivo a decine di milioni di diabetici con un prodotto standard di provata sicurezza ed efficacia.
Si possono ormai definire farmaci convenzionali anche altre proteine naturali, una volta note come 'modulatori di risposte biologiche' (BRM), la cui identificazione e caratterizzazione sono andate parallelamente allo sviluppo delle biotecnologie: ormoni (per es., l'ormone della crescita), fattori di crescita e differenziamento cellulare, perlopiù dotati di alta specificità di bersaglio e di alta efficienza (tutti i fattori di espansione delle varie serie emo- e mielopoietiche, come eritropoietina e GM-CSF, granulocyte macrophage colony stimulating factor, che sono risolutivi nel supporto a pazienti trattati con pesanti chemioterapie antineoplastiche e antivirali), citochine, chemochine, neurotrofine. Il concetto di farmaco biotecnologico deriva in questi casi dalla scoperta ‒ o dalla migliore conoscenza ‒ di molecole naturali di comunicazione intercellulare mediante le strategie biotecnologiche, ma anche dalle relative tecnologie di produzione industriale in tutto o in parte coincidenti con le tecnologie del DNA ricombinante.
In realtà rientrano tra i farmaci tradizionali ottenuti per via biotecnologica altre categorie di principî attivi, già noti o inediti (gli antibiotici ne sono un classico esempio), la cui produzione industriale prevede in misura variabile da caso a caso tecnologie nuove che permettono di superare, per esempio, dispendiose e poco efficienti tappe di sintesi chimica. L'impiego di enzimi (identificati ed estratti dalle fonti più idonee, oppure essi stessi clonati ed espressi su scala industriale), la loro immobilizzazione dettata da ragioni di riusabilità e di economicità, il disegno e la costruzione di bioreattori industriali ad alta prestazione (la cui gestione richiede complessi parametri di modellizzazione e di analisi) si stanno diffondendo sempre più negli stabilimenti di produzione industriale. La pervasività delle biotecnologie sta quindi determinando un deciso cambiamento di strategie della chimica farmaceutica.
b) La produzione di farmaci nuovi significa non solo ottenere industrialmente molecole di origine naturale, ma soprattutto creare nuove molecole candidate a rappresentare strumenti terapeutici definitivi. I concetti ispiratori e le strategie dell'attuale ricerca permettono infatti di disegnare su misura molecole inedite, sulla base dell'accurata definizione dei rapporti tra struttura e funzione e anche del preliminare avanzamento di conoscenze, a livello molecolare e meccanicistico, delle malattie da combattere (Tav. II).
Gli esempi al riguardo sono numerosi e confortanti, poiché provengono dal raffinamento della ricerca e si pongono nella linea di tendenza di una medicina personalizzata rispetto alle necessità dei singoli pazienti. Risolvere a livello atomico la struttura tridimensionale di un enzima determinante per avviare o completare un'infezione da virus (la neuramminidasi nel caso del virus influenzale, l'aspartato proteasi del virus HIV), oppure di uno specifico recettore cellulare necessario per innescare i meccanismi di infezione virale, significa fornire informazioni decisive per poter disegnare su misura farmaci in grado di interferire selettivamente con i corrispondenti processi patogenetici.
Naturalmente le metodologie di analisi cristallografica di tali proteine funzionalmente e patogeneticamente significative devono interfacciarsi in maniera 'progettuale' con quelle della chimica di sintesi e della chimica combinatoriale (un'associazione vincente di metodologie di sintesi organica 'a blocchi' e di analisi funzionale su larga scala delle molecole costruite) e con strategie di espressione genica anche orientate a produrre quantità adeguate delle stesse proteine e dei loro complessi con le molecole interagenti.
In altri casi la novità di un farmaco consiste nella costruzione di molecole chimeriche la cui attività possa essere preliminarmente calibrata in modo da fornire effetti organo-tessuto- e cellula-specifici. Disegnare strutture molecolari composte da più parti, di cui una, per esempio, conferente specificità di bersaglio (targeting) e un'altra dotata di proprietà predefinite (per es., attività citotossica nei confronti di cellule tumorali), significa concepire un farmaco 'intelligente', con ogni probabilità depurato di effetti collaterali o negativi su distretti cellulari sani. Naturalmente è necessario conciliare le conoscenze al momento disponibili con la previsione il più possibile razionale degli effetti biologici di molecole nuove e come tali non ancora collaudate: per esempio, la presenza di recettori per una proteina non solo nelle cellule su cui si vuole orientare il targeting, ma anche in altri distretti, potrebbe vanificare la costruzione di un farmaco efficace e sicuro. Di conseguenza, l'avanzamento degli strumenti terapeutici, soprattutto quelli di frontiera, è inevitabilmente ancorato all'avanzamento delle informazioni sugli aspetti molecolari della malattia che si vuole combattere e sulla fisiologia dei relativi sistemi (per es., endocrino, immunitario, nervoso).
Per queste ragioni la produzione biotecnologica di nuovi farmaci presenta vantaggi incalcolabili ma anche problemi di sicurezza che vanno ponderati con estrema attenzione, a prescindere dai risultati delle classiche prove di tossicità su animali. I vantaggi consistono nell'attuale potenzialità delle biotecnologie di creare farmaci in grado di curare qualsiasi malattia: tumori, malattie cardiovascolari, Parkinson, Alzheimer e AIDS. La condizione necessaria per ottenere questi risultati è un'informazione adeguata sulla patogenesi molecolare della malattia, tale da suggerire strumenti terapeutici sempre più mirati.
c) Le terapie cellulari si basano sulla possibilità di curare una serie di patologie ricorrendo a modificazioni su misura di cellule perlopiù autologhe. Di conseguenza, le fasi classiche di qualsiasi terapia cellulare sono: (1) il prelievo di cellule specifiche (per es., cellule del sangue); (2) la loro modificazione in vitro mediante metodologie aventi come obiettivo il ripristino di una funzione o l'acquisizione di funzioni nuove; (3) il controllo di qualità delle cellule modificate, seguito da (4) la loro somministrazione al paziente.
Quantunque la terapia genica rappresenti la versione più nota delle terapie cellulari, essa non è l'unica. I primi tentativi di immunoterapia adottiva di Steven Rosenberg (con trasferimento nei TIL, tumor-infiltrating lymphocytes, di un gene tracciante e non curativo) e l'attuale ricorso a eritrociti autologhi ingegnerizzati come trasportatori di farmaci e come bioreattori capaci di assicurare la conversione tempo-controllata e sede-specifica di profarmaci in farmaci (fabbriche circolanti di farmaci) ne sono ulteriori esempi. I successi registrati dai primi interventi di terapia genica nel trattamento di alcune immunodeficienze congenite e della malattia di Gaucher hanno prodotto aspettative considerevoli a cui non hanno finora corrisposto risultati clinici altrettanto risolutivi. A parte gli interrogativi sulla sicurezza di questi trattamenti, che al momento non sembrano legittimati da reali rischi per i pazienti, è verosimile che le attuali incertezze riflettano soprattutto due fatti: la mancanza di vettori realmente efficienti ai fini del trasferimento genico; il ricorso alla terapia genica in malattie non ancora sufficientemente caratterizzate sul piano molecolare. Esiste anche una terza possibilità, in parte legata alla seconda, e cioè che il trasferimento genico sia effettuato in cellule non del tutto idonee all'espletamento delle nuove funzioni, oppure in cellule idonee ma non nello stadio differenziativo più appropriato, oppure in quantità insufficienti alla correzione del difetto. Per questi motivi la ricerca biomedica punta con crescente convinzione all'ingegnerizzazione di cellule staminali totipotenti o multipotenti come quelle emopoietiche: in quest'ultimo caso l'obiettivo terapeutico (per es., ma non esclusivamente, la cura di emopatie ereditarie) è strettamente confinato nelle cellule oggetto del trasferimento del gene specifico.
In realtà, le tecnologie di trasferimento genico, oltre a correggere permanentemente malattie ereditarie monogeniche mediante normalizzazione di squilibri metabolici e funzionali, possono migliorare grandemente le conoscenze sulla biologia e sulla patologia di cellule, tessuti e organi attraverso la loro modificazione selettiva. Rientrano tra i numerosi esempi l'espressione di recettori normalmente assenti o a bassa densità, di enzimi inediti che rendono la cellula vulnerabile all'attacco di farmaci specifici ('suicidio' programmato di cellule tumorali), di enzimi direttamente responsabili dell'attivazione sede-specifica di profarmaci, o ancora di enzimi che conferiscono resistenza cellulo-specifica a chemioterapie antineoplastiche.
Valgono anche per le terapie cellulari le argomentazioni sui rapporti rischi/benefici già espresse per i nuovi farmaci. Pertanto, a fronte della necessità di una valutazione sperimentale e clinica particolarmente rigorosa, suggerita da eventuali incertezze sulla biologia dei sistemi e dei modelli terapeutici, si devono sottolineare le enormi potenzialità che questi interventi presentano sia per la correzione di malattie oggi ancora incurabili sia per approfondire le conoscenze nei termini atti a suggerire anche altre forme più tradizionali di terapia.
Genomica funzionale e proteomica
La fine del secondo millennio ha registrato la definizione della sequenza nucleotidica dei genomi di diversi organismi: nel 1996 del lievito Saccharomyces cerevisiae; nel 1998 del nematode Caenorhabditis elegans; nel 2000 del moscerino Drosophila melanogaster e della pianta Arabidopsis thaliana. Infine, nel febbraio 2001, l'annuncio più emozionante e più atteso, preceduto da tappe di avvicinamento rappresentate dalla sequenza di singoli cromosomi: quello della stesura semidefinitiva della sequenza del genoma di Homo sapiens, pubblicata sulla rivista "Nature" dal gruppo internazionale HGP (Human genome project) diretto da Francis Collins, e contemporaneamente sulla rivista "Science" dall'azienda privata Celera genomics (Rockville, Maryland) il cui responsabile era a quel tempo Craig Venter. L'evento è stato definito come una delle pietre miliari della storia dell'uomo e le motivazioni di questa definizione sono state espresse da Carina Dennis, Richard Gallagher e Philip Campbell nel modo seguente: "L'uomo rappresenta molto di più del prodotto di un genoma ma in un certo senso noi siamo definiti, sia come collettività sia come individui, nell'ambito del genoma. Pertanto la mappatura, il sequenziamento e l'analisi del genoma umano sono una tappa fondamentale nel processo di auto-conoscenza" (Dennis et al. 2001, p. 813).
Le caratteristiche del genoma umano sono così schematizzabili: 3,2×109 nucleotidi di cui 2,95×109 presenti nella cosiddetta eucromatina. Il 28% di queste sequenze polinucleotidiche è trascritto nell'RNA e l'1,1-1,4% è espresso in proteine. Il numero di geni codificanti per proteine non è stato ancora definito con esattezza, tuttavia sembra compreso tra 26.000 (Celera genomics) e 31.000 (HGP). Questi numeri appaiono incredibilmente piccoli se confrontati con quelli dei geni codificanti del lievito (6000), del moscerino (13.000), del nematode (18.000) e delle piante (26.000), cioè degli organismi i cui genomi sono stati sequenziati prima di quello umano. Tuttavia, va sottolineato che i geni umani presentano la massima complessità organizzativa e risultano espressi attraverso sistemi multipli di splicing alternativo che determinano la produzione di un più ampio numero di proteine.
Nessuno dei numeri riportati nel 2001 era ancora definitivo e infatti i risultati comunicati congiuntamente da HGP e dalla Celera genomics sono stati formalmente definiti come una 'prima bozza' di sequenza del genoma. Nel frattempo è proseguito con alacrità il completamento dell'intera sequenza, inteso come la definizione del 99% della stessa. Nell'aprile 2003 Francis Collins ha pubblicamente annunciato il raggiungimento di questo obiettivo da parte di HGP, tracciando al tempo stesso le linee-guida della ricerca genomica in un articolo destinato ad entrare a pieno diritto nella storia della scienza (Collins et al., 2003). Questo articolo è comparso sul numero di "Nature" del 24 aprile 2003, che coincide simbolicamente con il 50° anniversario della pubblicazione sulla stessa Rivista della struttura a doppia elica del DNA da parte di James D. Watson e Francis Crick (1953).
Quantunque non siano stati ancora forniti dettagli definitivi di questa informazione, dal lavoro di numerosi ricercatori impeganti nel progetto di sequenziamento genico del DNA umano si apprende che alcune delle anticipazioni fornite nel 2001 non erano corrette. Per esempio, nella recentissima presentazione dell'intera sequenza del cromosoma 7 umano, Stephans W. Scherer e i suoi collaboratori (2003), oltre a definire in 158,953 milioni di nucleotidi il numero di tale sequenza e in 1917 il numero di strutture corrispondenti a geni, hanno rilevato che le dimensioni medie di questi geni sono più grandi di quelle riportate nel 2001 da HGP e da Celera genomics. In ogni caso, risultati come quelli emersi nell'aprile 2003 segnano la strada definitiva per stabilire con precisione ed esattezza il numero dei geni presenti nel patrimonio genetico umano, sia di quelli che vengono trascritti in diverse specie di RNA (tRNA, rRNA, small nucleolar RNA o snoRNA, small nuclear RNA o snRNA), sia di quelli che codificano proteine. Per realizzare questo obiettivo, così importante perché strettamente correlato a nozioni di fisiologia cellulare, sono in fase di perfezionamento tecnologie avanzate in cui confluiscono la biologia molecolare e la bioinformatica, ma risulterà fondamentale anche l'insieme delle informazioni di genomica comparata derivanti dalle sequenze di altri genomi complessi, per esempio, quello del topo, la cui prima bozza di sequenza genomica è stata riportata dal gruppo internazionale MGSC (Mouse genome sequencing consortium) nel dicembre 2002 ("Nature" 2002).
Tuttavia, anche ammettendo che la genomica comparata possa rispondere al quesito ("che cosa ci rende umani?") formulato da David Baltimore (2001), un altro, forse ancora più emozionante, interrogativo è il seguente: quali sono le basi molecolari dell'individualità biologica? Un approccio alla soluzione di questo enigma è la compilazione completa dei polimorfismi a livello di un singolo nucleotide (SNP, single nucleotide polymorphism) che, sulla base di considerazioni statistiche, ammontano a 1,42 milioni di siti di variazione su tutto il genoma umano, ma che potrebbero essere ancora più numerosi. Oltre ad avvicinare frontiere da fantascienza, per esempio l'individualità biologica come determinante di qualità fisiche, intellettuali, etiche, emozionali, cognitive e comportamentali, la decifrazione degli SNP potrà fornire informazioni oggettive sulla risposta dei singoli individui a molti farmaci ('farmacogenomica'). Altre naturali conseguenze di questo catalogo degli SNP saranno la possibilità di definire la suscettibilità dei singoli soggetti a malattie, la decifrazione molecolare di malattie monogeniche e multigeniche, e pertanto la creazione di nuovi, efficaci strumenti di diagnosi e di prevenzione.
Molte di queste linee di tendenza della ricerca postgenomica (successiva cioè alla prima bozza di sequenza del genoma) sono, come sempre accade nelle biotecnologie, orientate a finalità conoscitive e insieme aperte ad applicazioni. Anche l'identificazione sistematica di sequenze polinucleotidiche che regolano l'espressione dei classici geni strutturali (compresi quelli non ancora caratterizzati) potrà fornire strumenti formidabili dalla duplice valenza, scientifica e applicativa. Tale identificazione viene perseguita attraverso strategie di genomica comparata (tra diverse specie) e mediante tecnologie di genomica funzionale legate allo studio sistematico dell'espressione genica. Lo strumento dei DNA chips (o DNA microarrays) sta entrando nella ricerca postgenomica in misura crescente al fine di esplorare quali dei geni noti o quali sequenze polinucleotidiche vengano differenzialmente espresse in condizioni tra loro paragonabili (per es., in un soggetto prima e dopo la somministrazione di un farmaco; in una serie di neoplasie anche istologicamente e biologicamente omogenee; in un sistema cellulare idoneo allo studio di processi di differenziamento). Le potenzialità di queste tecnologie altamente integrate e multidisciplinari (high throughput technologies) sono incredibilmente alte e le prospettive di successo spaziano dalla decifrazione funzionale di segmenti del genoma (a cosa servono e come) all'identificazione di determinanti molecolari di malattia. La bioinformatica costituisce un mezzo ineludibile per trasformare dati complessi e altrimenti non interpretabili in risultati che possano avere applicazioni anche immediate (per es., una mappatura differenziale di neoplasie).
Il flusso dell'informazione molecolare dal DNA all'RNA e da questo alle proteine, e l'enorme eterogeneità strutturale e funzionale delle proteine stesse, hanno portato a battezzare nuove e delimitate aree di ricerca con nomi un po' curiosi ma in ogni caso identificativi di settori della biochimica e della biologia molecolare e cellulare: 'transcriptoma' (l'insieme dei sistemi di trascrizione del DNA in una cellula), 'proteoma' (il complesso delle proteine in una singola cellula o in un singolo sistema biologico), 'metaboloma' (la rete di reazioni che costituiscono il metabolismo di un determinato organismo).
Come si è detto, il numero delle proteine presenti in un sistema biologico è grandemente superiore a quello dei geni che codificano tali proteine. Così, a un numero di geni strutturali non lontano da 30.000 nell'uomo, fa riscontro una moltitudine di proteine espresse a partire da quegli stessi geni che non si attesta ancora su numeri definitivi, ma che oscilla nelle varie previsioni da 500.000 a diversi milioni. Le ragioni di questa sproporzione numerica sono legate soprattutto a diverse possibilità di maturazione delle sequenze geniche (splicing alternativo di un singolo gene), ma anche a un gran numero di modificazioni posttraduzionali (meglio sarebbe definirle collettivamente postbiosintetiche) che comprendono diverse tipologie di modificazioni covalenti delle proteine, prima tra tutte, per diffusione nei sistemi biologici, la loro fosforilazione-defosforilazione.
Considerando l'incredibile eterogeneità delle funzioni espresse dalle proteine (catalitiche, regolatorie, strutturali, anticorpali, ormonali, differenziative e altre ancora), non sorprende certamente che una delle aree più complesse e più coltivate della ricerca postgenomica sia la proteomica. Essa, a stretto rigore di termine, comprende tutti gli studi di catalogazione delle diverse proteine prodotte in una cellula. Tuttavia sarebbe molto riduttivo considerare esclusivamente il numero e il tipo di proteine diverse espresse in un sistema biologico senza precisarne il momento di espressione e le mutevoli condizioni in cui avviene la loro produzione, ignorandone cioè il contesto dinamico. In aggiunta, anche il più sofisticato tra gli elaboratori elettronici non potrebbe fornire altro che dati puramente classificativi, utili per esempio a definire il profilo di espressione delle proteine di una cellula in diversi momenti del suo ciclo o in condizioni di analisi comparativa. In altri termini, queste informazioni possono essere preziose a fini diagnostici e analitici (in medicina, ma anche in settori come la zootecnia, l'agricoltura e l'ambiente), ma non illuminano a sufficienza sui ruoli e sulle funzioni di queste proteine. Pertanto il concetto di proteomica si è andato giustamente estendendo allo studio delle strutture delle proteine e sta sfumando sempre più in quello delle relative funzioni. Ciò significa ricorrere a strumenti analitici molto raffinati (per es., la compilazione di moduli di struttura tridimensionale comuni a più proteine, ovviamente effettuata attraverso dati di cristallografia ai raggi X o di spettroscopia NMR) ma, al tempo stesso, ripercorrere approcci sperimentali più tradizionali della biochimica e della biologia cellulare. Tra questi lo studio di interazioni tra proteine (o tra subunità di una stessa proteina) e la valutazione di loro funzioni mediante neutralizzazione con anticorpi specifici e di risupplementazione con le corrispondenti proteine purificate; oppure l'analisi del metabolismo, di cascate di regolazione, di processi come proliferazione, eccitazione, secrezione in cellule ipo- o sovra- esprimenti proteine specifiche; mutagenesi sito-specifiche per ottenere cellule modificate ad hoc ed esplorarne le varie funzioni; la perturbazione di stati stazionari di sistemi biologici con agenti regolatori quali fattori di crescita/differenziamento o anche con reagenti di laboratorio; la ricostruzione in vitro di sistemi metabolici semplici mediante proteine purificate e la successiva analisi delle proprietà funzionali e di regolazione.
Per la molteplicità di questi approcci e per la loro indiscussa correlazione con le informazioni derivanti dalla sequenza dei vari genomi, è stata coniata una definizione, 'genomica strutturale', che tende a ricompattare la genomica funzionale con la struttura delle proteine. Al di là della semantica (ispirata spesso da interessi accademici o frequentemente da interessi commerciali di aziende specializzate in slogan per vendere prodotti o servizi), la ricerca biotecnologica attuale deve tradurre cataloghi di geni e di sequenze polinucleotidiche in funzioni la cui esplorazione sta riabilitando il ruolo delle proteine. Come già accennato, negli anni Cinquanta-Sessanta il mondo delle proteine fu al centro delle ricerche che ne permisero le prime analisi di correlazione tra struttura e funzione, mentre le conoscenze sul DNA e sull'RNA erano grandemente limitate da problemi quantitativi. Dai primi anni Settanta, le tecnologie del DNA ricombinante ribaltarono la situazione, permettendo l'analisi fine degli acidi nucleici ma al tempo stesso consentendo di esprimere e quindi di caratterizzare numerose proteine. Oggi lo studio delle interazioni tra geni, che rappresenta l'obiettivo centrale della ricerca postgenomica, viene nuovamente esplorato in massima parte a livello delle proteine. Anche per queste ragioni è stato attivato un Consorzio internazionale, definito HUPO (Human proteome organization), in analogia a HUGO (Human genome organization). È stato giustamente sottolineato da più parti che sarebbe illusorio attendersi da HUPO tempi di lavoro altrettanto rapidi quanto quelli di HUGO: la chimica delle proteine, a dispetto dei loro pesi molecolari mediamente inferiori, è molto più complessa di quella degli acidi nucleici, per cui lo studio strutturale delle proteine richiede tecnologie in continua evoluzione, molte delle quali coperte da brevetti e quindi non immediatamente disponibili a utilizzatori accademici. Come è stato recentemente sottolineato da Sydney Brenner, è auspicabile che le scienze e le tecnologie della vita non abbandonino la loro stessa essenza, che consiste nel seguire l'approccio sperimentale per convalidare le ipotesi. Sarebbe ovviamente illusorio pensare che le nuove frontiere delle biotecnologie, identificate come genomica e proteomica, "ci possano liberare dal predominio delle ipotesi, cioè del pensiero e della riflessione" (Brenner 2000, p. 584).
Conclusioni
Le interazioni tra la ricerca e la società, mai sufficientemente consolidate, stanno registrando negli ultimi tempi un'accelerazione e un'intensificazione senza precedenti di cui le biotecnologie sono protagoniste quasi esclusive. Gli avanzamenti dell'elettronica, dell'informatica e delle telecomunicazioni hanno rimodellato la nostra vita quotidiana in misura anche superiore a quanto finora fatto dalle biotecnologie. Tuttavia, l'atteggiamento delle persone nei confronti di questa ondata di nuove tecnologie (high-tech) è in genere quello di utilizzatori in parte acritici dei relativi benefici e servizi. Da pochi anni, viceversa, le biotecnologie sono al centro dell'attenzione generale, sollevando quesiti ‒ giuridici, etici, filosofici oltre che scientifici ‒, alimentando discussioni spesso non obiettive né serene, interagendo con le scienze umane tipicamente rappresentate dalla sociologia e tradizionalmente influenzate dai valori dell'intelletto e dello spirito.
Questa crescente influenza delle biotecnologie sulla società ha motivazioni precise ma anche limiti. Le motivazioni risiedono nel fatto che qualsiasi individuo tende a usare le tecnologie informatiche ed elettroniche o in maniera passiva o al massimo sforzandosi di padroneggiarle a fini personali. I telefoni cellulari possono essere usati come semplice strumento di comunicazione a distanza o come sempre più elaborati sistemi di navigazione in rete a fini di lavoro, di svago e, in ogni caso, di espressione della propria personalità. Invece le scienze e le tecnologie della vita toccano direttamente l'individuo e la collettività proprio perché sono basate sullo studio, ma anche sulla modificazione pianificata, dei sistemi biologici che, come si è detto, spaziano da una semplice molecola all'uomo. Anche qualora la ricerca non riguardi direttamente l'uomo come sistema di studio, appaiono evidenti in ogni caso gli approcci e i risultati che le biotecnologie raggiungono nel controllo dell'ambiente, nella sicurezza degli alimenti e nell'agricoltura.
I limiti del serrato dialogo tra operatori, cittadini, politici, sociologi, giuristi, magistrati e teologi sul tema delle biotecnologie sono quelli di una crescente divaricazione tra i valori tecnico-scientifici (destinati inevitabilmente a migliorare per il circolo virtuoso più volte ricordato) e i contenuti delle scienze umane, il cui progresso è storicamente molto meno rapido. Soprattutto nella civiltà occidentale le ideologie, i costumi, le relazioni tra gli individui e tra le popolazioni, le riflessioni, le manifestazioni della creatività umana in tutte le arti, sono scandite da ritmi evolutivi decisamente più lenti rispetto a quelli che caratterizzano l'avanzamento della ricerca scientifica.
I ricercatori nell'ambito delle scienze e delle tecnologie della vita si sono trovati impreparati a gestire le reazioni dell'opinione pubblica per il peso progressivamente crescente della ricerca biotecnologica nel modo di vivere e soprattutto di condizionare il futuro dell'umanità. Ne sono derivate e tuttora ne derivano numerose discussioni, con il rischio di rinunciare al possibile miglioramento della qualità della vita offerto dall'impiego oculato ed eticamente responsabile di strumenti in continuo perfezionamento. Un esempio di questi problemi è rappresentato dalla generica accettazione delle biotecnologie in medicina ('biotecnologie rosse'), probabilmente dovuta al naturale desiderio di non rinunciare ai benefici derivanti dall'avanzamento delle scienze mediche, contrapposta al rifiuto delle biotecnologie agroalimentari ('biotecnologie verdi'), delle quali vengono percepiti, più che i vantaggi, gli eventuali problemi che ne possono derivare.
D'altronde, questi schieramenti sulle diverse applicazioni delle biotecnologie possono ribaltarsi; infatti la ricerca biotecnologica può rispettare l'ambiente terrestre e acquatico e proteggerlo attraverso interventi risolutivi di decontaminazione o di prevenzione degli inquinamenti. La clonazione animale può non soltanto realizzare la produzione di animali da allevamento o addirittura la conservazione di specie in via di estinzione (come nel caso recentemente dimostrato dei mufloni), ma anche fornire informazioni cruciali sui meccanismi di riprogrammazione dell'informazione genetica nel passaggio da cellule specializzate a cellule multipotenti.
Le agrobiotecnologie hanno già dimostrato di poter offrire sistemi sensibili di monitoraggio dei suoli, di produzione di piante utili come bioreattori per ottenere farmaci, anticorpi e vaccini da impiegare in medicina, e anche di piante a elevato valore agronomico. Viceversa, gli interventi delle biotecnologie in medicina potrebbero, se non adeguatamente controllati, avere conseguenze negative: gli screening diagnostici di massa potrebbero compromettere la riservatezza dei dati biologici individuali, farmaci innovativi potrebbero rivelarsi inefficaci o anche dotati di effetti collaterali non previsti, vaccini ritenuti efficaci per malattie non sufficientemente caratterizzate sul piano biologico potrebbero non fornire effetti di protezione.
In ogni caso, colpevolizzare o accettare per settori applicativi la ricerca biotecnologica sembra insensato, in quanto i suoi strumenti di lavoro sono universali e quindi vi è continuità dottrinale e applicativa delle varie metodologie.
Queste considerazioni portano a una conclusione molto semplice. Le biotecnologie devono assicurare il conseguimento di beni e servizi ma, ove impropriamente utilizzate, potrebbero generare disastri o quanto meno incertezze. Il paragone con l'energia nucleare, il cui uso perverso ha portato alla bomba atomica, ma che ha prodotto e sta producendo beni immensi all'umanità, è calzante e infatti viene spesso citato. Chi deve gestire la ricerca biotecnologica e le sue applicazioni? I ricercatori rappresentano i veri e probabilmente unici esperti, tuttavia potrebbero difettare di spirito critico anche perché direttamente protagonisti di un'attività emozionante, totalizzante e ai confini delle conoscenze. I cultori delle scienze umane possono certamente esercitare forme di controllo perché portatori di istanze generali, ma proprio per questo devono assicurare equilibrio e piena ricettività alle informazioni tecnico-scientifiche. I politici dovrebbero funzionare da collettori e mediatori delle diverse tendenze, non rinunciando al loro decisivo compito di attuatori di tali linee di indirizzo.
Pertanto è necessario che, modulando gli strumenti di valutazione e i successivi interventi in relazione agli ambiti di lavoro (un ente di ricerca, un'industria, una nazione, la comunità internazionale), l'attenzione alle biotecnologie innovative e al loro impiego venga considerata come un dovere ineludibile, affidato a persone responsabili e capaci di portare contributi diversificati ma qualitativamente di documentato e altissimo livello. Solamente la collegialità delle decisioni ‒ basata su trasparenza, correttezza e valore delle informazioni ‒ può assicurare la decisa attivazione, l'avanzamento e la raccolta dei benefici di ricerche che possono migliorare in maniera determinante la qualità di vita dell'umanità.
Infine, è opportuno fare due considerazioni. In primo luogo, la società ha tutto il diritto di discutere, valutare e decidere su questioni che la riguardano in maniera diretta. Tuttavia non bisogna dimenticare che la critica deve essere pienamente motivata e che non deve sostituire l'operatività della ricerca, per la quale sono necessarie considerevoli risorse umane e finanziarie: la spirale positiva ricerca-benessere-occupazione richiede investimenti e non parole. La seconda notazione, specifica delle biotecnologie, è che i valori oggettivi della ricerca (conoscenze) devono indurre filosofi, sociologi e teologi a considerare in maniera più completa i sistemi biologici ma anche entità finora astratte e la cui interpretazione è stata lasciata alle libere scelte individuali. Un esempio dei primi concerne la costruzione di animali transgenici: questa tecnologia può anche portare a realizzare bioreattori viventi e dotati di buona salute ai fini della produzione programmata di grandi quantità di farmaci utili (proteine dal latte, dal sangue), ma molto più spesso significa ottenere modelli di malattie umane per l'impostazione di nuove e risolutive terapie. Questa doppia potenzialità non deve identificare i primi come transgenici 'buoni' e i secondi come 'cattivi'. Un esempio delle entità da regolamentare giuridicamente ed eticamente riguarda la confusione tra la clonazione umana per la riproduzione in fotocopia di singoli individui e la clonazione a fini terapeutici (medicina rigenerativa per la cura di malattie invalidanti e letali). Tale confusione deve trovare soluzione nella definizione motivata delle differenze tra l'embrione vero e proprio e un 'organismo embrioide' (embryoid body, derivante dal trasferimento di nuclei di cellule somatiche in ovociti enucleati) come serbatoio di cellule multipotenti da indirizzare verso la produzione in vitro o in vivo di neoorgani e di neotessuti.
Tavola IV - DAL VITELLO GALILEO ALLA PULEDRA PROMETEA
Cesare Galli e i suoi collaboratori (1999) hanno microiniettato il nucleo di un leucocita circolante del toro Zoldo in una cellula uovo prelevata da una bovina frisona e precedentemente privata del proprio nucleo. La cellula uovo così preparata è lo stadio di blastocisti e successivamente è stato effettuato un secondo intervento di trasferimento nucleare mediante fusione di singoli blastomeri con cellule uovo enucleate come sopra descritto. Questo ha comportato il raggiungimento dello stadio di blastocisti (di seconda generazione) che è stato successivamente inserito nell’utero di una manza nella fase opportuna del ciclo estrale. Mentre le gravidanze in tal modo iniziate a partire da numerosi esperimenti di trasferimento nucleare si sono spontaneamente interrotte in massima parte prima del 45° giorno, una è arrivata a termine dopo 287 giorni, portando alla nascita di un vitello, chiamato Galileo. Questo risultato, dopo il primo successo registrato da Ian Wilmut (1997) con la nascita della pecora Dolly, ha sollevato in Italia polemiche e discussioni, ma è stato molto importante per almeno due ragioni. Anzitutto, ha dimostrato la possibilità di clonare un animale (Galileo è semplicemente un gemello monozigote del ‘padre’ Zoldo) partendo da cellule del sangue facilmente prelevabili e conservabili. Di conseguenza, questo insieme di tecnologie, relativamente semplici anche se con bassa percentuale di successo (cioè di gravidanze a termine), risulta molto promettente in zootecnia al fine di ottenere animali da allevamento pregiati o di conservare specie animali in via di estinzione – come è stato realizzato da Pasqualino Loi (2001) con la clonazione di un muflone selvatico mediante trasferimento nucleare da cellule somatiche di un muflone morto in cellule uovo enucleate prelevate da una specie domestica di pecora. In secondo luogo, esso dimostra la possibilità di ottenere la riprogrammazione delle informazioni contenute nel nucleo di una cellula adulta. Tale evento consiste nello sdifferenziamento di una cellula somatica già pienamente differenziata (cioè con proprietà ben precise e abbastanza bene identificate, come quelle di un leucocita), seguito dal differenziamento fino a poter ottenere i diversi tessuti e organi. Per tale motivo, la tecnica di trasferimento nucleare in cellule uovo enucleate viene attualmente considerata da alcuni come la possibilità più eticamente accettabile (anche se, per il momento, certamente non la migliore sul piano dei risultati pratici) di sviluppare cellule embrionali staminali. Queste possono essere così ottenute immunocompatibili da individui adulti ed essere utilizzate per la ricostruzione di tessuti e organi in quella che viene comunemente definita ‘medicina rigenerativa’.
Dopo la nascita di Galileo la ricerca sulla clonazione è proseguita e si è sviluppata in altre specie inclusa quella equina. Nel 2003 il gruppo di Cesare Galli ottiene il primo cavallo clonato al mondo, una puledra di razza avelignese (Prometea). La procedura di clonazione è simile a quella utilizzata per il bovino ma con alcune peculiarità tecniche come la rimozione del guscio glicoproteico che protegge gli ovociti, prima della enucleazione degli stessi. In questo modo la fusione tra l’ovocita enucleato e la cellula somatica è facilitata. Anche la fase di attivazione che segue alla fusione tra ovocita e cellula somatica è modificata rispetto al bovino e alle altre specie clonate in precedenza. Nel caso di Prometea le cellule utilizzate per la clonazione derivano da una biopsia cutanea dalla quale è stata ottenuta una linea cellulare fibroblastica. L’eccezionalità di questo risultato è duplice perché non solo si tratta del primo equino derivato da clonazione somatica da adulto, ma anche di una gravidanza autologa poiché Prometea è stata partorita dalla stessa cavalla dalla quale sono state prelevate le cellule cutanee utilizzate per la clonazione. Importanti sono le implicazioni di questo risultato per quanto riguarda lo studio della immunologia della gravidanza. Infatti esiste notevole evidenza sperimentale che dimostra come l’instaurarsi della gravidanza dipende da un adeguato riconoscimento immunologico del feto come diverso (not self ) rispetto alla madre. Prometea dimostra che la clonazione somatica è una tecnica che consente di studiare e di capire fondamentali processi biologici legati alla riprogrammazione e al differenziamento cellularema anchemeccanismi immunologici più specifici che intervengono nel mantenimento della gravidanza. (C. Galli)
Bibliografia
Baltimore 2001: Baltimore, David, Our genome unveiled, "Nature", 409, 2001, pp. 814-816.
Brenner 2000: Brenner, Sydney, Biochemistry strikes back, "Trends in biochemical sciences", 25, 2000, p. 584.
Cohen 1973: Cohen, Stanley N. - Chang, Annie C.Y. - Boyer, Herbert W. - Helling, Robert B., Construction of biologically functional bacterial plasmids in vitro, "Proceedings of the National Academy of Sciences USA", 70, 1973, pp. 3240-3244.
Collins, 2003: Collins, Francis S. - Green, Eric D. - Guttmacher, Alan E. - Guyer, Mark S., A vision for the future of genomic research. A blueprint for the genomic era, "Nature", 422, 2003, pp. 835-847.
De Flora 2000: De Flora, Antonio, Biochimica, in: Appendice 2000, Roma, Istituto della Enciclopedia Italiana, 2000, I, pp. 203-209.
Dennis 2001: Dennis, Carina - Gallagher, Richard - Campbell, Philip, Everyone's genome, "Nature", 409, 2001, p. 813.
Galli 1999: Galli, Cesare - Duchi, Roberto - Moor, Robert M. - Lazzari, Giovanna, Mammalian leukocytes contain all the genetic information necessary for the development of a new individual, "Cloning", 1, 1999, pp. 161-170.
Kan, Dozy 1978: Kan, Yuet Wai - Dozy, Andrée M., Polymorphism of DNA sequence adjacent to human β-globin structural gene: relationship to sickle mutation, "Proceedings of the National Academy of Sciences USA", 75, 1978, pp. 5631-5635.
Kaplan, Delpech 1993: Kaplan, Jean-Claude - Delpech, Marc, Biologie moléculaire et médecine, Paris, Flammarion, 1993 (trad. it.: Biologia molecolare e medicina, Napoli, G. Gnocchi ed., 1995).
Köhler, Milstein 1975: Köhler, Georges - Milstein, César, Continuous cultures of fused cells secreting antibody of predefined specificity, "Nature", 256, 1975, pp. 495-497.
Lodish 1995: Lodish, Harvey - Baltimore, David - Berk, Arnold - Zipursky, S. Laurence - Matsudaira, Paul - Darnell, James, Molecular cell biology, 3. ed., New York, Freeman, 1995 (1. ed.: New York, Scientific American Books, 1986).
Loi 2001: Loi, Pasqualino - Ptak, Grazyna - Barboni, Barbara - Fulka, Josef jr - Cappai, Pietro - Clinton, Michael, Genetic rescue of an endangered mammal by cross-species nuclear transfer using post-mortem somatic cells, "Nature biotechnology", 19, 2001, pp. 962-964.
Maxam, Gilbert 1977: Maxam, Alan M. - Gilbert, Walter, A new method for sequencing DNA, "Proceedings of the National Academy of Sciences USA", 74, 1977, pp. 560-564.
Mullis, Faloona 1987: Mullis, Kary - Faloona, Fred A., Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction, "Methods in enzymology", 155, 1987, pp. 335-350.
Nathans, Smith 1975: Nathans, Daniel - Smith, Hamilton O., Restriction endonucleases in the analysis and restructuring of DNA molecules, "Annual review of biochemistry", 44, 1975, pp. 273-293.
Palmiter 1982: Palmiter, Richard D. - Brinster, Ralph L. - Hammer, Robert E. - Trumbauer, Myrna E. - Rosenfeld, Michael G. - Birnberg, N.C. - Evans, Ronald M., Dramatic growth of mice that develop from eggs microinjected with metallothionein-growth hormone fusion genes, "Nature", 300, 1982, pp. 611-615.
Pirastu 1987: Pirastu, Mario - Galanello, Renzo - Doherty, Marie A. - Tueri, Teresa - Cao, Antonio - Kan Ywet Wai, The same β-globin gene mutation is present on nine different β-thalassemia chromosomes in a Sardinian population, "Proceedings of the National Academy of Sciences USA", 84, 1987, pp. 2882-2885.
Scherer, 2003: Scherer, Stephens W. e altri, Human chromosome 7: DNA sequence and biology, "Science", 300, 2003, pp. 767-772.
Southern 1975: Southern, Edwin M., Detection of a specific sequence among DNA fragments separated by gel electrophoresis, "Journal of molecular biology", 98, 1975, pp. 503-517.
Stryer 1995: Stryer, Lubert, Biochemistry, 4. ed., New York, Freeman, 1995 (1. ed.: San Francisco, Freeman, 1975).
The human genome, "Science", 291 (numero monografico), 2001.
Waterston 2002: Waterston, Robert H. e altri, Initial sequencing and comparative analysis of the mouse genome, "Nature", 420, 2002, pp. 520-562.
Wilmut 1997: Wilmut, Ian - Schnieke, Angelica E. - McWhir, Jim - Kind, A.J. - Campbell, Keith H., Viable offspring derived from fetal and adult mammalian cells, "Nature", 385, 1997, pp. 810-813.